silva做菌种鉴定
简介:
SILVA一词起源于拉丁文silva(意为forest),它是一个包含三域微生物(细菌、古菌、真核)rRNA基因序列的综合数据库,其数据库涵盖了原核和真核微生物的小亚基rRNA基因序列(简称SSU,即16S和18SrRNA)和大亚基rRNA基因序列(简称LSU,即23S和28SrRNA)。目前其最新数据库版本为SILVA SSU andLSU databases 128,更新时间为2016年9月29日,最新版本数据库包含的数据信息见下表1所示。
表1 SILVA SSU andLSU databases 128数据库基本参数信息
SSU参考序列 | SSU非冗余参考序列 | LSU总序列 | LSU参考序列 | |
版本 | 128 | 128 | 128 | 128 |
总序列 | 1,922,213 | 645,151 | 735,238 | 154,297 |
细菌 | 1,719,541 | 552,377 | 176,194 | 130,965 |
古菌 | 64,390 | 24,315 | 1528 | 1271 |
真核 | 140,020 | 68,996 | 557,769 | 22,105 |
可培养 | 36,747 | 36,747 | 24,664 | 8232 |
模式株 | 22,334 | 22,334 | 5809 | 4675 |
因为SILVA数据库更新比较及时,因此是目前rRNA基因高通量测序后最常选用的参考数据库之一。此外,SILVA也可被用于平时菌种鉴定时,对少量rRNA基因测序后的物种进行分类鉴定,此时主要用其SINA Alignment Service功能(https://www.arb-silva.de/aligner/),可非常方便地确定某条rRNA基因序列从门到属/种水平的分类信息并给出各分类水平相应的置信度。
主页:
https://www.arb-silva.de/
菌种鉴定方法:
1.选择序列搜索。
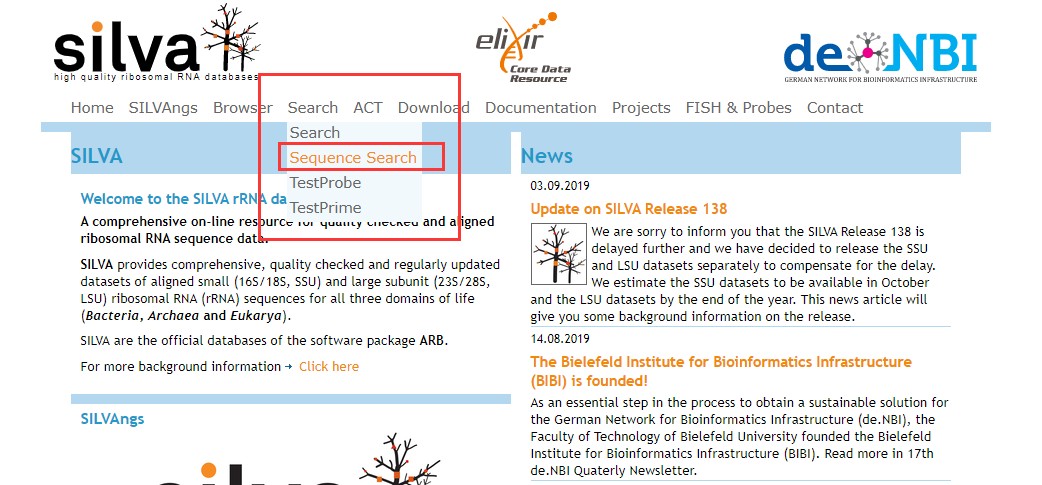
2.输入fasta格式的序列
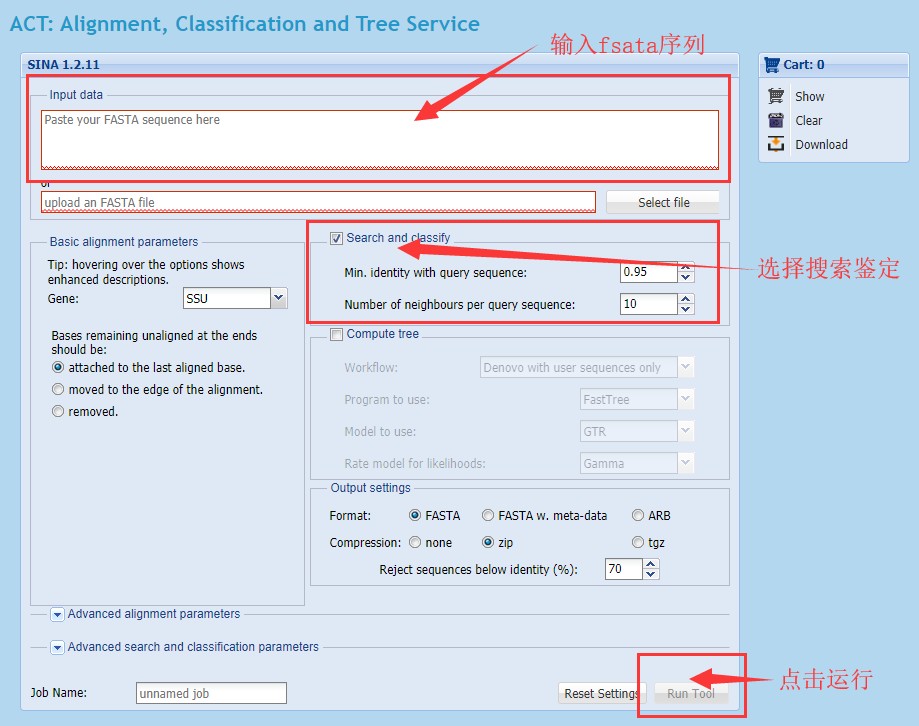
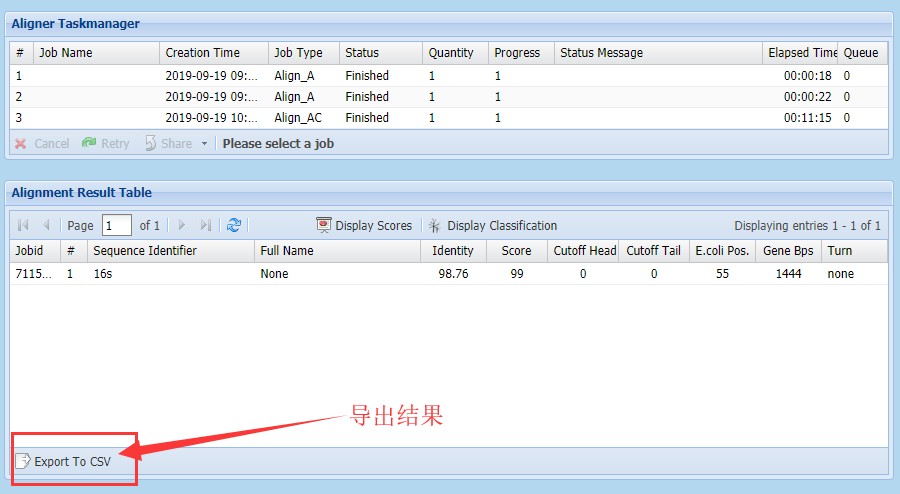
3.搜索结果如下,可将结果导出,方便查看。
此外,我们在网易云课堂上有各种教学视频,有兴趣可以了解一下:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘
6. 更多学习内容:linux、perl、R语言画图,更多免费课程请点击以下链接:
- 发表于 2019-09-20 13:18
- 阅读 ( 4926 )
- 分类:软件工具
你可能感兴趣的文章
- qiime2 分类器建立 SILVA数据库 10284 浏览
