SignalP:信号肽预测工具
SignalP:信号肽预测工具
SignalP是一个信号肽预测服务器,它的功能是预测给定的氨基酸序列中是否存在潜在的信号肽剪切位点及其所在,原核生物和真核生物都可以进行预测。目前服务器提供的是SignalP 5.0版本。在线服务器网址:http://www.cbs.dtu.dk/services/SignalP/
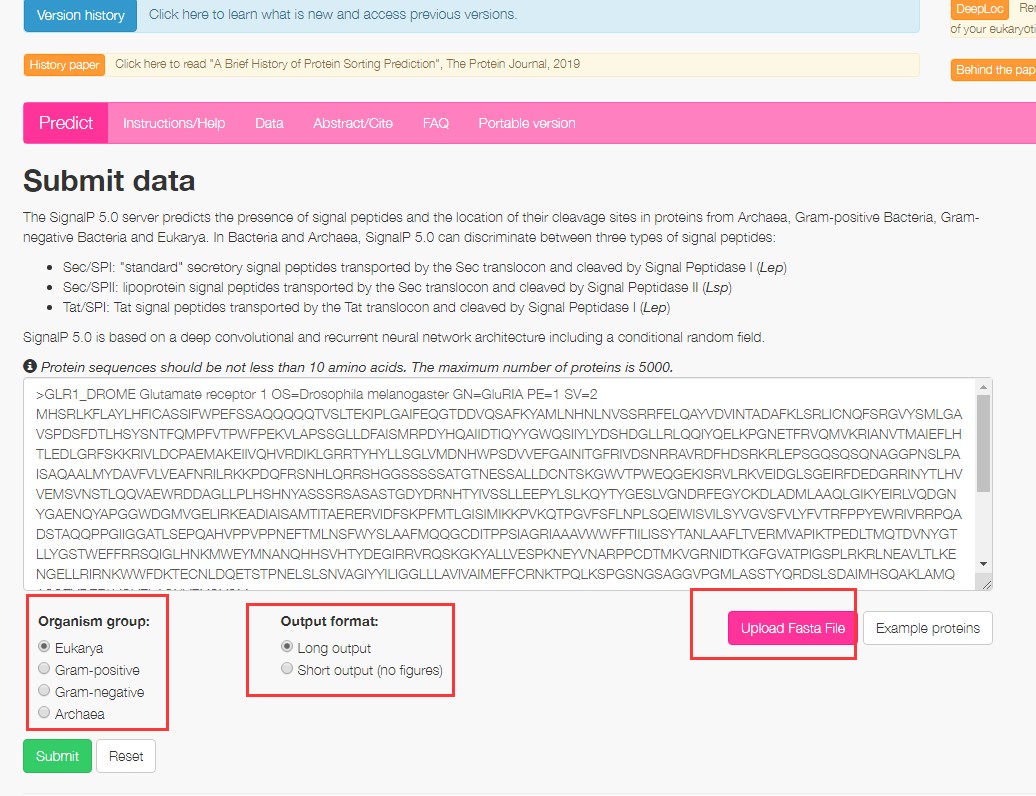
A.序列输入方式:
有两种方式:一种是直接输入FASTA格式的氨基酸序列,可以是一条也可以是多条;另一种是本地上传包含含有FASTA格式序列的文件。
这里要注意我们必须输入氨基酸序列,看过有的朋友用核酸序列去预测的;输入的氨基酸序列为单字母简写形式。除了20中氨基酸外,仅允许使用X代表未知氨基酸。所有序列中的其他字母(非20种氨基酸简写和X的)被视为X;所有的非字母字符将会自动忽略。
还要注意的是对序列长度和多少的限制:每次最多提交2000条序列进行分析;总氨基酸残基不能超过20万;每条序列不能超过6000残基。其实一般来讲我们都不会有这么多序列一起去预测的O(∩_∩)O哈哈~
B.物种选择:
根据你预测的蛋白质来源选择革兰氏阳性细菌、革兰氏阴性细菌或真核生物。
- 发表于 2019-10-21 10:17
- 阅读 ( 6488 )
- 分类:软件工具
你可能感兴趣的文章
相关问题
0 条评论
请先 登录 后评论
