TCGA数据库中临床样品编号详解(Barcode)
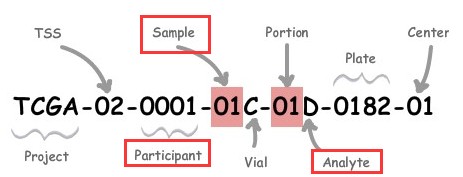
接触和分析过TCGA数据的朋友肯定会经常处理TCGA barcode的7个编码信息,每个编码信息用横杠-隔开,如下所示:
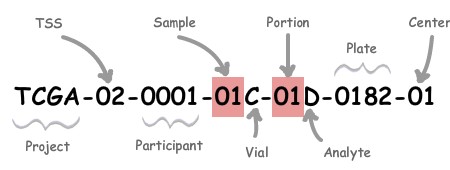 第一次分析TCGA数据看着这么长的样品编号感觉很是难以理解(例如:TCGA-3M-AB46-01A-11D-A410-08,TCGA-3M-AB47-01A-22D-A410-08,TCGA-B7-5816-01A-21D-1600-08,TCGA-B7-5818-01A-11D-1600-08),但是这个编号里面又蕴含了很多关于样品来源的信息,如果了解样品编号原则,就可以很好的区分样品,有利于数据的分析与处理。
第一次分析TCGA数据看着这么长的样品编号感觉很是难以理解(例如:TCGA-3M-AB46-01A-11D-A410-08,TCGA-3M-AB47-01A-22D-A410-08,TCGA-B7-5816-01A-21D-1600-08,TCGA-B7-5818-01A-11D-1600-08),但是这个编号里面又蕴含了很多关于样品来源的信息,如果了解样品编号原则,就可以很好的区分样品,有利于数据的分析与处理。
之所以有这么复杂的样品编号是因为: 不同的数据之间需要关联,同一个病人有多种数据,甚至一种数据也有多个,比如转录组数据某病人就有癌症和癌旁,还有芯片数据,甲基化数据,SNP突变数据等等都需要用ID来进行关联和区分。
TCGA数据处理过程
要想充分理解样品barcode的编码规律,就要先从BCR处理样品的过程说起:
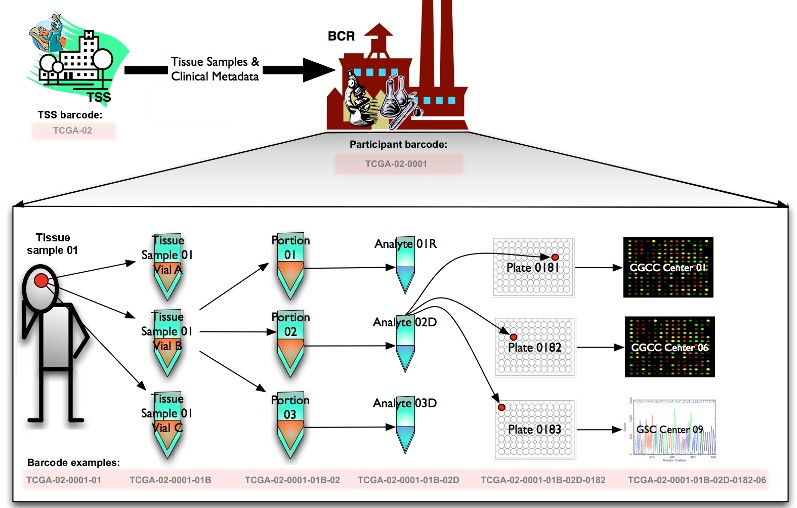
图中总结了TCGA中从样品到数据处理流程:
BCR从TSS收到参与者的样本和他们相关的元数据。然后BCRs分配人可读的IDs(barcode),也就是TCGA barcode给参与者的元数据和样本。TCGA barcode用来把扩展到整个TCGA网络中的数据联系在一起,因为IDs可以唯一识别一个特定样本的一组结果。关于BCR TSS等组织机构信息见:https://www.omicsclass.com/article/1077
barcode编码各部分介绍:

| Label | Identifier for | 解释 |
| Project | Project name | 来自哪个项目: 如TCGA、TARGET等等 |
| TSS | Tissue source site | 样品来自哪个组织机构:01 代表International Genomics Consortium, 更多见:TSS |
| Participant | Study participant | 样品唯一编号(可以理解为一个病人唯一编号) |
| Sample | Sample type | 样品来自人体组织类型,如:01代表Primary Solid Tumor, 更多见:SampleType |
| Vial | Order of sample in a sequence of samples | 一份样品被分割成好几份,表示第几份,通常是A-Z编号 |
| Portion | Order of portion in a sequence of 100-120 mg sample portions | 每份样品再分割成不同的小样品:01-99等等编号,代表第几份 |
| Analyte | Molecular type of analyte for analysis | 实验数据来源分子类型,如R代表 RNA,D代表DNA等等,更多见:Portion / Analyte Codes |
| Plate | Order of plate in a sequence of 96-well plates | 96孔序列中板的顺序,4个数字组成 |
| Center | Sequencing or characterization center that will receive the aliquot for analysis | 数据由哪个机构分析:如 01代表The Broad Institute GCC,更多见:Center |
更多barcode类型总结:
上面提到的barcode编码,只是barcode的一种组织类型,更多类型的barcode见下图,一个病人除了他的组织样编号,还有他的临床信息编号,如,用药,化疗信息等等,这些编号的共同点是前面的TSS标号,Participant编号是一致的。
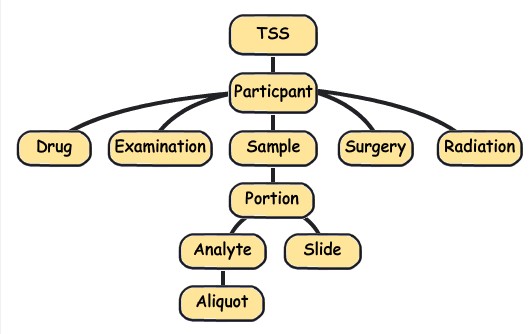
下表显示了不同barcode,所代表的不同意义:层次结构级别:
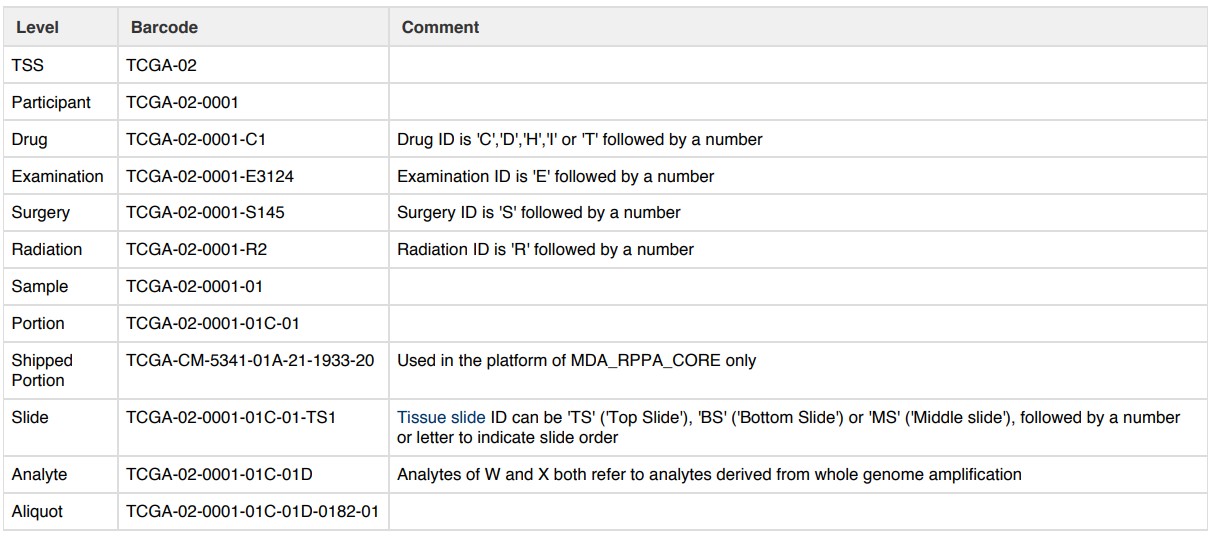
我觉得区分样品最总要的就是以下几部分,大家觉得呢?
延伸阅读
更多生物信息课程:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程、基因家族文献思路解读
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘、转录组文献解读
5. 微生物16S/ITS/18S分析原理及结果解读、OTU网络图绘制、cytoscape与网络图绘制课程
6. 生物信息入门到精通必修基础课:linux系统使用、biolinux搭建生物信息分析环境、linux命令处理生物大数据、perl入门到精通、perl语言高级、R语言画图、R语言快速入门与提高、python语言入门到精通
7. 医学相关数据挖掘课程,不用做实验也能发文章:TCGA-差异基因分析、GEO芯片数据挖掘、 GEO芯片数据不同平台标准化 、GSEA富集分析课程、TCGA临床数据生存分析、TCGA-转录因子分析、TCGA-ceRNA调控网络分析
8.其他,二代测序转录组数据自主分析、NCBI数据上传、二代fastq测序数据解读、
9.全部课程可点击:组学大讲堂视频课程
- 发表于 2019-10-23 17:26
- 阅读 ( 22320 )
- 分类:TCGA
你可能感兴趣的文章
相关问题
- 请教一下TCGA的蛋白数据怎么理解,它是有正负值的,这个正负值代表什么意思,为什么蛋白表达会有负值?我只想给它分成高表达,低表达和表达缺失组应该怎么处理呀? 0 回答
- TCGA下载 docker报错 0 回答
- TCGA差异分析如何根据临床信息分组? 1 回答
- TCGA用R数据下载错误 1 回答
- 老师,想请教一下TCGA基因表达数据的问题,我从xena.ucsc网页上下载了基因表达数据TCGA-CESC.htseq_counts.tsv;然后发现该数据中只有Ensembl格式的基因ID ,没有SYMBOL格式的。所以接下来进行基因ID格式转换,却发现同一个SYMBOL ID对应的多个Ensembl格式的ID,想问下老师,这种情况该怎么处理?同一个SYMBOL ID所对应的多个Ensembl格式ID的基因表达数据应该留下哪一个? 1 回答
- TCGA的RNA-seq count annotation过滤,有些不知道是否需要删除 0 回答
