微生物组间差异分析之LEfSe
LEfSe分析,可以分析组间菌群差异,找出各组间差异的微生物种类,有助于开发biomaker等研究,因此LEfSe分析在微生物相关文章中经常出现。我们今天来详细讲解一下LEfSe分析的原理及图表解读。
LEfSe分析原理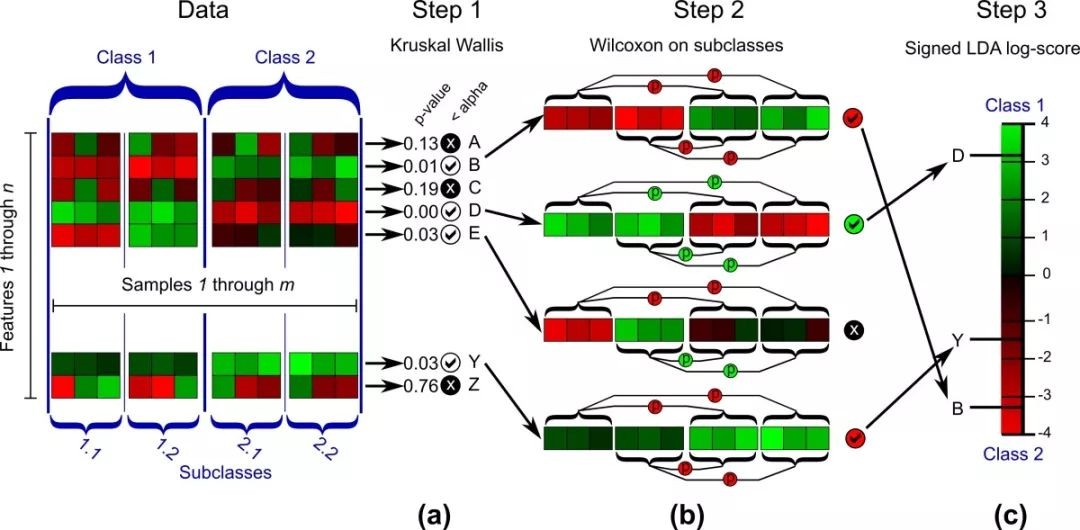
运行LEfSe软件主要分三大步骤:第一步:需要把普通的物种、基因等等的丰度信息的表格转化成LEfSe识别的格式。这一步会生成.in结尾的文件
第二步:这一步也是最关键的一步,统计显著差异的biomarker、统计子组组间差异、统计effect sizes(LDA score),会生成.res格式的文件。如下图所示
Step1:两组或两组以上的样本中采用的非参数因子Kruskal-Wallis秩和检验检测出biomarker。
Step2:基于上步的显著差异物种基因,进行两两组之间的Wilcoxon秩和检验,检测出组间差异。
Step3:线性判别分析(LDA)对biomarker进行评估差异显著的物种的影响力(即LDA score),最终获得biomarker。
第三步:基于第二大步的数据,绘制各种图片。
前两步的Kruskal-Wallis秩和检验、Wilcoxon秩和检验 比较简单,类似T检验或者方差检验等,只不过T检验和方差分析为参数检验(要求数据符合方差齐性、正态分布),而在微生物多样性分析中,样品物种丰度分布不确定,多采用非参数检验,所以采用非参数的Kruskal-Wallis秩和检验、Wilcoxon秩和检验。比较复杂一点的就是最后的LDA分析。
LDA是一种监督学习的降维技术,也就是说其数据集中的每个样本是有类别输出的。是在目前机器学习、数据挖掘领域经典且热门的一个算法这点和PCA不同。PCA是不考虑样本类别输出的无监督降维技术。LDA是有监督的,所以LDA算法可以很好的利用样本的分组信息,得到的结果更可靠,这就是LDA分析优势。理解了LDA分析的原理,就不难理解LEfSe的分析结果了。
LDA分析原理:
LDA是一种经典的降维方法线性判别分析(Linear Discriminant Analysis)。LDA的思想可以用一句话概括,就是“投影后类内方差最小,类间方差最大”。什么意思呢? 我们要将数据在低维度上进行投影,投影后希望每一种类别数据的投影点尽可能的接近,而不同类别的数据的类别中心之间的距离尽可能的大。
可能还是有点抽象,我们先看看最简单的情况。假设我们有两类数据 分别为红色和蓝色,如下图所示,这些数据特征是二维的,我们希望将这些数据投影到一维的一条直线,让每一种类别数据的投影点尽可能的接近,而红色和蓝色数据中心之间的距离尽可能的大。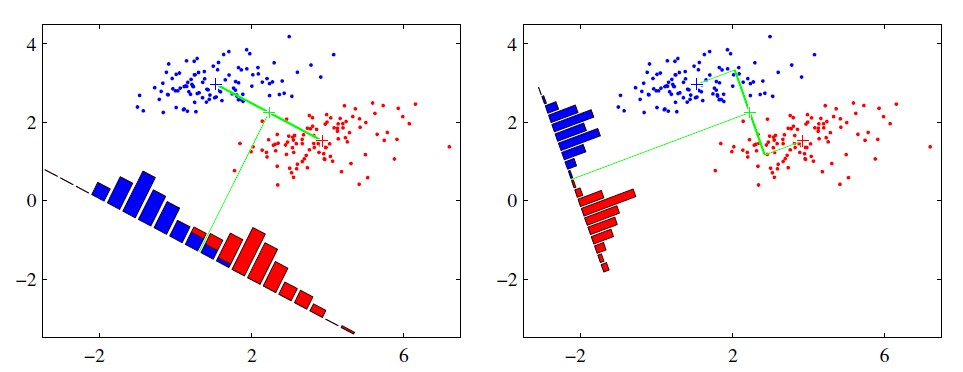
上图中提供了两种投影方式,哪一种能更好的满足我们的标准呢?从直观上可以看出,右图要比左图的投影效果好。因为右图的黑色数据和蓝色数据各个较为集中,且类别之间的距离明显,而左图则在边界处数据混杂。以上就是LDA的主要思想了,当然在实际应用中,我们的数据是多个类别的,我们的原始数据一般也是超过二维的,投影后的也一般不是直线,而是一个低维的超平面。
LEfSe分析结果:LDA值分布柱状图:
图中展示了LDA Score大于设定值的物种(less_strict 设为2;more_strict 设为4),即组间具有统计学差异的Biomarker。展示了不同组中丰度差异显著的物种,柱状图的长度代表差异物种的显著性(即为 LDA Score)。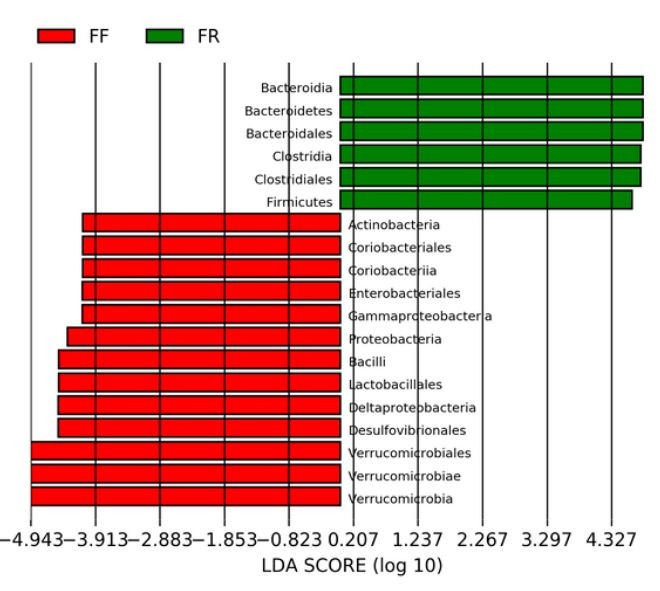
进化分支图:
在进化分支图中,由内至外辐射的圆圈代表了由界(单个圆圈)至属(或种)的分类级别(不同的分类水平下圆圈的层数不同,下图为order水平下进化图,所以有4层)。在不同分类级别上的每一个小圆圈代表该水平下的一个分类,小圆圈直径大小与相对丰度大小呈正比。着色原则:无显著差异的物种统一着色为黄色,差异物种 Biomarker跟随组进行着色,红色节点表示在红色组别中起到重要作用的微生物类群,绿色节点表示在绿色组别中起到重要作用的微生物类群,其它圈颜色意义类同。图中英文字母表示的物种名称在右侧图例中进行展示。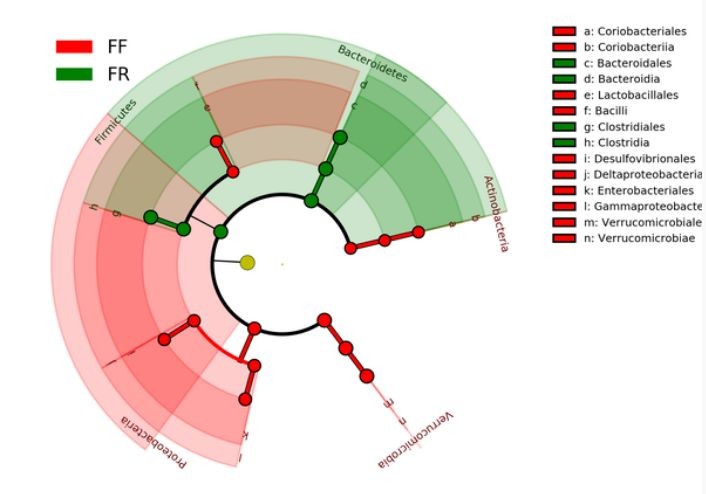
课程推荐:微生物扩增子分析课程实操 微生物16S/ITS/18S分析原理及结果解读
延伸阅读
微生物测序原理|肠道君|什么是OTU|alpha多样性|Beta多样性|GraPhlAn树状图|OTU网络图MENA

- 发表于 2018-05-17 21:52
- 阅读 ( 33590 )
- 分类:宏基因组
你可能感兴趣的文章
- lefse 运行报错python3中 1473 浏览
