STRING蛋白互作数据库在线分析基因互作网络(PPI)
STRING蛋白互作数据库在线分析网络
今天为大家介绍一个有名的搜索蛋白互作网络的数据库:STRING。教大家如何制作一个蛋白互作网络
STRING数据库介绍:
nString数据库是一个搜索已知蛋白质之间和预测蛋白质之间相互作用的数据库,该数据库可应用于2031个物种,包含960万种蛋白和1380万中蛋白质之间的相互作用。它除了包含有实验数据、从PubMed摘要中文本挖掘的结果和综合其他数据库数据外,还有利用生物信息学的方法预测的结果。
n研究蛋白之间的相互作用网络,有助于挖掘核心的调控基因,目前已经有很多的蛋白质相互作用的数据库,而string绝对是其中覆盖的物种最多,相互作用信息做大的一个,网址如下:
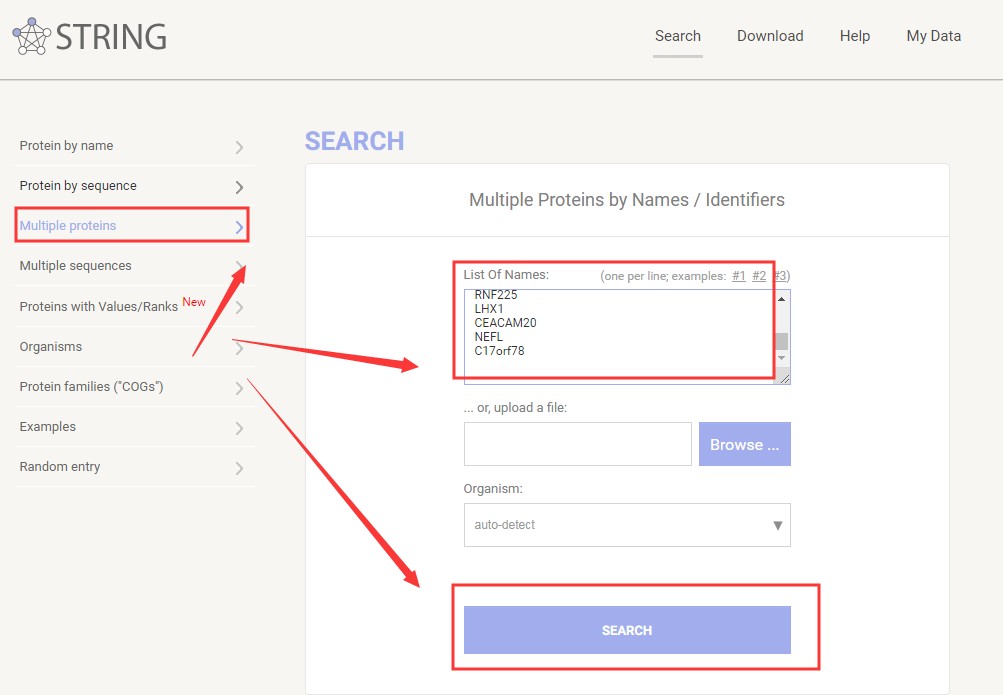
1.已知单个基因查找互作网络
首先,通过浏览器进入STRING数据库,它的主页面是这样的:
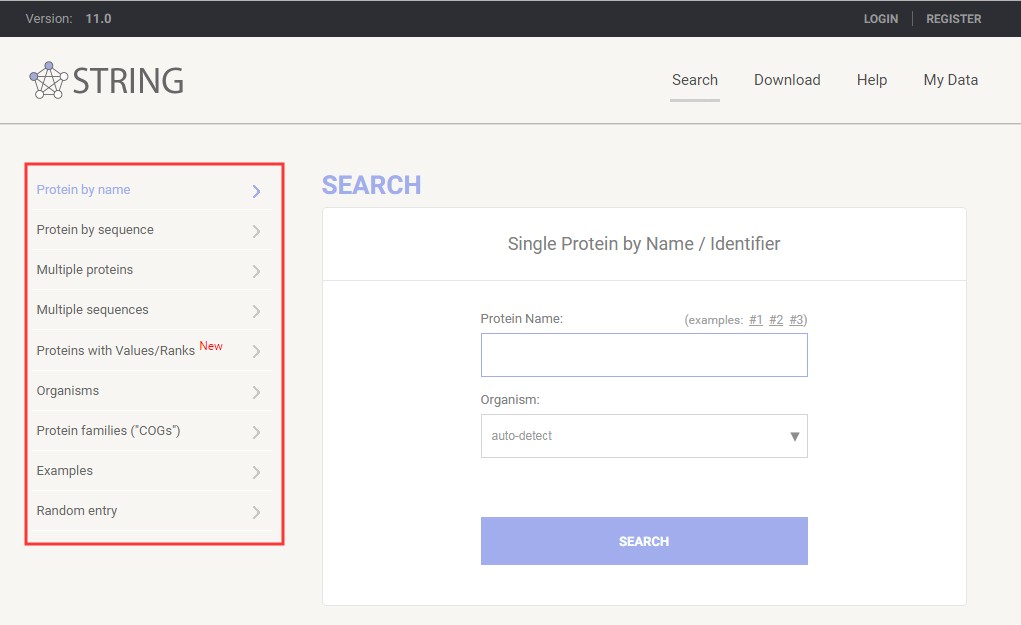
它可以选择不同方式去查找互作网络(上图左边红框框可分别点击),比如可通过输入单个蛋白的名称(或氨基酸序列)查找其互作网络,也可输入多个蛋白的名称或序列,查找他们之间的互作关系等。
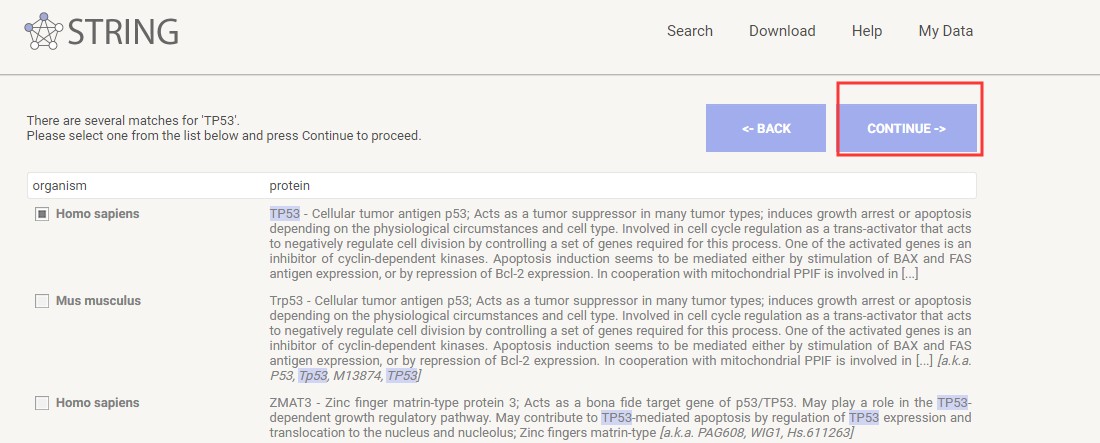
这里就以抑癌基因TP53为例,查找一下它的互作网络。Protein Name这里输入TP53,物种选人类,点SEARCH按钮。
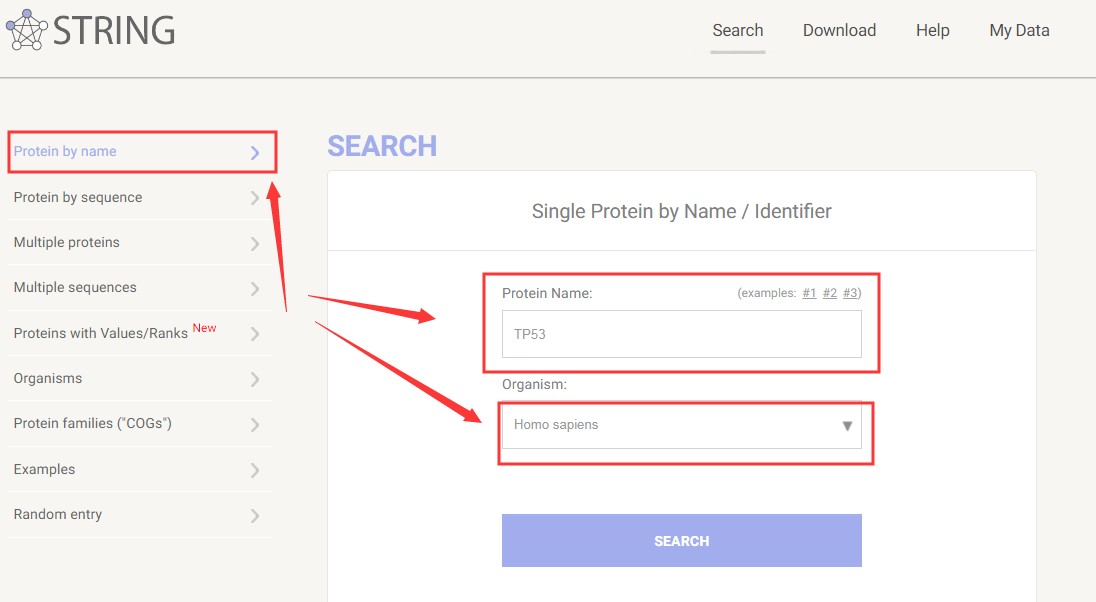
点击search,很快就得到结果,在Viewers为Network模式下,彩色的“玻璃球 “(或称为节点,Node)表示具直接作用的蛋白,“玻璃球 “之间的不同颜色样式的连线(或称为边,edge)表示不同的作用类型,具体的图例见下图。提示不同物种的TP53,第一个人类continue就行
 得到相关的蛋白互作网络图:
得到相关的蛋白互作网络图:
 点击图中的“玻璃球“可以查看蛋白质相关信息,空的”玻璃球“表示暂无该蛋白的3D结构;点击连线可以查看相互作用信息。
点击图中的“玻璃球“可以查看蛋白质相关信息,空的”玻璃球“表示暂无该蛋白的3D结构;点击连线可以查看相互作用信息。
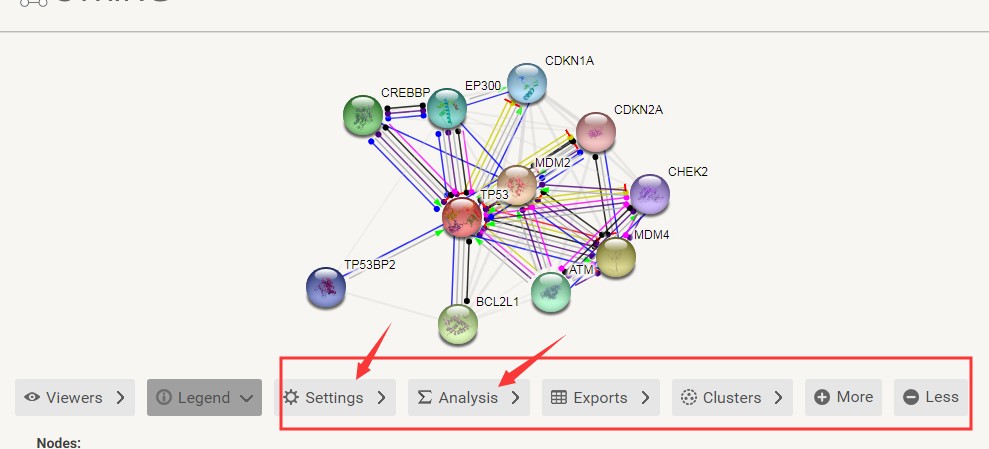
在Setting列表,可设置“连线“的展示类型以及互作数据的来源,“连线“的展示模式这里选择molecular action,如下图。
在Analysis列表,除了给出互作网络的相关信息,还给出了相应的GO和KEGG富集分析结果,感兴趣的话也可以将分析结果下载下来(如下图)。
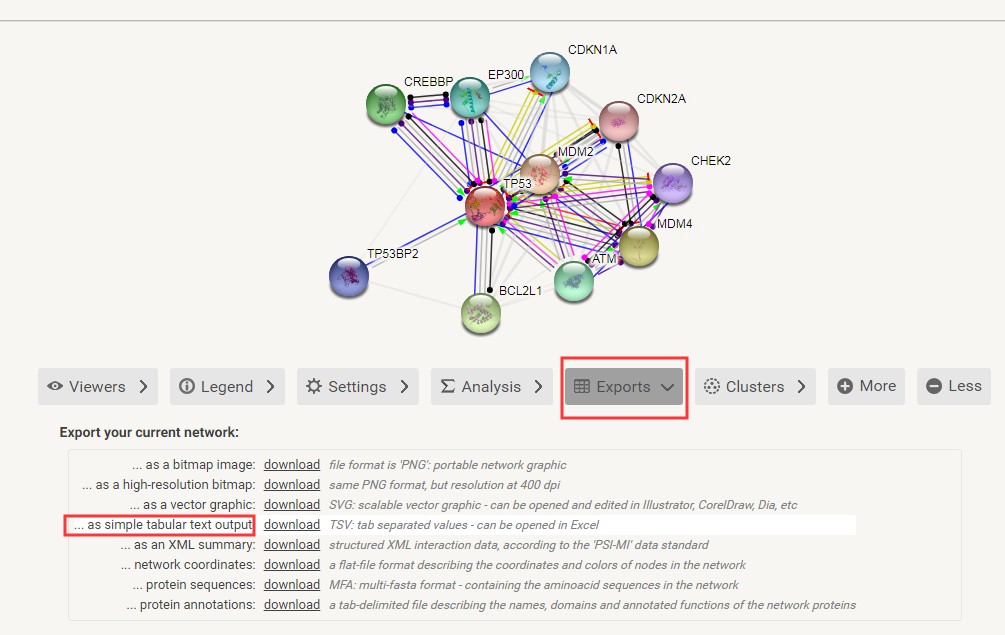
在Exports列表,可导出(download)两种格式(png和svg)的网络图(如下图),若果觉得这里的“玻璃球“比较丑,也可以表格的形式导出文本文件(tsv格式)。
2.基因集(蛋白集)间的互作关系
回到分析主界面,如下图所示,填上基因集,然后搜索,如果有提示,选择在人类中搜索:
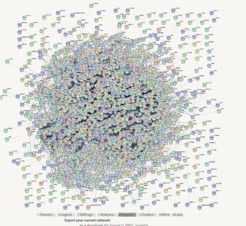
由于我输入了1400个基因,最后会得到一个庞大复杂的网络,网络太复杂不利于我们查看与分析,可以将网络,导出到cytoscape中分析查找子网络。
- 发表于 2019-12-02 14:42
- 阅读 ( 30898 )
- 分类:软件工具
你可能感兴趣的文章
- ppi_network.r 蛋白互作网络分析 3827 浏览
- 鉴定domain同时分析蛋白互作 8668 浏览
0 条评论
请先 登录 后评论