如何将SNP标记转化成可跑胶验证的CAPS标记
现如今,基于高通量测序得到的SNP信息无疑是海量的,然而SNP标记的验证却并不容易实现。其最好的验证方式是一代sanger测序,不过价格较高,而且通量低。如果SNP刚好位于酶切位点上,就可以将其转化为CAPS标记,利用跑胶的方式对其进行验证,既便宜又好用。
CAPS简介
CAPS实质上是基于SNP的PCR技术与限制性内切酶(Restriction Endonuclease RE)技术结合的分子标记技术,简单理解就是某SNP恰好位于酶切位点上,通过对该SNP位点设计引物,经PCR后获得的相应产物片段经酶切、电泳等步骤,最终通过观察不同的条带判断样本的多态性。
CAPS的工作原理
如下图所示:
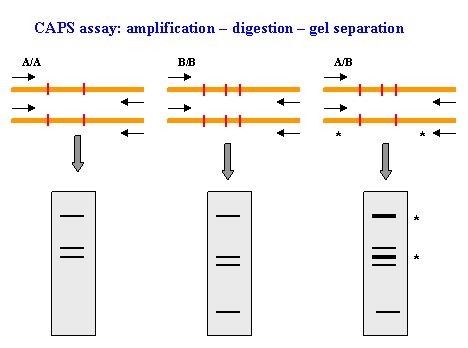
使用特异性的PCR引物来扩增来自两个相关个体(例如,来自两种不同表型),A / A和B / B以及杂合子A / B的特定DNA序列。 来自A / A和B / B的PCR扩增片段分别含有两个和三个RE识别位点。而 在杂合子A / B中,将获得两种不同的PCR产物,一种被切割三次,另一种切割两次。 当通过琼脂糖或丙烯酰胺凝胶电泳分离时,由RE切割的PCR产物将容易区分出三种不同的模式。
CAPS的优势
引物与限制性内切酶组合非常多,增加了揭示多态性的机会,而且操作简便,可用琼脂糖电泳分析;
在真核生物中,CAPS标记呈共显性,即可区分纯合基因型和杂合基因型;
所需DNA的量很少,且对DNA的浓度要求不严格;
使用的引物较长,扩增的结果比较稳定且避免了RFLP分析中的膜转印这一步骤,又能保持RFLP分析的精确度。另外,由于很多限制性内切酶均可与扩增DNA酶切,所以检测到多态性机会较大。
操作简便、快捷和自动化程度高。
由于具有这些优势,CAPS标记技术已成为现在生物学研究的一个非常重要的分子标记技术。其在种质鉴定、辅助育种、基因鉴定和图谱构建等领域得到相当广泛的应用。
- 发表于 2018-05-18 08:46
- 阅读 ( 10757 )
- 分类:实验相关
