用R语言进行KM生存分析
R是数据分析常用的软件之一,通过各种功能强大的R包,可以简单方便的实现各种分析。在R语言中,能够进行生存分析的R包很多,survival和survminer是其中最基本的两个,survival负责分析,survimner负责可视化,二者相结合,可以轻松实现生存分析。具体过程如下
1. 准备生存数据
对于每个个体而言,其生存数据会出现两种情况,第一种是观测到生存时间,通常用1表示,第二种则是删失。通常用0表示。survival自带了一个测试数据lung, 内容如下所示
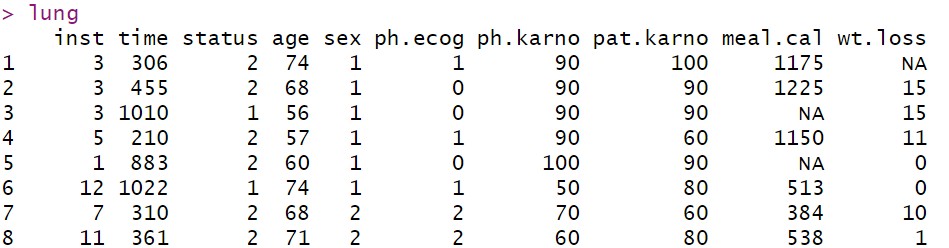
每一行代表一个样本,time表示生存时间,status表示删失情况,这里只有1和2两种取值,默认排序后的第一个level对应的值为删失,这里则为1表示删失。其他列为样本对应的性别,年龄等基本信息。
2. 进行生存分析
这里根据性别这个二分类变量,采用KM算法来估计生存曲线,代码如下(summary结果只显示部分)
> library("survival")
> library("survminer")
> kmfit<-survfit(Surv(time, status) ~ sex, data = lung)
> summary(kmfit)
Call: survfit(formula = Surv(time, status) ~ sex, data = lung)
sex=1
time n.risk n.event survival std.err lower 95% CI upper 95% CI
11 138 3 0.9783 0.0124 0.9542 1.000
12 135 1 0.9710 0.0143 0.9434 0.999
13 134 2 0.9565 0.0174 0.9231 0.991
15 132 1 0.9493 0.0187 0.9134 0.987
26 131 1 0.9420 0.0199 0.9038 0.982
30 130 1 0.9348 0.0210 0.8945 0.977
从kmfit中summary可以看到已经包含了每个时间点的生存概率,删失等信息,通过这些信息,完全可以自己写代码来画图。为了方便,我们直接采用survminer中的函数来进行可视化。
3. 分析结果的可视化
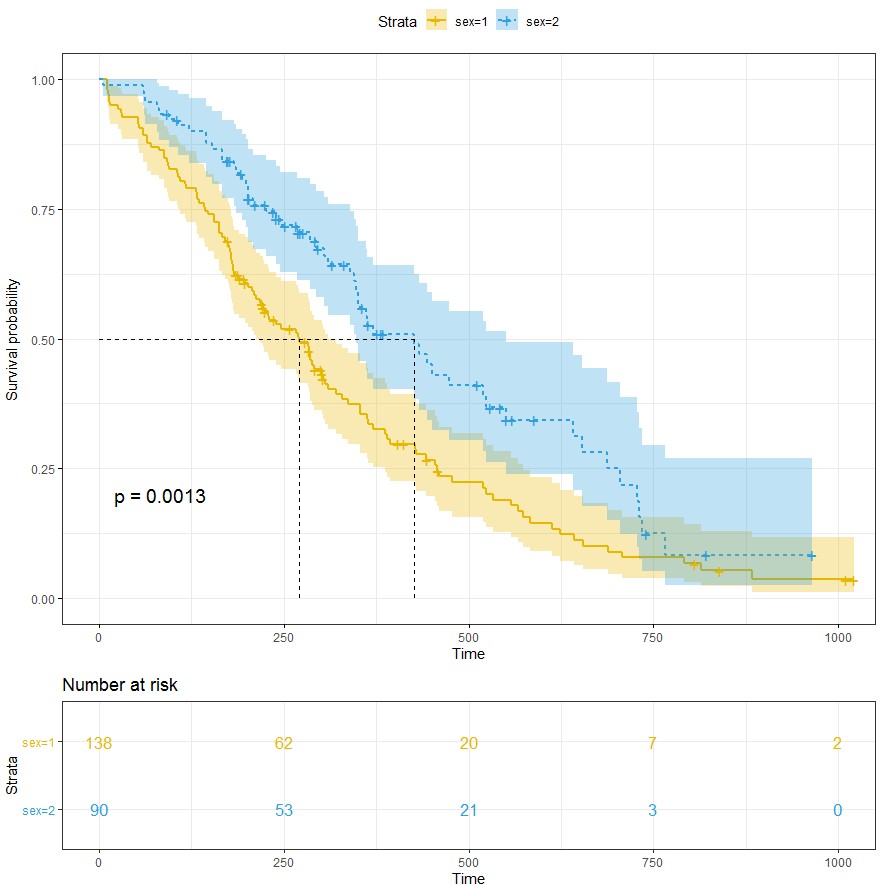
最基本的可视化方式如下
library("survminer")
ggsurvplot(kmfit,
pval = TRUE, conf.int = TRUE,
risk.table = TRUE,
risk.table.col = "strata",
linetype = "strata",
surv.median.line = "hv",
ggtheme = theme_bw(),
palette = c("#E7B800", "#2E9FDF"))
更多生物信息课程:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程、基因家族文献思路解读
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘、转录组文献解读
5. 微生物16S/ITS/18S分析原理及结果解读、OTU网络图绘制、cytoscape与网络图绘制课程
6. 生物信息入门到精通必修基础课:linux系统使用、perl入门到精通、perl语言高级、R语言画图
7. 医学相关数据挖掘课程,不用做实验也能发文章:TCGA-差异基因分析、GEO芯片数据挖掘、 GEO芯片数据不同平台标准化 、GSEA富集分析课程、TCGA临床数据生存分析、TCGA-转录因子分析、TCGA-ceRNA调控网络分析
8.其他,二代测序转录组数据自主分析、NCBI数据上传、二代测序数据解读、
9.更多课程可点击:组学大讲堂视频课程
- 发表于 2019-12-06 21:13
- 阅读 ( 5648 )
- 分类:TCGA
你可能感兴趣的文章
- Cox与KM生存分析及结果解读 37553 浏览
- KM-plotter在线生存分析 14587 浏览
