微生物多样性16S计算物种累计曲线
R语言 vegan包计算物种累计曲线
R语言 vegan包计算物种累计曲线
vegan 包是进行群落数据分析最常用的R包,其中的 specaccum 函数用来计算物种的累计曲线。
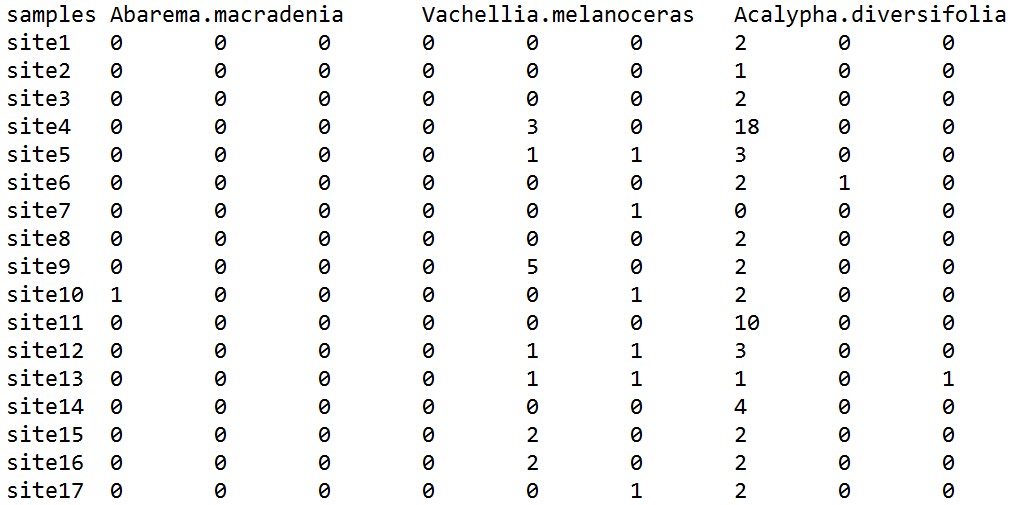
输入数据,otutable:列表示不同的物种,行表示不同的采样点,中间的数字代表物种丰度:
示例代码:
require(vegan)
otu <- read.table("otu_table.txt", header = T,
sep = "\t",row.names=1)
all <- specaccum(otu, method = "random")
png(file="spe.png",h=2000,w=2000,res=300)
plot(all, ci.type = "poly", col = "blue", lwd = 2, ci.lty = 0,
ci.col = "lightblue", main = "Species Accumulation Curves", xlab = "Number of samples",
ylab = "Number of species")
boxplot(all, col = "yellow", add = TRUE, pch = "+")
dev.off()
绘图结果:
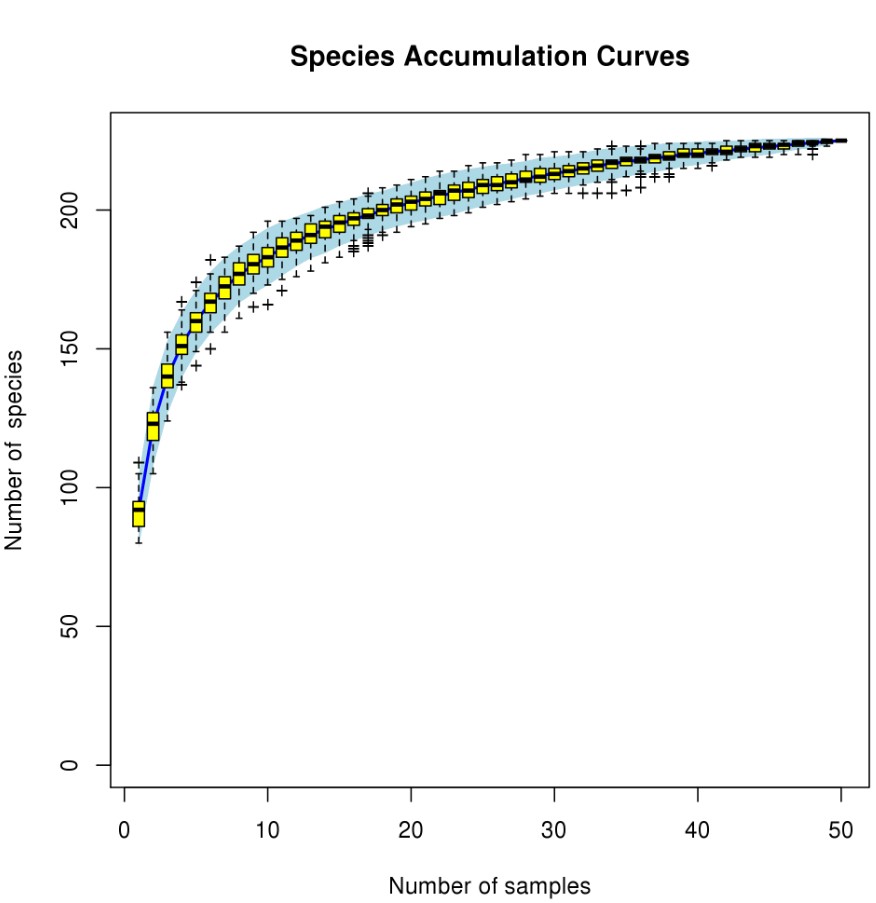
最终的物种累计曲线中,横坐标是样本个数,纵坐标是发现的物种个数,随着样本个数的增加,发现的物种个数也不断增加;物种累计曲线反应的就是抽样个数对物种多样性的影响;如果曲线末端部分呈现 急剧上升的趋势,表明抽样量不足;增加样本量,还能继续发现新的物种;当曲线末端上升趋势趋于平缓时,则表明采样量足够。
示例数据下载:otu_table.rar
生物信息入门到精通必修基础课:linux系统使用、biolinux搭建生物信息分析环境、linux命令处理生物大数据、perl入门到精通、perl语言高级、R语言画图、R语言快速入门与提高、python语言入门到精通
- 发表于 2020-01-15 13:38
- 阅读 ( 8042 )
- 分类:宏基因组
你可能感兴趣的文章
- corrplot包绘制相关性图,简单又好看! 90 浏览
- 已知集合交集数量绘制韦恩图 382 浏览
- 绘制Upset 图 1671 浏览
- R公式中如何加入变量 755 浏览
- R语言基础入门—数据结构(2) 774 浏览
- R语言基础入门—数据结构(1) 723 浏览
1 条评论
请先 登录 后评论
