如何使用NCBI进行blast比对
BLAST是生命科学研究中常用的一套在蛋白质数据库或核酸数据库中进行序列相似性比对的一套分析工具。英文全称是Basic Local Alignment Search Tool。NCBI作为生命科学研究领域使用最为广泛的数据库之一,深受广大科研工作者青睐。今天,小编就给大家简单介绍一下NCBI上的blast比对。
NCBI上的blast分为四种:Nucleotide BLAST:核酸比核酸;blastx:核酸比蛋白;blastn:蛋白比核酸;Protein BLAST:蛋白比蛋白。
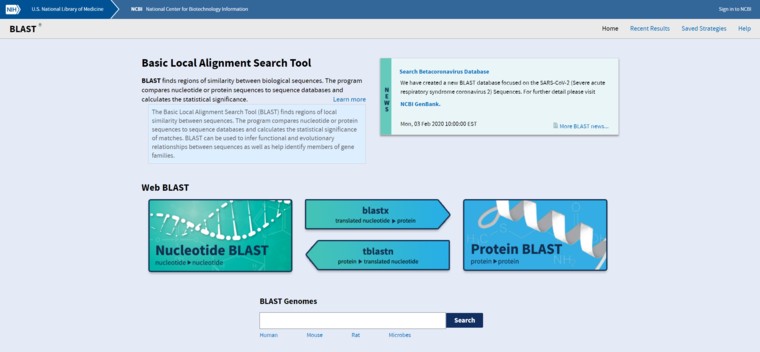
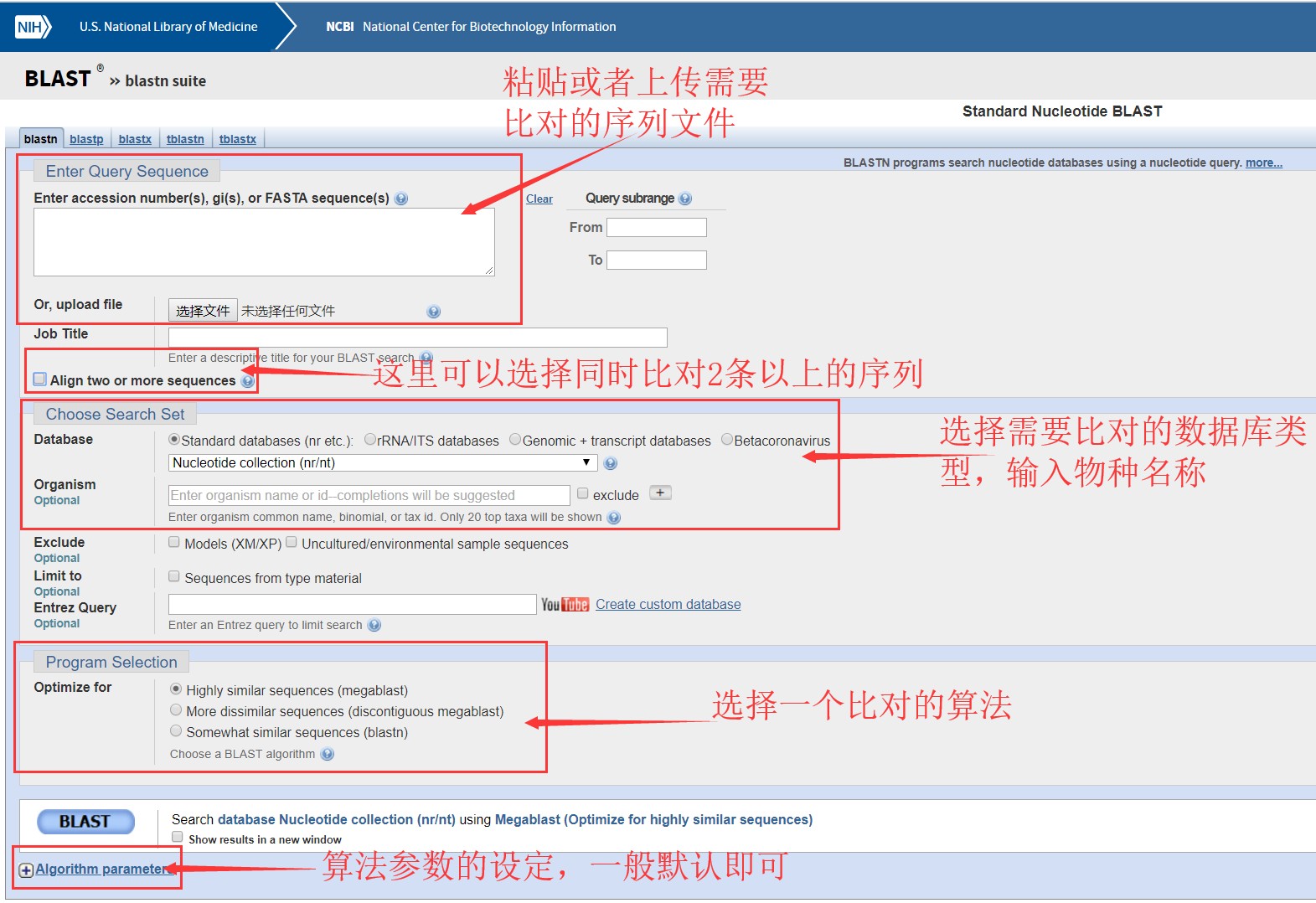
在这里小编以Nucleotide BLAST的操作为例,演示一下如何进行序列比对。点击上图中“Nucleotide BLAST”,打开比对界面,粘贴或者上传需要比对的序列,选择数据库、填入物种,调整各个参数(一般默认即可),点击BLAST进行比对。需要注意的是,在参数调整时,E值是最重要的参数,可以根据情况进行适当的调整。
比对之后,就可以查看比对结果了。这里小编以小鼠为例,为大家简单介绍一下比对结果。提交BLAST比对之后,稍等片刻(时间可长可短,与提交的序列有关),即可得到如下的比对结果页面。
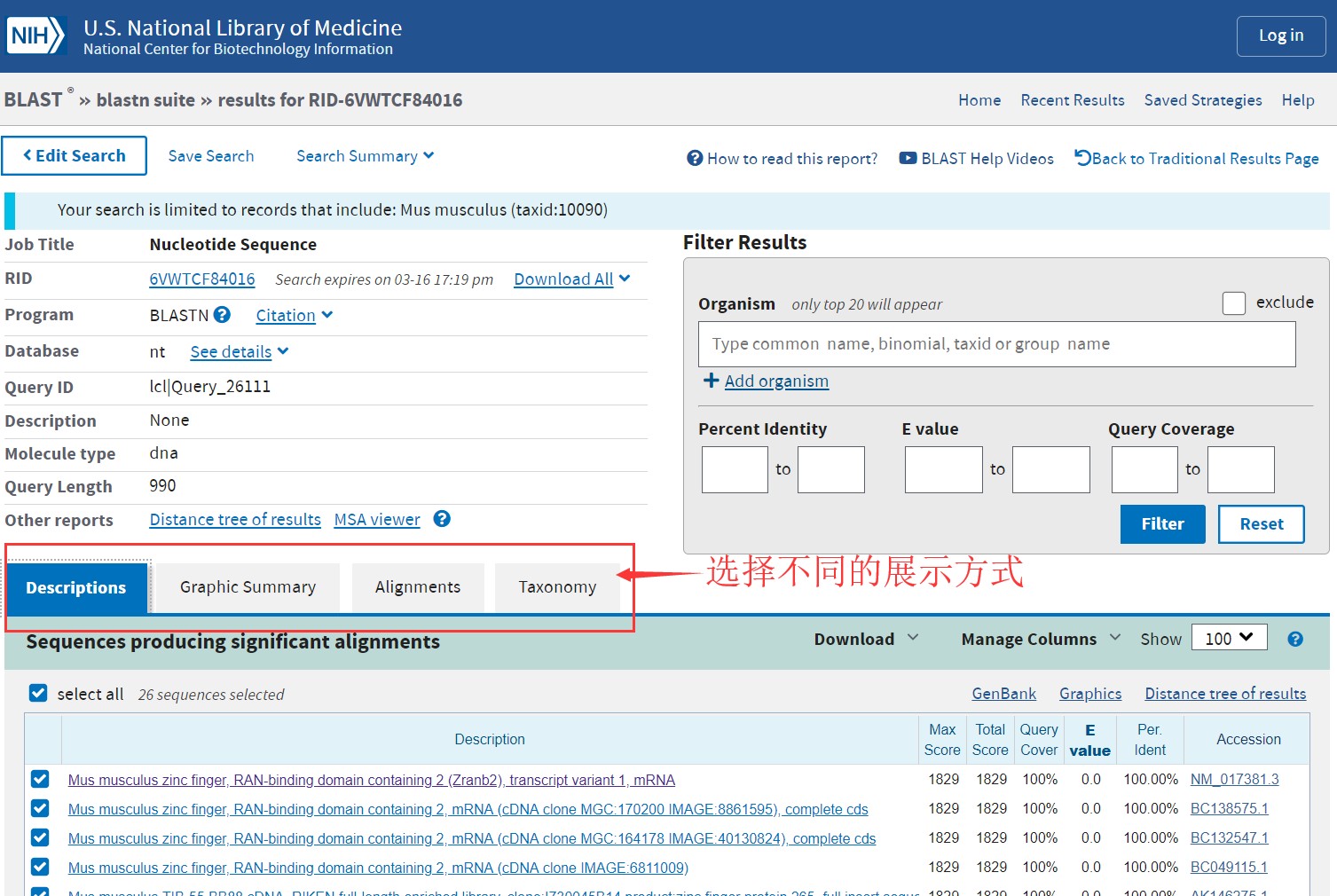
在查看比对结果时,E值以及Score值最重要,E值越小、Score值越大,则比对结果越可靠。
在Graphic summary界面下,每一条线段代表一个比对结果,鼠标点击会有相应的比对信息。
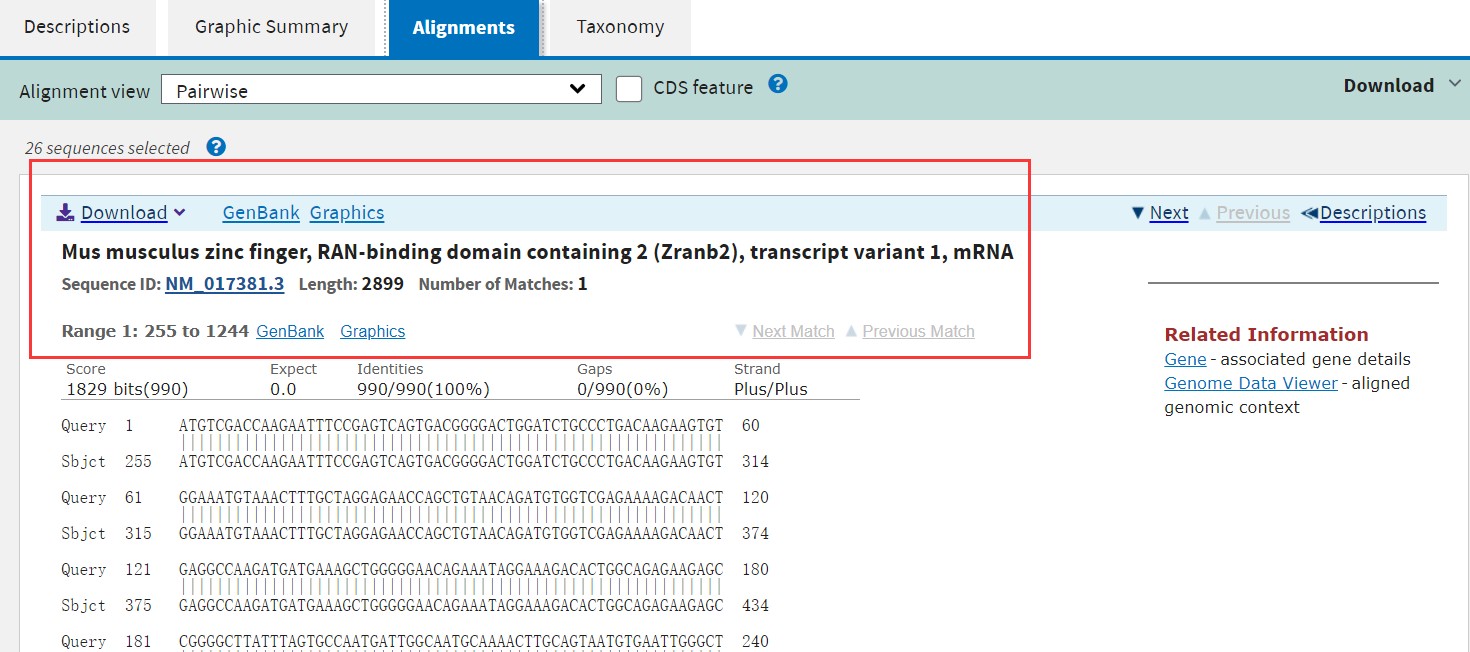
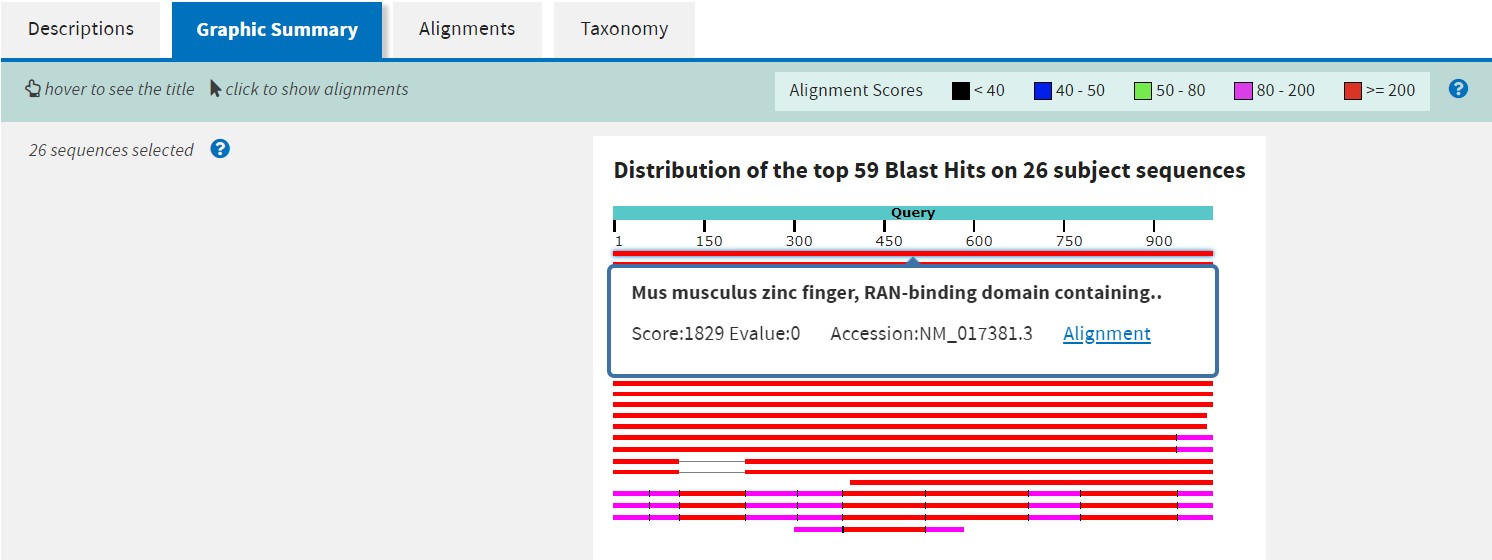 在Alignments界面下,有每一个比对结果的多序列比对结果展示。点击“Download”可以下载比对结果,也可以点击“gene bank”了解比对结果的详细信息。
在Alignments界面下,有每一个比对结果的多序列比对结果展示。点击“Download”可以下载比对结果,也可以点击“gene bank”了解比对结果的详细信息。
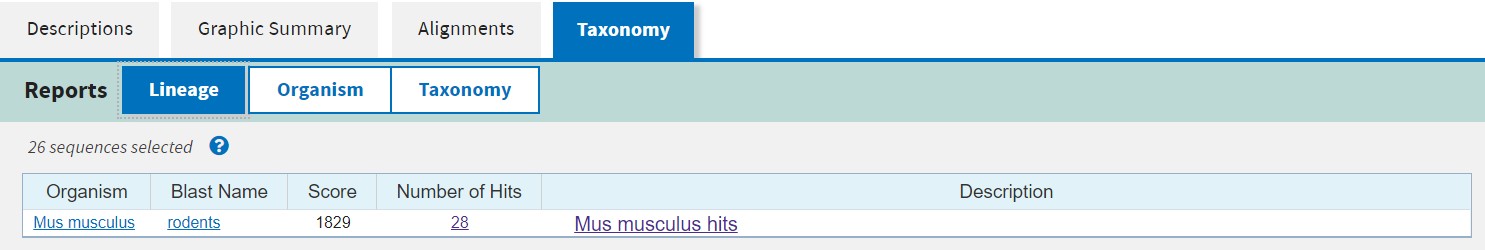
 Taxonomy界面主要告诉我们所比对的序列在分类学上是什么物种。
Taxonomy界面主要告诉我们所比对的序列在分类学上是什么物种。
到这里,比对结果就展示完了。但是,如果您还不理解某些参数的具体含义,也不要紧。NCBI也有对BLAST的官方说明,具体可以参考“BLAST topics”,网址如下:https://blast.ncbi.nlm.nih.gov/Blast.cgi?CMD=Web&PAGE_TYPE=BlastDocs&DOC_TYPE=BlastHelp#expect
总的来说,技巧虽小,但操作不易,每一步都有各种参数需要调整。在实际分析中要学会因地制宜、因时制宜,根据不同的情况选择不同的参数,这样才能得到最好的结果。每一个数据库或者软件都有说明文档,在不知道如何操作时,要懂得寻求“help”,要有不怕出错的耐心和毅力。另外,我们组学大讲堂还有更多的生信分析技巧,具体可以扫描下方二维码进行选择学习。

更多生物信息课程:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程、基因家族文献思路解读
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘、转录组文献解读
5. 微生物16S/ITS/18S分析原理及结果解读、OTU网络图绘制、cytoscape与网络图绘制课程
6. 生物信息入门到精通必修基础课,学习链接:linux系统使用、perl入门到精通、perl语言高级、R语言画图
7. 医学相关数据挖掘课程,不用做实验也能发文章,学习链接:TCGA-差异基因分析、GEO芯片数据挖掘、GSEA富集分析课程、TCGA临床数据生存分析、TCGA-转录因子分析、TCGA-ceRNA调控网络分析
8.其他课程链接:二代测序转录组数据自主分析、NCBI数据上传、二代测序数据解读。
- 发表于 2020-03-20 16:26
- 阅读 ( 41551 )
- 分类:软件工具
你可能感兴趣的文章
- 探索基因大数据的利器——SRA Explorer 2483 浏览
- NCBI 基因组文件下载——datasets 2372 浏览
- 使用NCBI——Batch Entrez批量下载蛋白序列 11763 浏览
- 按物种拆分blast本地库 2945 浏览
- NCBI的SRA Lite和SRA Normalized数据有什么区别 7571 浏览
- blastn使用教程及结果详解 6457 浏览
