fastqc质控报告查看
1.Summary
为保证下游分析输入数据的可靠性,需要对下机的原始测序数据进行质控。通常我们会使用FASTQC软件对测序数据进行质控。fastqC会生成一个html的结果报告,下面是软件对质控结果进行判断:绿色代表PASS;黄色代表WARN;红色代表FAIL(当出现黄色时说明需要查看结果)。
fastq文件了解,可观看课程:https://study.163.com/course/courseMain.htm?share=2&shareId=400000000234009&courseId=1005231058

2.Basic Statistics
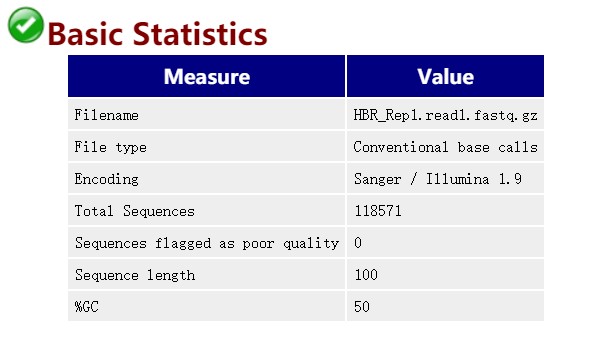
Filename:文件名
File type: 文件类型
Encoding:测序平台的版本和相应的编码版本号
Total Sequences: total reads的数量
Sequence length: 测序长度
%GC: GC含量,表示整体序列的GC含量,由于二代测序GC偏好性高,且深度越高,GC含量会越高。
3.Per base sequence quality:碱基质量统计
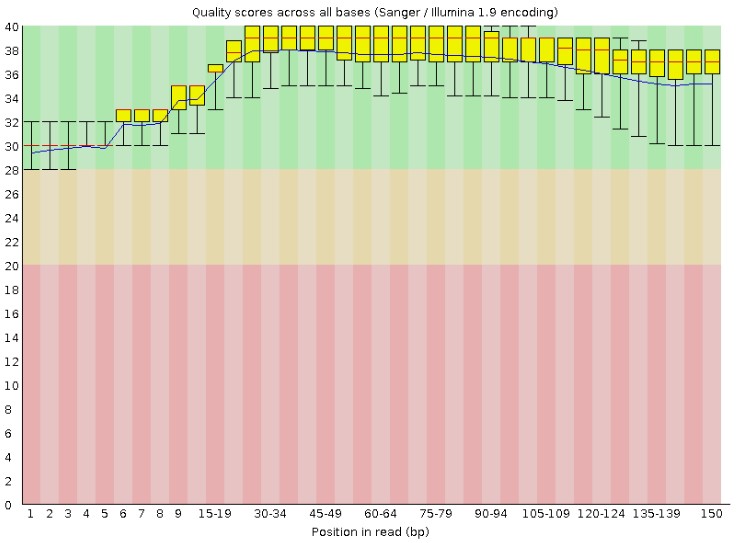
所有reads碱基的测序质量统计结果。箱线图中红色线表示中位数,黄色是25%-75%区间,延伸线是10%-90%区间,蓝线是平均数曲线。若任一位置碱基的下四分位数低于10或中位数低于25,报"WARN";若任一位置的下四分位数低于5或中位数低于20,报"FAIL"。
4.Per tile sequence quality
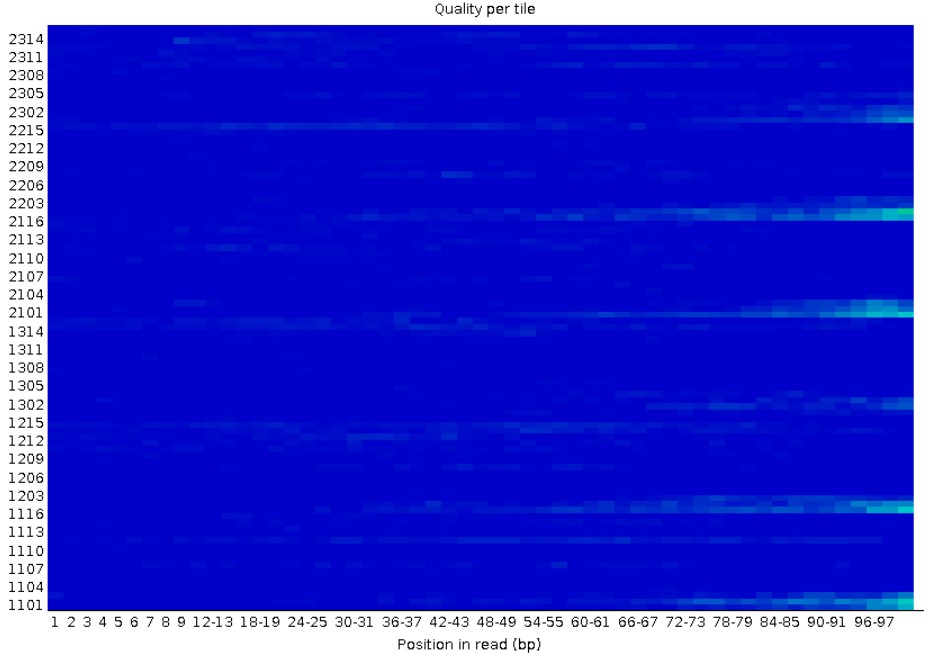 流通池中不同芯片(tile)的碱基测序质量平均值对比,显示了测序仪的系统差错。热图中蓝色部分是质量较好的点,红色越明显则是测序质量越低。纵坐标为tile编号,如果某tile的测序质量很低,可以考虑去除该tile的序列数据。
流通池中不同芯片(tile)的碱基测序质量平均值对比,显示了测序仪的系统差错。热图中蓝色部分是质量较好的点,红色越明显则是测序质量越低。纵坐标为tile编号,如果某tile的测序质量很低,可以考虑去除该tile的序列数据。
5.Per sequence quality scores
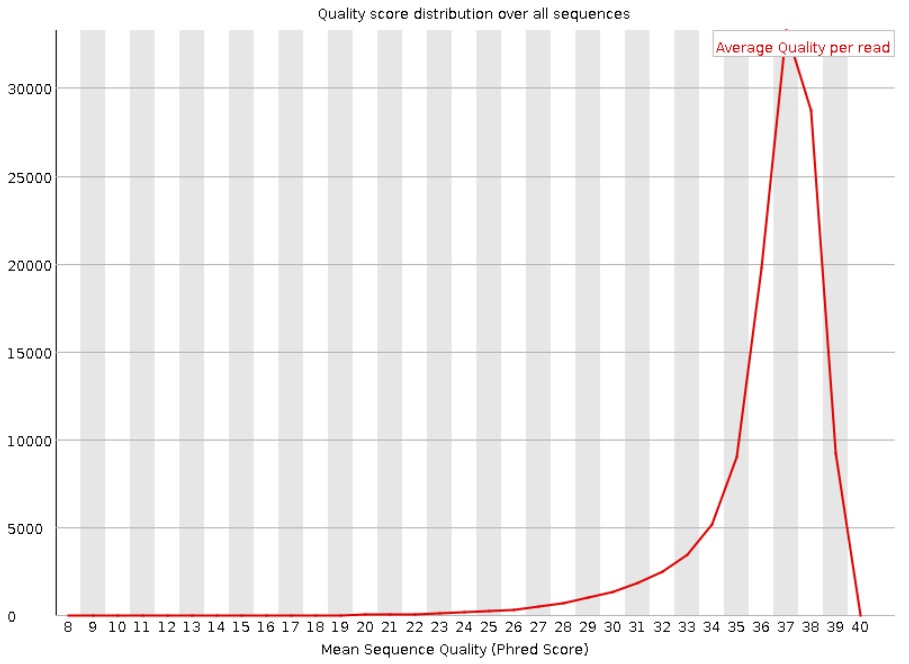 每条read的碱基质量均值的统计结果。横轴为测序质量quality,纵轴是read数目。从图中可以容易得看出不同质量范围内的read数量。其中当峰值也即最大read质量小于27(错误率0.2%)时报"WARN",当峰值小于20(错误率1%)时报"FAIL"。
每条read的碱基质量均值的统计结果。横轴为测序质量quality,纵轴是read数目。从图中可以容易得看出不同质量范围内的read数量。其中当峰值也即最大read质量小于27(错误率0.2%)时报"WARN",当峰值小于20(错误率1%)时报"FAIL"。
6.Per base sequence content
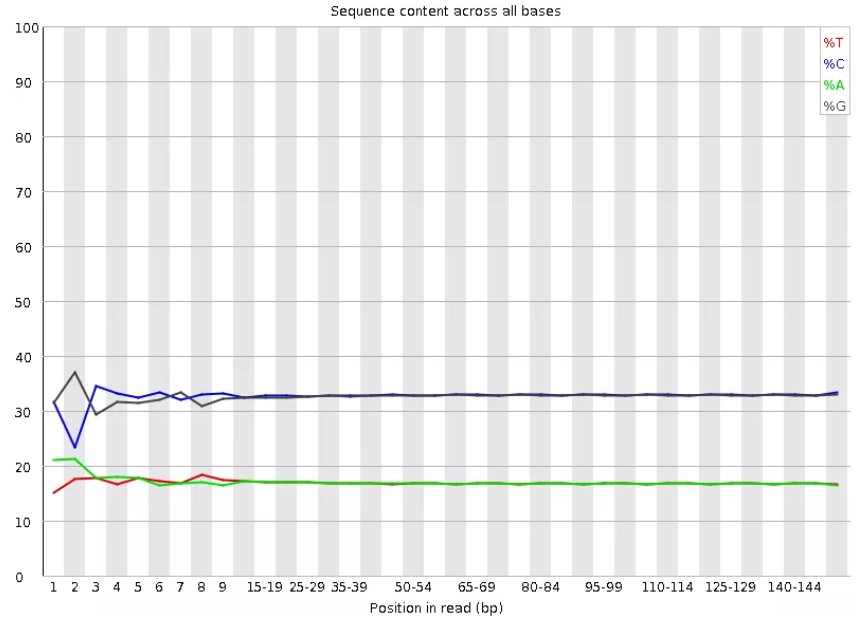 对所有reads的每一个位置,统计ATCG四种碱基(正常情况)比例的分布情况。横轴为碱基位置,纵轴为百分比。正常情况下四种碱基的出现频率应该是接近的,而且没有位置差异。因此好的样本中四条线应该平行且接近。当部分位置碱基的比例出现bias时,即四条线在某些位置纷乱交织,往往提示我们有overrepresentedsequence的污染。当所有位置的碱基比例一致的表现出bias时,即四条线平行但分开,往往代表文库有bias(建库过程或本身特点),或者是测序中的系统误差。当任一位置的A/T比例与G/C比例相差超过10%,报"WARN";当任一位置的A/T比例与G/C比例相差超过20%,报"FAIL"。
对所有reads的每一个位置,统计ATCG四种碱基(正常情况)比例的分布情况。横轴为碱基位置,纵轴为百分比。正常情况下四种碱基的出现频率应该是接近的,而且没有位置差异。因此好的样本中四条线应该平行且接近。当部分位置碱基的比例出现bias时,即四条线在某些位置纷乱交织,往往提示我们有overrepresentedsequence的污染。当所有位置的碱基比例一致的表现出bias时,即四条线平行但分开,往往代表文库有bias(建库过程或本身特点),或者是测序中的系统误差。当任一位置的A/T比例与G/C比例相差超过10%,报"WARN";当任一位置的A/T比例与G/C比例相差超过20%,报"FAIL"。
7.Per sequence GC content
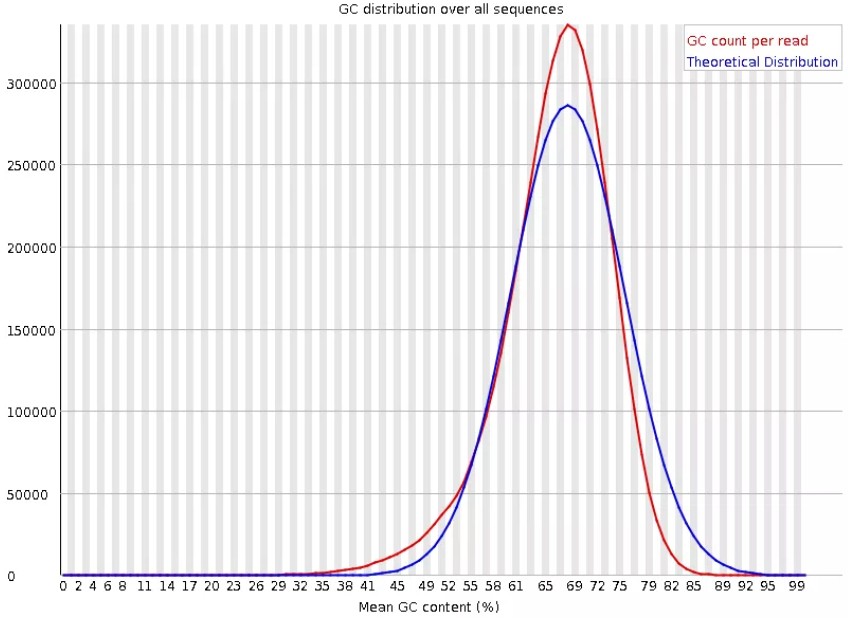 统计reads的平均GC含量的分布。横轴为GC比例,纵轴为reads数量。红线是实际情况,蓝线是理论分布(正态分布,均值不一定在50%,而是由平均GC含量推断的)。曲线形状的偏差往往是由于文库的污染或是部分reads构成的子集有偏差(overrepresentedreads)。形状接近正态但偏离理论分布的情况提示我们可能有系统偏差。偏离理论分布的reads超过15%时,报"WARN";偏离理论分布的reads超过30%时,报"FAIL"。
统计reads的平均GC含量的分布。横轴为GC比例,纵轴为reads数量。红线是实际情况,蓝线是理论分布(正态分布,均值不一定在50%,而是由平均GC含量推断的)。曲线形状的偏差往往是由于文库的污染或是部分reads构成的子集有偏差(overrepresentedreads)。形状接近正态但偏离理论分布的情况提示我们可能有系统偏差。偏离理论分布的reads超过15%时,报"WARN";偏离理论分布的reads超过30%时,报"FAIL"。
8.Per base N content
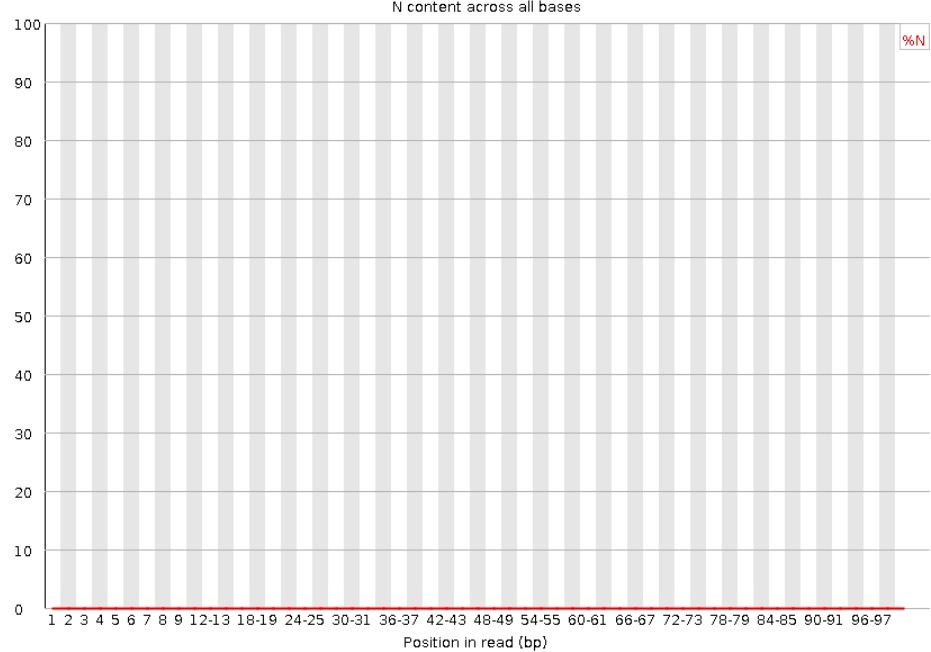 正常情况下N的比例是很小的,所以图上常常看到一条直线,但放大Y轴之后会发现还是有N的存在,这不算问题。当Y轴在0%-100%的范围内也能看到“凸起”时,说明测序系统出了问题。当任意位置的N的比例超过5%,报"WARN";当任意位置的N的比例超过20%,报"FAIL"。
正常情况下N的比例是很小的,所以图上常常看到一条直线,但放大Y轴之后会发现还是有N的存在,这不算问题。当Y轴在0%-100%的范围内也能看到“凸起”时,说明测序系统出了问题。当任意位置的N的比例超过5%,报"WARN";当任意位置的N的比例超过20%,报"FAIL"。
9.Sequence Length Distribution
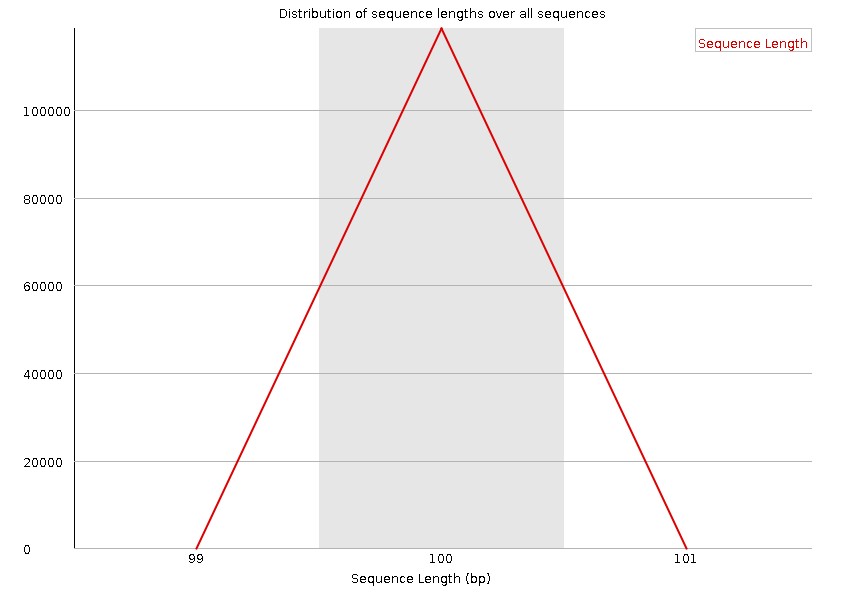 统计reads长度的分布。横轴为片段长度,纵轴为read数量。当reads长度不一致时报"WARN";当有长度为0的read时报"FAIL"。
统计reads长度的分布。横轴为片段长度,纵轴为read数量。当reads长度不一致时报"WARN";当有长度为0的read时报"FAIL"。
10.Sequence Duplication Levels
统计序列的重复度(duplication level,也即一个文库中某条序列的copy数),理论上大部分序列都只出现一次,低的重复度意味着高的基因组覆盖率。测序深度越高,越容易产生一定程度的重复,这是正常的现象;但如果duplication的程度很高,就提示我们可能有bias的存在(如建库过程中的PCR duplication)。可以想象,如果原始数据很大(事实往往如此),对所有序列的比较将会需要很大内存,所以FastQC只用前100,000条reads来进行统计,以反映全部数据中序列重复度情况。而且,大于75bp的reads只取前50bp进行比较,由于reads越长越不容易完全相同(由测序错误导致),所以这样做使得重复度的统计更加严格。序列duplication level分布图将会展示文库中不同重复度的序列所占比例,其中横坐标是duplication levels,纵坐标是duplicated reads的比例。图中蓝色线展示了全部序列中不同重复度序列的百分比,红线显示的是有重复序列中不同重复度序列的百分比(所有序列的重复度减去1)。
由于展示范围的限制,重复数目大于等于10的reads会被按照区间合并统计,造成在duplicationlevel为10的时候曲线突然凸起,结果如下所示:
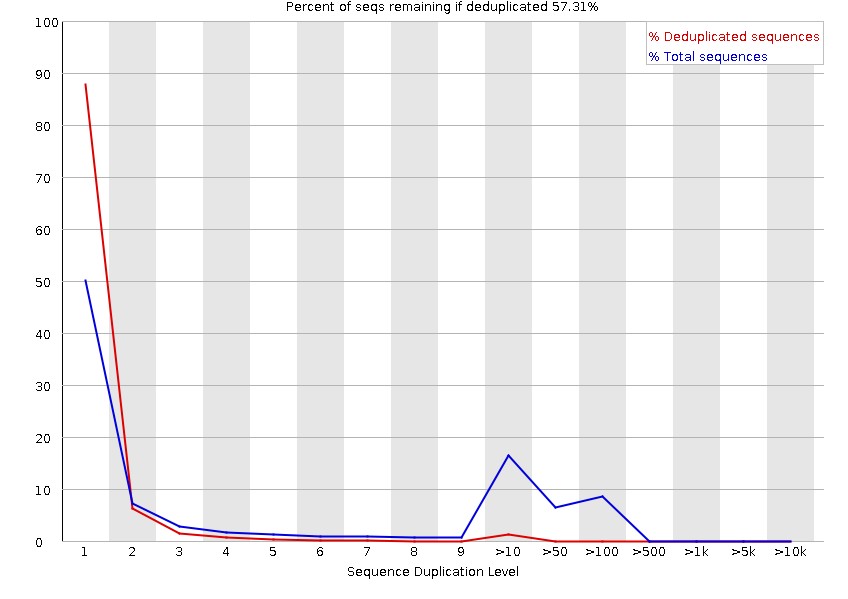
当非unique(也即duplication level大于1)的reads占总数的比例大于20%时,报"WARN";当非unique的reads占总数的比例大于50%时,报"FAIL"。
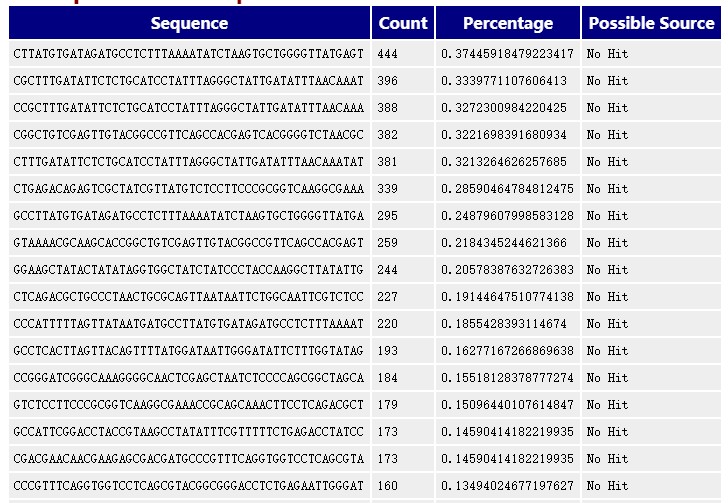
11.Overrepresented sequences
如果有某个序列大量出现,就叫做over-represented。FastQC的标准是占全部reads的0.1%以上。和上面的duplicate analysis一样,为了计算方便,只取了fastq数据的前100,000条reads进行统计,所以有可能over-represented reads不全在里面。而且大于75bp的reads也是只取50bp。统计结果以列表形式展示,当发现超过总reads数0.1%的reads时报"WARN",当发现超过总reads数1%的reads时报"FAIL"。
12.Adapter Content
统计接头序列的含量。一般测序仪自带软件会切去接头序列,所以下机数据并没有接头序列。
- 发表于 2020-04-15 15:31
- 阅读 ( 11297 )
- 分类:软件工具
你可能感兴趣的文章
- 使用fastp软件对fastq文件进行剪切 1575 浏览
- BWA比对及Samtools提取mapped reads 7333 浏览
- NCBI下载SRR并转换为fastq文件 12452 浏览
- fastq文件转换成fasta 文件perl 2269 浏览
- 给一个fastq ID列表,输出指定ID对应的双端fastq文件: 3647 浏览
相关问题
- fq.gz文件转fa文件 1 回答
- 批量srr转fastq 0 回答
- fastqc $datadir/*.gz -o $workdir/1.fastqc 报错 1 回答
- sra文件转换为fastq文件 1 回答
- fastqc运行报错是为什么?命令是:fastqc -o /root -t 2 /root/clean_reads.1.fq.gz /root/clean_reads.2.fq.gz 1 回答
- 使用bio-linux中sratoolkit里prefetch下载数据,但是无法打开试过用自带的sratoolkit,也自己去下了个最新版的放在用户文件夹也不行,我按照网上的教程设置环境变量,用source ~/.zshrc使配置生效了(好像bio-linux用bashrc不行?),但是无论如何也打不开,不知道哪里出了问题,有大佬知道吗,小白实在不懂 1 回答
