生成OTU方法如何选择,OTU/ASV?
扩增子分析视频课程推荐:https://bdtcd.xetslk.com/s/qZRiF
微生物多样性分析中最重要的就是OTU特征表,一切后续分析都围绕OTU表来进行。生成OTU除了聚类的方法,还有降噪的方法。本文分析DADA2、Deblur、Unoise3共三种降噪方法的异同点。
1DADA2
Callahan等人在2016年推出DADA的更新版DADA2,文章发表在Nature
Method上。文中对比了DADA2的方法和UPARSE聚类方法的得到OUT的效果(图1),结果表明,DADA2可以识别更多的真实序列,比UPARSE输出更少的伪序列,提高了精确度。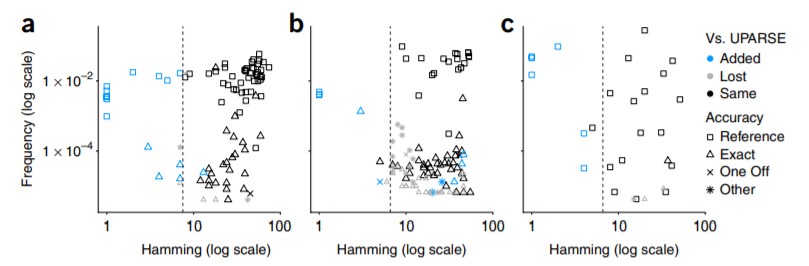
同时文中对孕期女性阴道样本进行分析,用DADA2的方法观察到了Lactobacilluscrispatus菌株水平的差异(图2),而传统的OUT聚类方法则无法观察到。
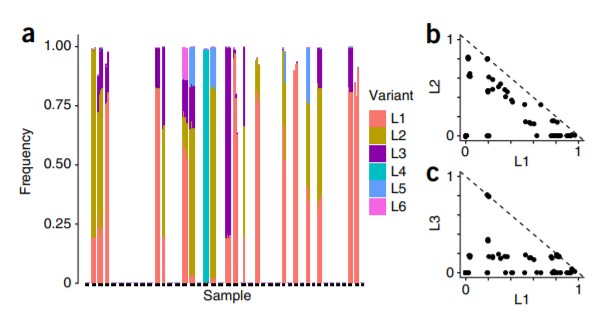
安装方法:(R包)
source("https://bioconductor.org/biocLite.R")
biocLite("dada2")
2Deblur
Amir等人在16年出的不聚类OUT分析方法,文中作者比较了Deblur与DADA2、UNOISE2的效果。结果表明,与DADA2相似,Deblur同样可以获得稳定的OTUs (图2A)。两种方法得到的序列与NCBI中的nr/nt数据库进行比对,DEblur方法的误配更少,说明DEblur比DADA2更可靠(图B)。Deblur的运算速度比UNIOSE2低一个数量级,却比DADA2 高出一个数量级(图3D)。
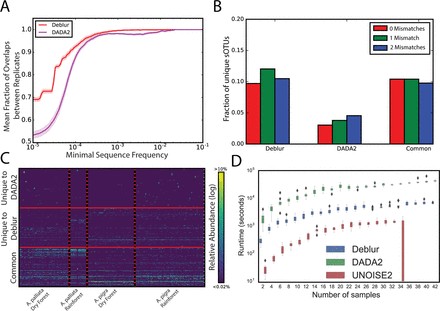
且Deblur可以基于单个样品分析,而DADA2和Unoise2只能对混合样本进行分析。
安装方法:(Github)
https://github.com/biocore/deblur
3UNIOSE3
UNIOSE3是UNIOSE2的更新版,UNIOSE2比DADA2的方法更精准,文章中对比了聚类(图4左)和去噪(图4右)两种方法得到OTU的效果。可以看到,左图中,高丰度的序列周围存在很多相近的低丰度序列,大部分是由于PCR和测序过程引入的,而右图中为去噪后的结果。可以看出,去噪的方法会保留更多真实序列,但也有可能留下嵌合体。
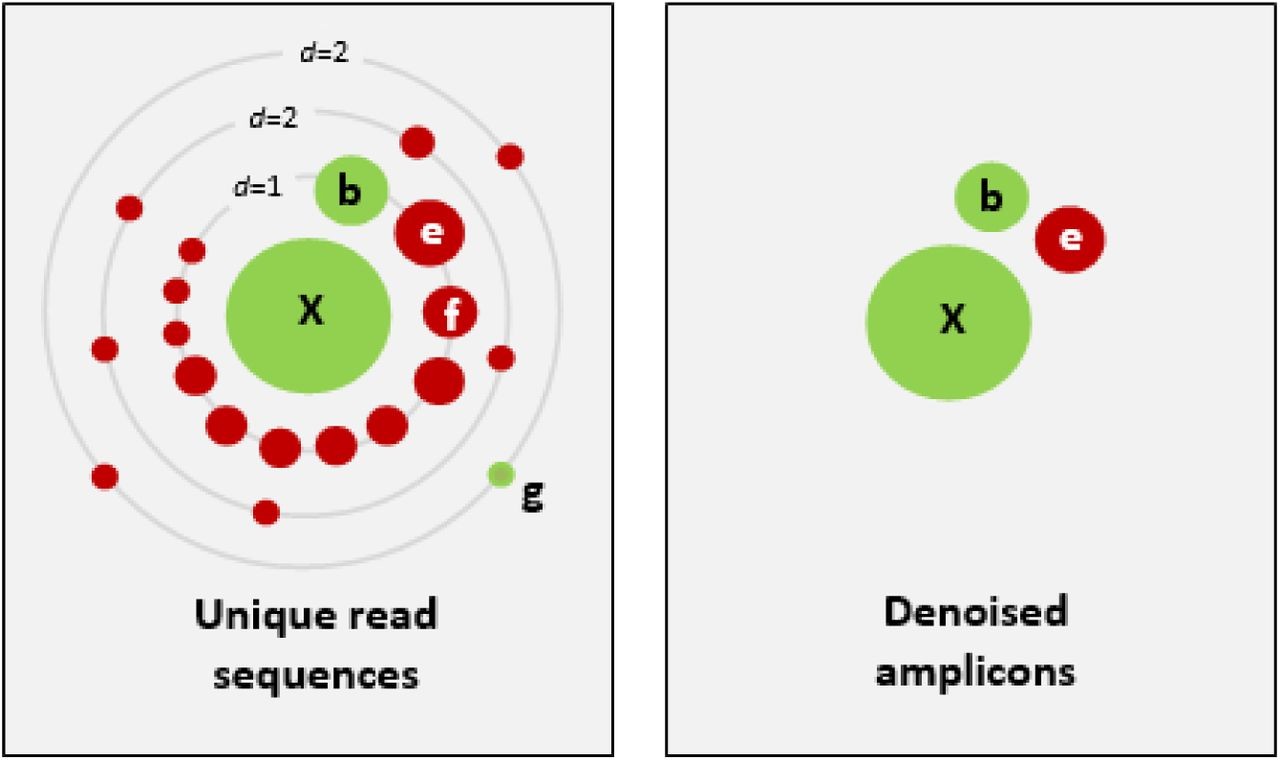
安装地址:(32位免费)
http://www.drive5.com/usearch/
总结:
DADA2和Deblur默认去掉singletons,DADA2丢弃的reads相对较少,而Deblur运行速度更快,且可以基于单样本分析;UNOISE3默认保留丰度在8以上的序列,运行速度比Deblur更快。UPARSE得到的是所有正确的生物学序列的子集,每两条序列之间的相似程度在97%以上,但会丢失一些具有意义的生物学序列;而UNOISE3得到的是所有正确的生物学序列,但由于种内差异,对结果亦会造成影响,但要检测单一环境下的菌株,则UNOISE3更有优势。对于是选择聚类还是降噪的方法, Edgar作者本人给出的建议是:最好两种方法都尝试,毕竟没有完美的算法,总会存在一些错误。如果两种方法得出的结论不同,可能两个结果都不可信,需找一下原因;如果结果相同,则更有说服力。具体选择哪种方法还需根据研究内容谨慎考虑。
参考文章:
1.Deblur Rapidly Resolves Single-Nucleotide Community Sequence Patterns
2.UNOISE2: improved error-correction for Illumina 16S and ITS amplicon sequencing
3.DADA2: High-resolution sample inference from Illumina amplicon data
此外,我们在网易云课堂上有各种教学视频,有兴趣可以了解一下:
2.OTU网络图
6. 更多学习内容:linux、perl语言画图,更多免费课程请点击以下链接:
http://m.study.163.com/provider/400000000234009/index.htm?share=1&shareId=1433306807
- 发表于 2020-09-17 14:37
- 阅读 ( 15689 )
- 分类:宏基因组
你可能感兴趣的文章
- 16S聚类还是降噪? 5870 浏览
- OTUtable标准化方法 5072 浏览
- MENA如何结合OTU共现性网络与环境因子 13395 浏览
- 做微生物研究必懂的OTU table相关知识 40092 浏览
- 你不知道的微生物互作分析方法 19449 浏览
