进化树构建的方法及原理详解
系统进化树介绍
研究分子进化所要构建的系统发生树(Phylogenetic tree),也叫分子树,对于一个未知的基因或蛋白质序列,可以利用系统发生树确定与其亲缘关系最近的物种。比如你得到了一个新发现的细菌的核糖体RNA,你可以将它跟所有已知的核糖体RNA放在一起,然后用他们构建一棵系统发生树。这样就可以从树上推测出谁和这个新细菌的关系最近。系统发生树还可以预测一个新发现的基因或蛋白质的功能。系统发生树还分为有根树和无根树(下图),顾名思义,有根树就是有根,无根树就是无根。其实两者是可以互换的。如果我们按住无根树上某一个点,然后用把梳子将树上所有的枝条都以这个点为中心向右梳理,就能把它梳成有根树的样子。按住的这个点就是根。所以对于一棵树来说,根的位置是主观的,你想让他在哪它就在哪里。但是你不能随意指定哪个内节点当根,毕竟根有其自身的生物学意义,它应该是所有叶子的共同祖先。那么我们如何确定根的位置呢?可以通过外类群(outgroup)来确定,从而把无根树变成有根树。有根树反映了树上基因或蛋白质进化的时间顺序,通过分析有根树的树枝的长度,可以了解不同的基因或蛋白质以什么方式和速率进化。而无根树只反映分类单元之间的距离,而不涉及谁是谁的祖先问题。做有根树需要指定外类群。所谓外类群,就是你所研究的内容之外的一个群。比如你要分析某一个基因在不同人种间的进化关系,那就可以额外选择黑猩猩加入进来,作为外类群一同参与建树。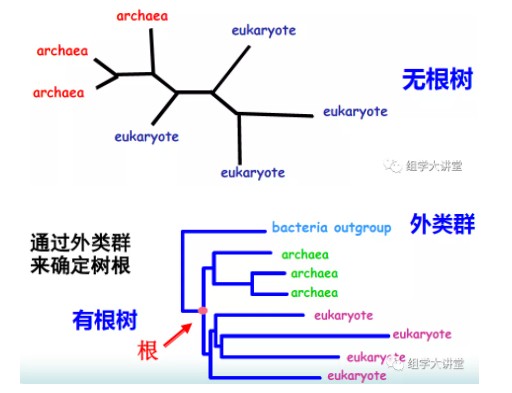
进化树的构建
(1)数据准备
在进行系统发育分析时需要通过构建系统发育树来描述不同物种或者基因之间的进化关系,通过同源DNA的核苷酸序列或者同源蛋白质分子的氨基酸序列可以实现构建进化树的构建。
(3)序列比对
为了保证序列的同源性和所得系统发育关系的可靠性,需要对原始序列进行比对和校正。自动比对序列的软件包括Clustalw 、MAFFT、MUSCLE等。
(4)保守区用于构建进化树
保守区选择是系统发育分析过程中一个重要的步骤。分析时可以选择保守位点,也可以选择基因全长序列,但是当序列差异大时,建议保留保守序列用于进化树构建。常用的保留序列保守区的软件有Gblock、MEME等。
进化树构建方法的选择
| 算法英文名 | 算法中文名 |
| ML,Maximum likelihood | 最大似然法 |
| NJ,Neighbor-Joining | 邻接法 |
| MP,Maximum parsimony | 最大简约法 |
| ME,Minimum Evolution | 最小进化法 |
| Bayesian | 贝叶斯推断 |
| UPGMA | 不常用 |
从计算速度来看,最快的是基于距离的方法,几十条序列几秒钟即可完成。其次是最大简约法。最大似然法就要慢得多。最慢的是贝叶斯法。但是从计算准确度来看,算得最慢的贝叶斯法确是最准确,而算得最快的基于距离法结果确是最粗糙。从实用的角度,建议使用最大似然法。因为这种方法无论从速度还是准确度都比较适中。
进化树构建方法讲解
虽然软件可以快速自动地完成系统发生树的构建,但是对于基本算法的了解还是必不可少的。以非加权分组平均法(UPGMA法)为例,介绍如何通过计算所有序列两两间的距离,再根据距离远近构建系统发生树。序列两两间的距离可以用双序列比对得出的一致度/相似度代表,或用其他简化值代替。
第一种:UPGMA法(非加权配对算术平均法)
比如,有如下 A、B、C、D 四条序列:
A:TAGG
B:TACG
C:AAGC
D:AGCC
在接下来的例子里,我们用序列间不同的碱基数目作为序列间遗传距离的度量。首先,计算出每两条序列间有几个碱基不同,并以用矩阵的形式记录下这些距离,找出距离最小的一对序列。A和B之间的距离最小,d[AB]=1。然后将 A 与 B 合并聚集,其分支点为 d[AB]/2=1/2=0.5。即,A、B之间的距离等于1,从中间折叠后每边各 0.5。现在,把(AB)看成一个整体,分别计算它们与C和D的距离。(AB)和C的距离等于A和 C 的距离加上B和C的距离除以2,即,d[(AB)C]=(d[AC]+d[BC])/2=(2+3)/2=2.5。同样,(AB)和D的距离等于A和D的距离加上B和D的距离除以2,即,d[(AB)D]=(d[AD]+d[BD])/2=(4+3)/2=3.5。据此,计算出新的距离矩阵,并找出新矩阵中最小的距离。C 和 D 之间的距离最小,d[CD]=2。将C和D进行合并聚集,其分支点为 d[CD]/2=2/2=1。接下来,把(CD)看成一个整体,计算它们与(AB)之间的距离。(CD)与(AB)之间的距离等于C和(AB)的距离加上D和(AB)的距离除以2,即,d[(CD)(AB)]= (d[C(AB)]+d[D(AB)])/2=(2.5+3.5)/2=3。最后,将(AB)与(CD)进行合并聚集,归为一类,分支点为 d[(CD)(AB)]/2=3/2=1.5。这样,A、B、C、D 四条序列的系统发生树就构建好了。树上,枝的长短直接反应了它们与共同祖先的距离。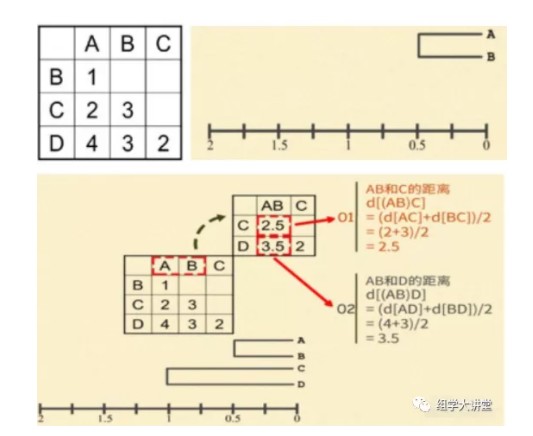
第二种:邻接法NJ法(neighbor joining method)
是一种推论叠加树的方法。在概念上与UPGMA法相同,但是有四点区别
a. NJ法不要求距离符合超度量特性,但要求数据应非常接近或符合叠加性条件,即该方法要求对距离进行校正。
b. 邻接法在成聚过程中连接的是分类单元之间的节点(node),而不是分类单元本身。
c. NJ法中原始距离数据用于估算系统树上所有端结分类单元之间的距离矩阵,校正后的距离用于确定节点之间的连接顺序。
d. 在重建系统发育树时,NJ法取消了UPGMA法所做的假定,认为在此进化分支上,发生趋异的次数可以不同。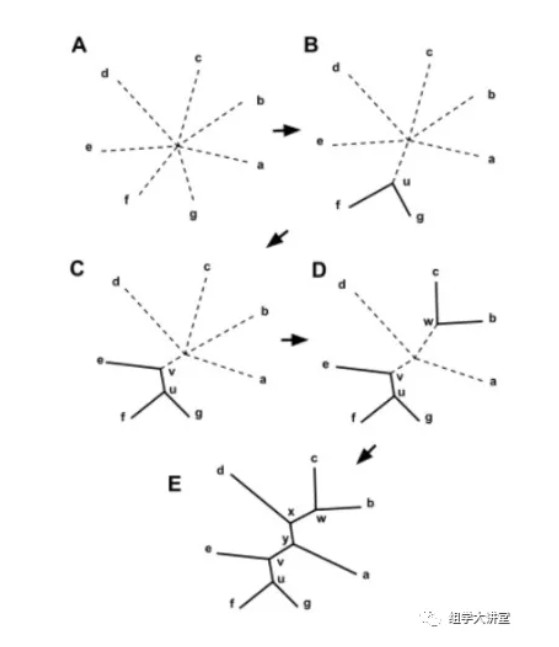
① 对于给定距离矩阵中的每一端结i,用下式计算与其它分类单元之间的净趋异量(Ri) (t:矩阵中的分类单元数)
② 建立一个速率校正距离矩阵M,其元素由下式确定:
③ 定义一个新节点u,u的三个分支分别与节点i,j和树的其余部分相连,并且Dij为矩阵中距离最小者,u到节点i和j的分支长度定义为
④ 定义u到树的其它节点k(k≠i和j外的所有节点)的距离:
⑤ 从距离矩阵中删除i和j的距离,矩阵减少一阶。
⑥ 如果矩阵仍然多于两个的节点,重复第①-⑤步,否测除最外两个节点的分支长度来确定外,树上其余节点都确定,最后是剩余的2个的分支长度Sy=Dij
第三种:最大简约法(Maximum Parsimony Method)
最大简约法的理论基础是奥卡姆(Ockham)哲学原则,这个原则认为:解释一个过程的最好理论是所需假设数目最少的那一个。方法:计算所有可能的拓扑结构,计算出所需替代数最小的那个拓扑结构,作为最优树。
第四种:最大似然法(Maximum likelihood)
这个方法最早是遗传学家以及统计学家罗纳德·费雪爵士在 1912 年至1922 年间开始使用的 。基本思想是:当从模型总体随机抽取n组样本观测值后,最合理的参数估计量应该使得从模型中抽取该n组样本观测值的概率最大,而不是像最小二乘估计法旨在得到使得模型能最好地拟合样本数据的参数估计量。方法:选取一个特定的替代模型来分析给定的一组序列数据,使得获得的每一个拓扑结构的似然率都为最大值,然后再挑出其中似然率最大的拓扑结构作为最优树(所以分析时间比较长)
进化树检验:Bootstrap
不同的方法可能会得到不同的结论,我们需要用不同的方法以及不同的参数,加上对生物问题的理解来构建最好的进化树来帮助我们更好的理解生物学问题。其中一个衡量树的好坏的方法就是看bootstrap的值,值越大越好。Bootstrap值是指根据所选的统计计算模型,设定初始值1000次,就是把序列的位点都重排,重排后的序列再用相同的办法构树,如此让模型计算并绘制1000株系统发育树,这是命令阶段产生的。如果原来树的分枝在重排后构建的树中也出现了,就给这个分枝打上1分,如果没出现就给0分,这样给进化树打分后,每个分枝就都得出分值。系统发育树中每个节点上的数字则代表在命令阶段要求的1000次进化树分析中,有多少次最后一般换算重百分数。一般bootstrap的值>70%,则认为重建的进化树较为可靠。如果bootstrap的值太低,则有可能进化树的拓扑结构有错误,进化树是不可靠的。因此,一般推荐用两种以上不同的方法构建进化树,如果所得到的进化树类似,且bootstrap值总体较高,则得到的结果较为可靠。通常情况下,只要选择了合适的方法和模型,构出的树均是有意义的,研究者可根据自己研究的需要选择最佳的树进行分析。
替换模型的选择
选择进化距离模型是构建进化树的基础。DNA 分子中基因的进化距离是通过对核苷酸替代数进行估计获得的,要估计核苷酸替代数,就必须应用核苷酸替代的数学模型。举个例子:在DNA中,碱基之间存在四种转换(A→G,G→A,C→T,T→C)和颠换(A→C,A→T,C→G,G→T),通常情况下转换频率比颠换频率高。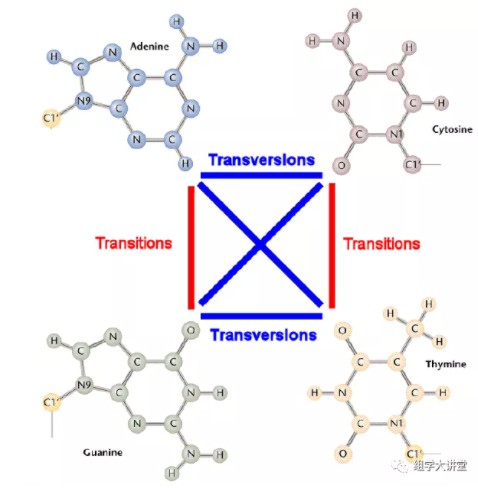
这些偏向会影响两个序列之间的预计分歧。由于DNA 序列的进化、演化比较复杂,因此许多学者提出了不同的核苷酸替代模型。当今常用的核苷酸替代模型有JC69 、K80 、F81、TN93,HKY和GTR 模型等。当然氨基酸也有很多替换模型。这些不同的替换模型确定了不同的进化距离和不同的系统发育树。但实际的生物进化历史是唯一的,我们并不能从这么多的模型中确定真实的核苷酸替代过程是依照哪种模型发生的。理论上应该尝试各种模型,根据检验结果选择最合适的模型进行计算。在系统发育分析中,最大似然法(ML)和贝叶斯法(BI)是对替换模型非常敏感的两种算法,因此利用ML法或BI法重建系统发育树前,核苷酸替换模型的选择是必不可少的过程;最大简约法(Maximum parsimony)不依赖任何进化模型,因此通过最大简约法构建系统发育树时,不需要此步骤。一般来讲,如果模型合适,ML的效果较好。对近缘序列,有人喜欢MP,因为用的假设最少。MP一般不用在远缘序列上,这时一般用NJ或ML。对相似度很低的序列,NJ往往出现Long-branch attraction(LBA,长枝吸引现象),有时严重干扰进化树的构建。贝叶斯的方法则太慢。对于各种方法构建分子进化树的准确性,一篇综述(Hall BG. Mol Biol Evol 2005, 22(3):792-802)认为贝叶斯的方法最好,其次是ML,然后是MP。其实如果序列的相似性较高,各种方法都会得到不错的结果,模型间的差别也不大。
小结
比较以上几种主要的构树方法,一般情况下,若有合适的分子进化模型可供选择,用最大似然法构树获得的结果较好;对于近缘物种序列,通常情况下使用最大简约法;而对于远缘物种序列,一般使用邻接法或最大似然法。对于相似度很低的序列,邻接法往往出现长枝吸引(branch attraction)现象,有时严重干扰进化树的构建。对于各种方法重建进化树的准确性,Hall (2005)认为贝叶斯法最好,其次是最大似然法,然后是最大简约法。其实如果序列的相似性较高,各种方法都会得到不错的结果,模型间的差别也不大。邻接法和最大似然法是需要选择模型的。蛋白质序列和DNA序列的模型选择是不同的。蛋白质序列的构树模型一般选择Poissoncorrection(泊松修正),而核酸序列的构树模型一般选择Kimura2-parameter (Kimura一2参数)。如果对各种模型的理解并不深入,最好不要使用其他复杂的模型。参数的设置推荐使用缺省的参数。
参考:
Hall BG. Comparison of the accuracies of several phylogenetic methods using protein and DNA sequences. Mol Biol Evol. 2005 Mar;22(3):792-802. doi: 10.1093/molbev/msi066. Epub 2004 Dec 8. Erratum in: Mol Biol Evol. 2005 Apr;22(4):1160. PMID: 15590907.模型计算公式:https://cran.r-project.org/web/packages/jackalope/vignettes/sub-models.html
更多生物信息课程:https://study.omicsclass.com/index
- 发表于 2020-09-29 16:52
- 阅读 ( 48386 )
- 分类:遗传进化
你可能感兴趣的文章
- 给进化树标定化石时间 4199 浏览
- 快速生成iTOL配置文件美化你的进化树 3457 浏览
- 最全进化树构建知识点大盘点 17164 浏览
- 群体遗传进化进化树分析方法: 4566 浏览
- 如何用R语言绘制进化树+热图 11562 浏览
- 重测序群体遗传进化分析之—进化树构建 18149 浏览
相关问题
- fasttree 建树时没有找到这个软件 2 回答
- 群体遗传分析过程中无法构建进化树 2 回答
- iqtree2 iqtree2 -s supergene.phy -st DNA -m TEST -B 1000 -bnni -alrt 1000 --prefix iqtree出现报错 1 回答
- 已知一个基因家族有多个亚家族,在做进化树时,如何在进化树中将各个亚族区分开? 0 回答
- 经旋转调整后evolview进化树依然出现分支节点跟mega11不匹配的问题 1 回答
- 请问,用Evolview为树枝染色时,如果一个添加多个,只有第一个被染色,除非换行。怎么像教程里使用“mouse,human blue”一次染色多个不挨着的树枝呢 1 回答
