连锁不平衡原理与计算(LD)
连锁不平衡 (linkage disequilibrium, LD):指在某一群体中,不同座位上某两个基因同时遗传的频率明显高于预期的随机频率的现象。下面通过一个例子来说明。
基因A的两个allele 分别用A和a 表示,基因B的allele分别用B和b表示,如果这两个基因完全独立遗传,也就是说其allele 完全随机组合,那么后代中会出现4种单倍型,AB, Ab, aB, ab, 而且出现的概率都是相同的,都是0.25;如果这两个基因在遗传时不是独立的,意味着后代中单倍型出现的概率不是完全随机的了,我们就可以说两个基因是存在连锁关系的,基因在遗传时出现连锁的现象就叫做连锁不平衡。
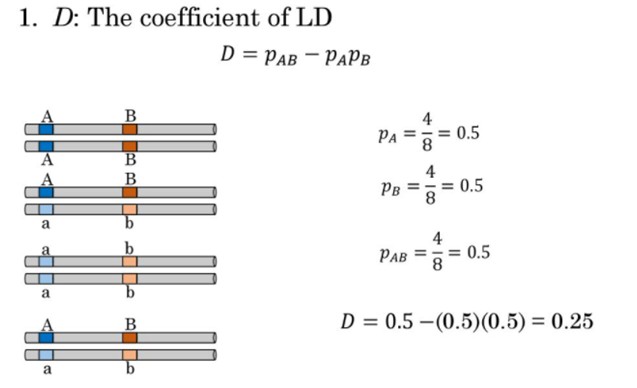
从上面的例子可以看出,在连锁不平衡中,单倍型出现的概率与随机组合的概率之间存在了偏移。这个偏移的程度就决定了连锁不平衡的程度。接着上面的例子,独立遗传时,单倍型AB出现的概率为PA*PB, 这个概率我们暂且称之为理论概率;当出现了连锁不平衡时,单倍型AB出现的概率用PAB表示,我们暂且称之为实际观测到的概率,这两个概率之间的差,就反应了连锁不平衡的程度。计算公式:D=PAB-PA*PB (PAB 观测频率,PA*PB 理论频率 )
D值不等于0,就可以说两个基因之间是连锁不平衡的,D绝对值大小直接反应了两个基因之间的连锁程度的大小,绝对值越大,连锁程度越大。但是D值无法比较不同基因之间连锁程度的大小, 因为它是根据每个基因allel的频率计算出来的。

为了能够比较基因连锁程度的大小,提出了D'的概念,D'值可以看做是归一化之后的D值,归一化之后的值可以用于比较不同基因连锁程度的大小。D’的取值范围为0到1,D’ = 0 表示完全连锁平衡,独立遗传;D’ = 1 表示完全连锁不平衡。

除了D’ 值之外,还有一个衡量连锁不平衡程度的标准,就是r2值。r2等于0时,表示完全连锁平衡,独立遗传;r2等于1时, 表示完全连锁不平衡。
- 发表于 2020-11-12 14:25
- 阅读 ( 12279 )
- 分类:遗传进化
