SCI期刊选择困难症有治了!推荐一款选刊神器!速领!
发文章时你有没有期刊选择困难症?有没有到处询问自己的论文适合发哪些期刊?其实投稿也有方法,走对路事半功倍!今天就给大家推荐一款选刊神器,帮助您轻松选择对口期刊!

搞科研离不开发论文,发论文难的是选择期刊,大部分人选期刊其实就是靠经验相传,不过熟悉的期刊也就那几个,极大地限制了投稿的成功率。
今天我给大家推荐一款客观、快速的选刊神器—Find journals,为您投稿保驾护航。
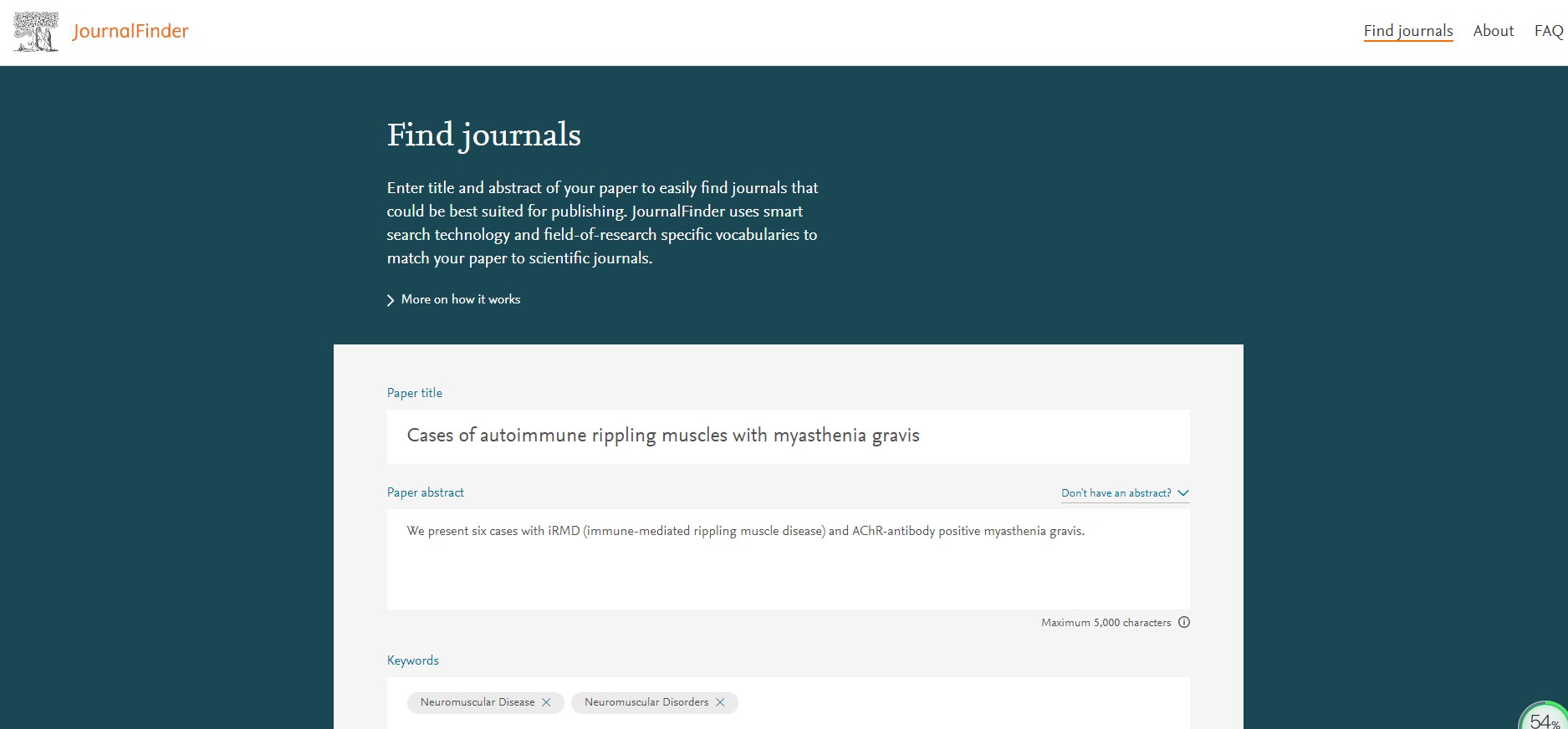
Find journals的使用
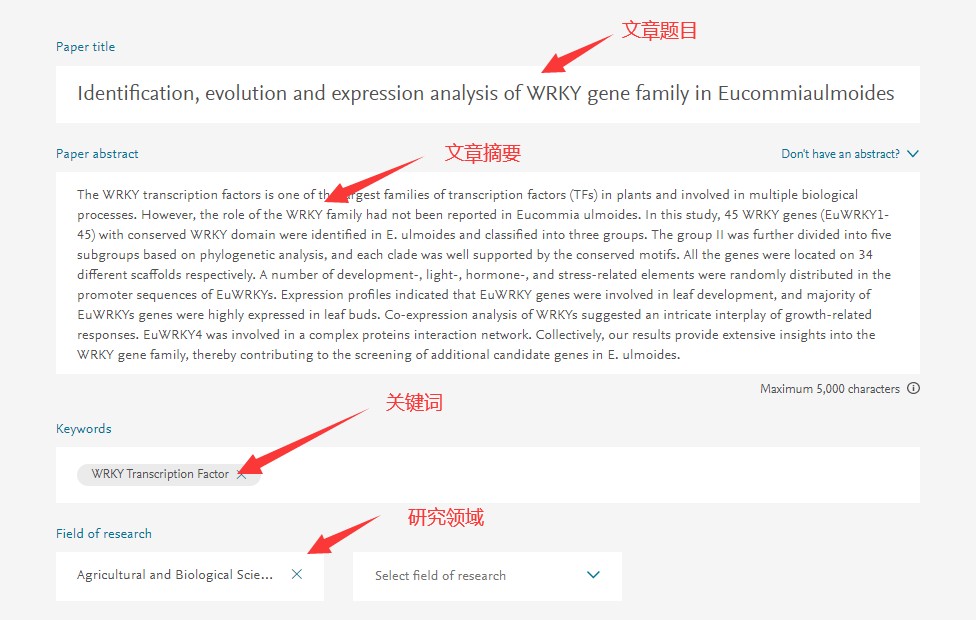
Find journals 是一个在线网站,使用十分简单,只需提交自己的论文题目、摘要、关键词,然后选择研究领域基本本可以了;假如有更多需求,如期刊性质(OA或者订阅)、影响因子、审稿周期等可以进一步进行设置,具体设置见下图。
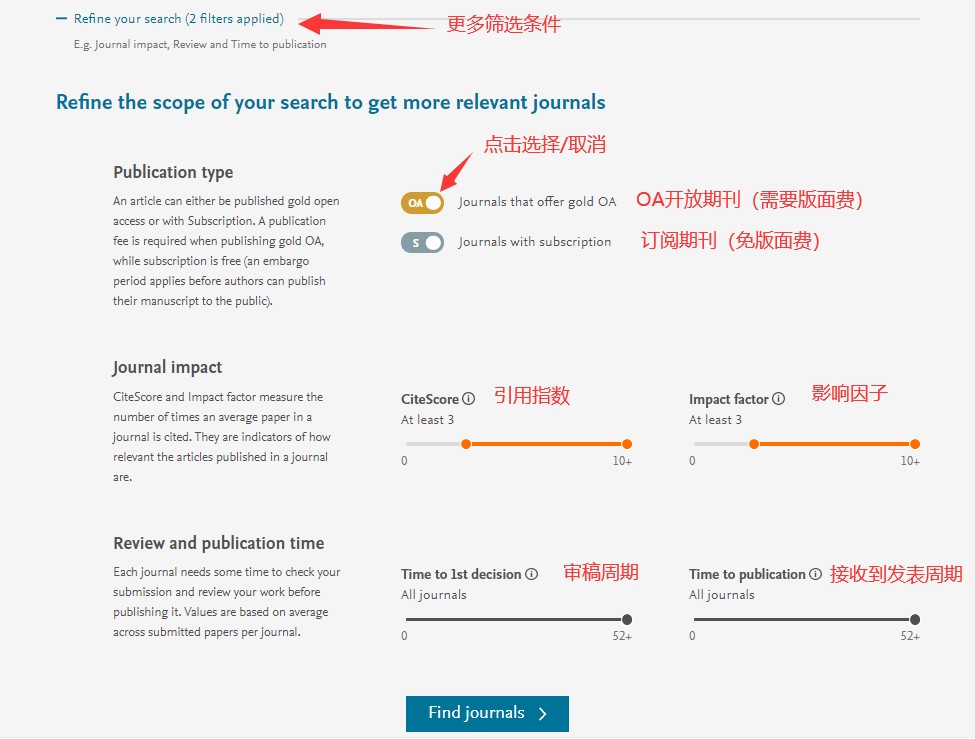 设置好以上参数后,点击下方Find journals开始筛选期刊,结果如下图,Edit your search下还可以继续修改筛选参数;再下方是筛选出的期刊列表,信息也很直观。
设置好以上参数后,点击下方Find journals开始筛选期刊,结果如下图,Edit your search下还可以继续修改筛选参数;再下方是筛选出的期刊列表,信息也很直观。
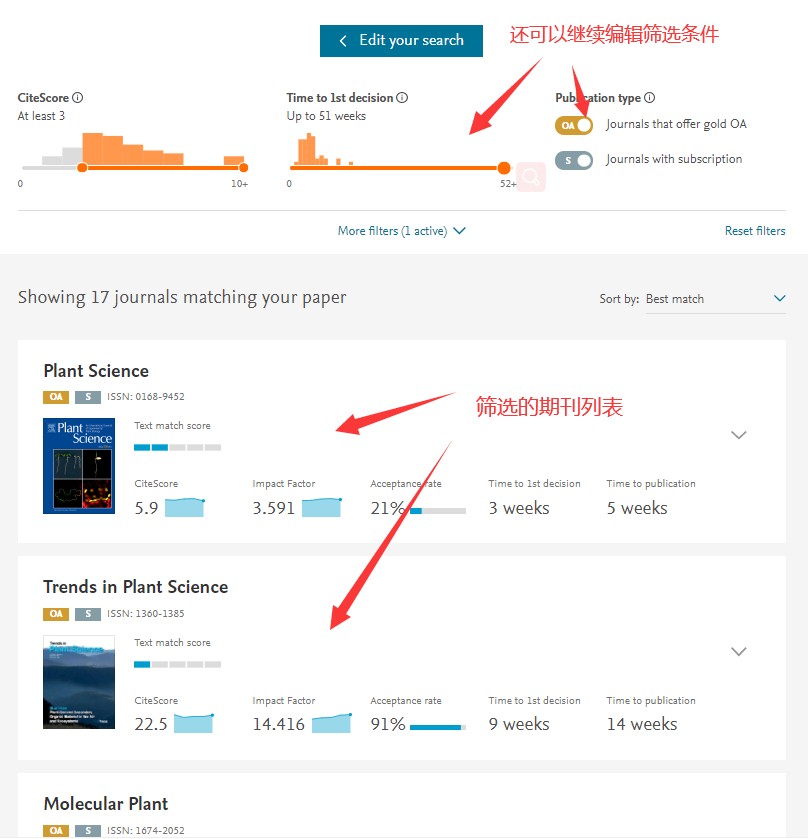 点击具体期刊可以进一步查看该期刊信息,如下图,展示信息很是丰富,像引用指数、影响因子、接收比例、审稿/出版周期、版面费等,另外在页面右上有投稿入口链接,点击可直达投稿页面。
点击具体期刊可以进一步查看该期刊信息,如下图,展示信息很是丰富,像引用指数、影响因子、接收比例、审稿/出版周期、版面费等,另外在页面右上有投稿入口链接,点击可直达投稿页面。

好了,Find journals先介绍到这里,最后网址奉上:
https://journalfinder.elsevier.com
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,又能发文章,又能学生信技能,一举两得的SCI好思路;基因家族分析课程已更新分析内容,最新版课程学习链接:基因家族分析实操课程
2. 转录组数据结果理解不深入?图表看不懂?这就是你转录组数据不会挖掘、文章不会撰写的原因,让我带你一起深入了解转录组数据结果,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?多学点数据处理技能:学习链接:转录组标准分析后的数据挖掘
5.微生物多样性分析很简单,但是分析内容项目并不少,理解起来有困难?看我深入浅出讲给你听,学习链接: 微生物16S/ITS/18S分析原理及结果解读
6. 学生物的必学生信技能:Docker安装与使用、linux系统入门、Perl语言入门到精通、Python生信入门到精通、perl语言高级编程
7. 生信绘图、科研绘图技能:Cytoscape与网络图绘制、微生物OTU网络图绘制(Cytoscape)、R语言基础与绘图、R语言绘图基础(ggplot2)
8.生物信息实战技能,0基础也可以学会,内含脚本及demo数据:RNAseq有参转录组自主分析、基因组重测序自主分析、微生物多样性自主分析
9. 更多学习内容:linux、perl、R语言画图,更多免费课程请扫描下方二维码进入组学大讲堂网校学习:
- 发表于 2021-05-27 11:06
- 阅读 ( 2800 )
- 分类:软件工具

