heatmap_group_plot.r 分组热图展示免疫侵润结果
heatmap_group_plot.r 分组热图展示免疫侵润结果
使用参数说明:
$ Rscript heatmap_group_plot.r -h
usage: heatmap_group_plot.r [-h] -i filepath -m
filepath -g group
[group ...]
[-l filepath]
[-t topnumber]
[--scale SCALE]
[--log2]
[--cluster_rows]
[--cluster_cols]
[--show_rownames]
[--show_colnames]
[-c color [color ...]]
[-o outdir]
[-p prefix]
[-H height]
[-W width]
plot heatmap
optional arguments:
-h, --help show this help message and exit
-i filepath, --input filepath
input the immune res data,[required]
-m filepath, --metadata filepath
input metadata file path[required]
-g group [group ...], --group group [group ...]
input group id in metadata file, can set multi
groups[required]
-l filepath, --genelist filepath
gene list file to keep [default NULL]
-t topnumber, --top topnumber
The top var to plot [default NULL]
--scale SCALE character indicating if the values should be centered
and scaled in either the row direction or the column
direction, or none. Corresponding values are "row",
"column" and "none" [default="row"]
--log2 whether do log2 transfrom [optional, default: False]
--cluster_rows whether cluster_rows [optional, default: False]
--cluster_cols whether cluster_cols [optional, default: False]
--show_rownames whether show_rownames [optional, default: False]
--show_colnames whether show_colnames [optional, default: False]
-c color [color ...], --color color [color ...]
color map of heatmap, give three color
[default=c('blue', 'white', 'red')
-o outdir, --outdir outdir
output file directory [default cwd]
-p prefix, --prefix prefix
out file name prefix [default demo]
-H height, --height height
the height of pic inches [default 9]
-W width, --width width
the width of pic inches [default 10]
参数说明:
结果展示:
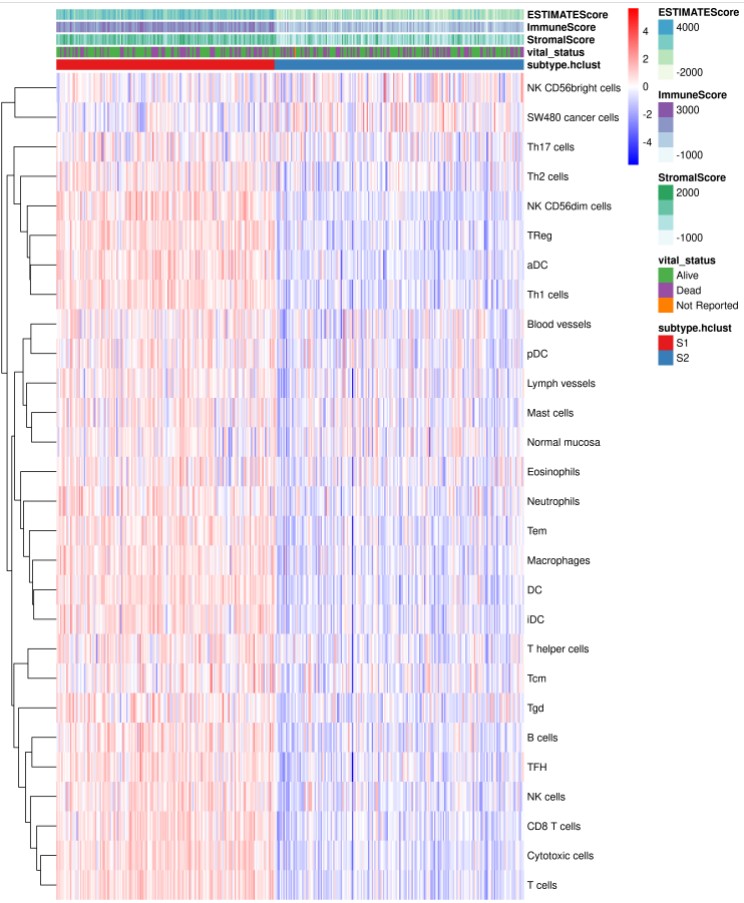
注意事项:
样本分组 metadata文件中,不要有NA值;不然会报错:
Error in names(ann_colors[[opt$group[i]]]) <- unique(mygroup) :
'names' attribute [12] must be the same length as the vector [11]
- 发表于 2021-06-22 09:54
- 阅读 ( 2373 )
- 分类:TCGA
你可能感兴趣的文章
- pheatmap绘制热图详解 28971 浏览
- 在线绘制聚类热图(heatmapper) 15299 浏览
0 条评论
请先 登录 后评论
