最新2区转录组文章!看人家是怎么写的!
2021年转录组有多便宜,转录组文章就有多难发,一般人对期刊已经放弃要求;优秀的人却仍可以游走在2区行列,人家是怎么做到的呢?我们一起去看看!
文章简介

该文章是6月21日发表在BMC Genomics(IF=3.594,中科院分区:2区)的一篇研究拟南芥春化作用和冷驯化关系的转录组文章,下面我们一起看一下这篇文章都有哪些内容。
1. 拟南芥春化动态转录组结果
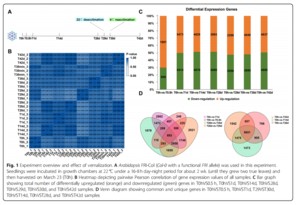
作者以拟南芥FRI-Col (Col-0 突变,拟南芥花期延后)设置了8个时间点,如下图A,该部分作者展示了转录组数据的基本情况,均是比较套路的写法,如样本相关性,差异基因数,不同组合间韦恩图,全部基因时序表达热图及不同Go term注释基因数等。
2. 冷驯化和春化的关系
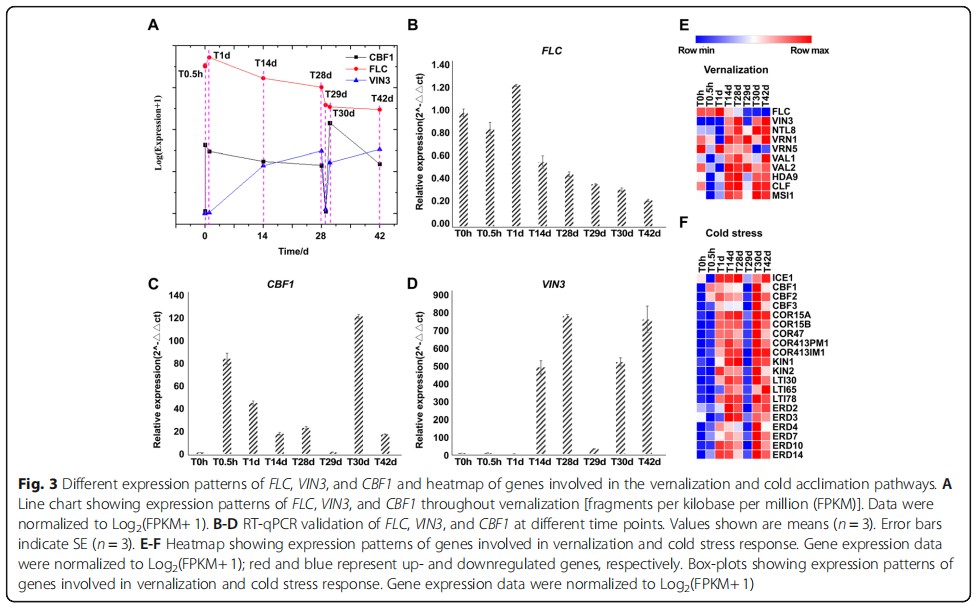
接下来作者重点研究了与冷驯化和春化相关通路的基因,如FLC, VIN3,CBF1和HOS1等,并且Qpcr验证了相关基因的表达模式,从冷驯化和春化两个通路关键基因响应时间先后分析,两个生物学过程是相对独立的。
3. 冷驯化和春化反应的加权基因共表达网络分析(WGCNA)
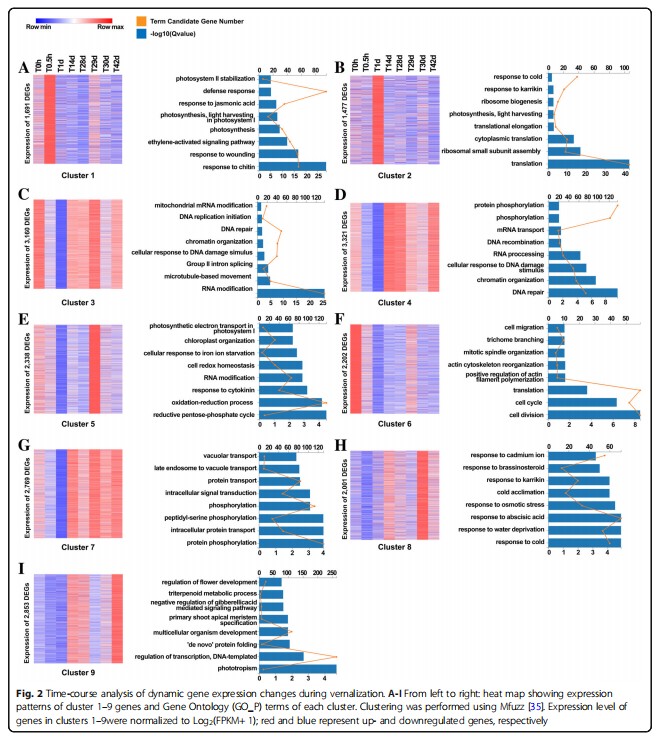
作者进一步利用加权基因共表达网络分析(WGCNA)分析冷驯化和春化之间的关系,如下图,分析表明冷驯化涉及3个阶段:一是即时冷反应,与乙烯和茉莉酸(JA)及C2H2锌指信号有关;二是对冷处理28-30d温度变化的快速、持久的响应,与生长素途径有关;三是对长期冷驯化的响应,与泛素化途径有关。

4. 春化反应中的可变剪接事件
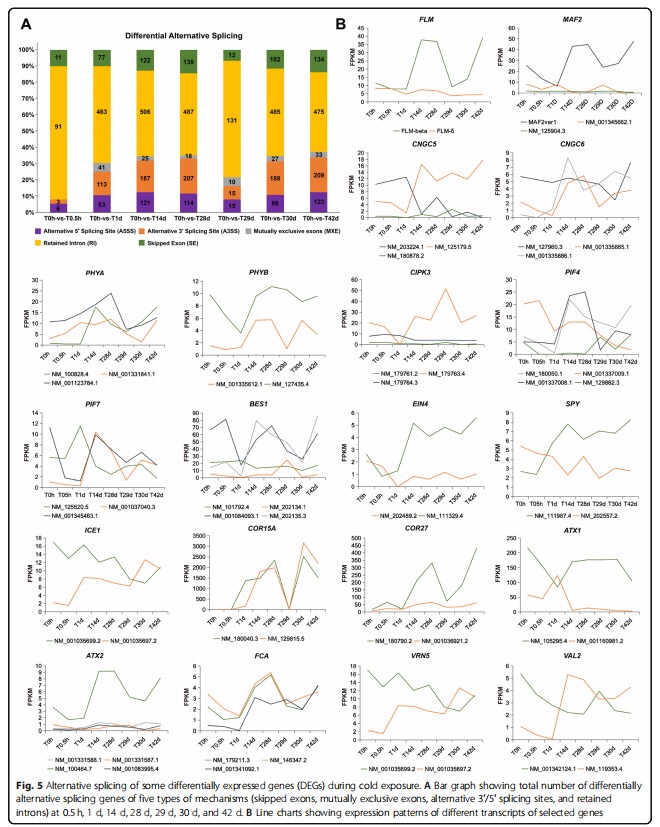
可变剪接与温度响应有关,作者又分析了冷驯化期间可变剪接事件变化情况,如下图,冷暴露期间内含子保留(RIs)的比例下降,外显子互斥(MXEs)1d后出现,3′端可变剪接(A3SS)的比例也显著增加,表明植物通过改变可变剪接事件应对冷驯化。
总结
BMC Genomics是2区期刊,2015年之前,转录组发表在该期刊是比较容易的,甚至做一个样品的无参组装就可以发,不过这已经成为老黄历了。
目前看,没有像样的实验数据加以支持,仅靠转录组数据想发表在这个期刊是很难的!不过,该篇拟南芥春化和冷驯化的文章没有实验数据为啥能发呢?
我觉得有以下几点:
1. 材料特殊:作者拟南芥FRI-Col (Col-0 突变,拟南芥花期延后),一般拟南芥发芽后4周左右开花,难以模拟40天左右的春化作用,FRI-Col突变株是研究拟南芥春化作用的好材料。
2. 设置取样时间点较多,贯穿实验全程,可更好观察春化过程中基因表达的动态变化;另外,较多时间点非常适合WGCNA分析,这也是本文亮点之一。
3. 关注性状相关通路及基因梳理较为清晰,并根据转录组数据提出春化和冷驯化相关的信号通路是相互独立的。
4. 关注可变剪接事件与冷驯化的关系,可变剪接数据一般人很少分析,更是研究热点,值得借鉴。
综上,这是一篇非常不错的值得借鉴的文章,近期有转录组文章需求的老师可以下载原文查看更多详细信息。
文章链接:https://www.ncbi.nlm.nih.gov/pmc/articles/PMC8218483/
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,又能发文章,又能学生信技能,一举两得的SCI好思路;基因家族分析课程已更新分析内容,最新版课程学习链接:基因家族分析实操课程
2. 转录组数据结果理解不深入?图表看不懂?这就是你转录组数据不会挖掘、文章不会撰写的原因,让我带你一起深入了解转录组数据结果,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?多学点数据处理技能:学习链接:转录组标准分析后的数据挖掘
5.微生物多样性分析很简单,但是分析内容项目并不少,理解起来有困难?看我深入浅出讲给你听,学习链接: 微生物16S/ITS/18S分析原理及结果解读
6. 学生物的必学生信技能:Docker安装与使用、linux系统入门、Perl语言入门到精通、Python生信入门到精通、perl语言高级编程
7. 生信绘图、科研绘图技能:Cytoscape与网络图绘制、微生物OTU网络图绘制(Cytoscape)、R语言基础与绘图、R语言绘图基础(ggplot2)
8.生物信息实战技能,0基础也可以学会,内含脚本及demo数据:RNAseq有参转录组自主分析、基因组重测序自主分析、微生物多样性自主分析
9. 更多学习内容:linux、perl、R语言画图,更多免费课程请扫描下方二维码进入组学大讲堂网校学习:
- 发表于 2021-06-29 09:56
- 阅读 ( 5313 )
- 分类:文献解读

