blast+命令选择
blast+命令选择
blast+包含有五种BLAST,如下图所示:
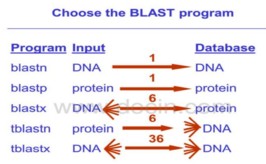
每条命令作用如下:
blastp:使用blastp来将一个蛋白质查询序列与一个蛋白质数据库进行比较。
blastn:使用blastn来将一个DNA查询序列的两条链与一个DNA数据库进行比较。
blastx:使用blastx来将一个DNA序列用所有可能的阅读框翻译成6个蛋白质序列,然后将它们逐一与蛋白质数据库进行比较。
tblastn:使用tblastn来将一个DNA数据库中的每一条序列翻译成6种可能的蛋白质,然后将你要查询的蛋白质序列与翻译的蛋白质逐一进行比较。
tblastx:使用tblastx将查询DNA以及数据库中的DNA都翻译成6种可能的蛋白质,然后进行36次蛋白质-蛋白质数据库搜索。
需要根据自己需求选择合适的命令。
此外,我们在网易云课堂上有各种教学视频,有兴趣可以了解一下:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘
6. 更多学习内容:linux、perl、R语言画图,更多免费课程请点击以下链接:
- 发表于 2021-09-10 13:37
- 阅读 ( 2438 )
- 分类:软件工具
你可能感兴趣的文章
- fastq序列做NR库比对 2822 浏览
- blast批量注释输出结果指定需要的列信息 2755 浏览
- blast报错 12570 浏览
- BLAST+使用方法 7070 浏览
0 条评论
请先 登录 后评论
