进化树+基因表达热图二合一靓图绘制教程
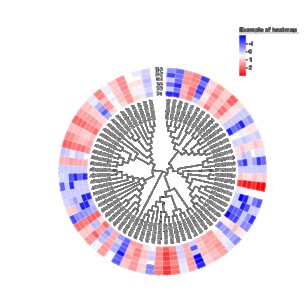
在阅读文章的时候,我们经常会看到一棵漂亮的进化树,如下图,拟南芥WRKY基因家族的进化树,最外层是基因表达量热图,中层是基因ID,里层为拟南芥基因之间的进化关系,不仅直观地展现了物种的进化发育系统,还把每个基因的表达量信息也展现出来,是不是很漂亮!今天我就教大家绘制上面这个图,让你的图成为进化树中最靓的仔!
1. 数据准备
(1) 拟南芥基因的氨基酸序列
 (2) 拟南芥基因表达量数据(标准化后)
(2) 拟南芥基因表达量数据(标准化后)

2. 序列比对
构建系统进化树的软件有很多种,但最常用的是MEGA,在官网下载MEGA软件进行构建。
(1)进入MEGA主界面,点击“ALIGN”下面的“Edit/Build Alignment”创建新的比对,弹出页面中选择“Create a new alignment”,之后再选择建立蛋白序列进入比对页面。
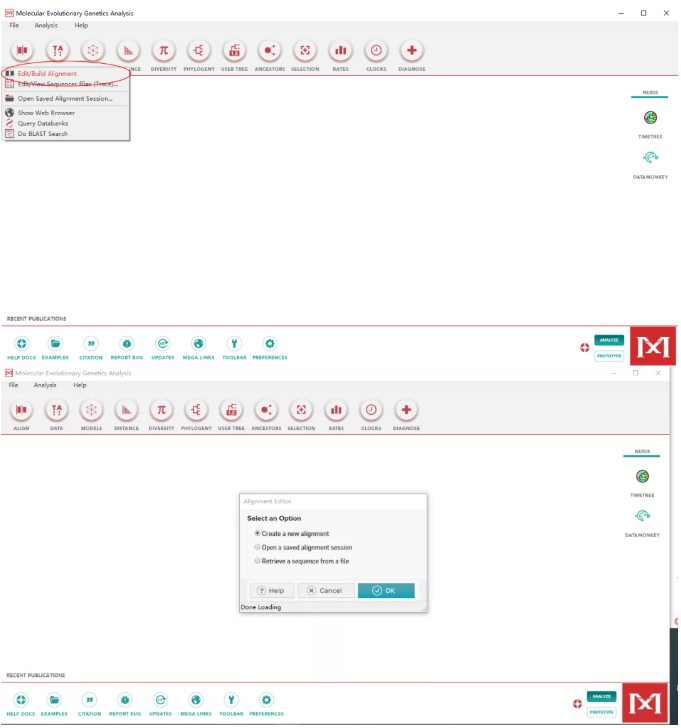 (2)在比对页面,点击Edit下面的“insert sequence from file”,选择拟南芥基因氨基酸序列。
(2)在比对页面,点击Edit下面的“insert sequence from file”,选择拟南芥基因氨基酸序列。
 (3)选择Alignmen下面的“Align by ClustalW”比对方式,不需要更改参数,直接点击“OK”。比对结束之后,按照自己需要进行碱基列的删减。比对结果可保存为多种格式mas、meg等。
(3)选择Alignmen下面的“Align by ClustalW”比对方式,不需要更改参数,直接点击“OK”。比对结束之后,按照自己需要进行碱基列的删减。比对结果可保存为多种格式mas、meg等。

3. 构建进化树
(1)回到MEGA主页面,选择PHYLOGENY下“Neighbor-Joining”进行构建。导入meg格式的比对结果文件,在弹出页面中将参数“Bootstrap method"更改为500或1000。
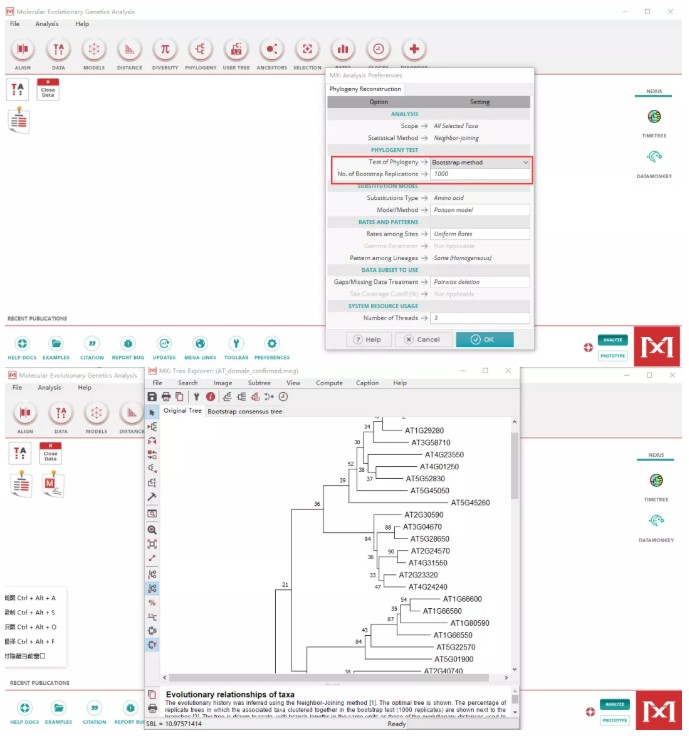 (2)将进化树根据自己需要进行简单修改,选择左上角File的“Export Current Tree(Newick)”进行保存。
(2)将进化树根据自己需要进行简单修改,选择左上角File的“Export Current Tree(Newick)”进行保存。4. 进化树美化
进化树美化最常用Evolview网站,官方网址:https://evolgenius.info//evolview-v2/#login ,不支持IE浏览器和360浏览器,推荐Google。建议使用Evolview之前注册账号,可以保留使用历史记录。
(1)选择Basic下的“upload tree file”提交基因家族进化树文件.nwk。并从线性排列改为圆形排列。
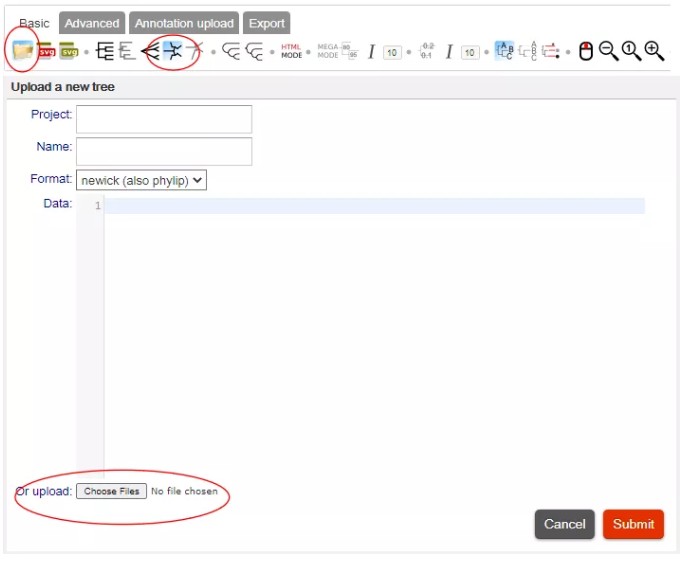
(2)点击“Annotation upload”下的第11个选项绘制热图,点击“upload data for heatmap(help)”可进入绘制热图说明文档,参数设置可以参考左上角heatmaps案例,在弹出页面上提交拟南芥基因表达量,其它得到最终进化树
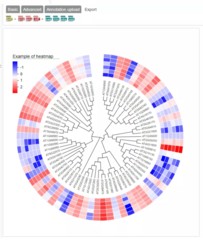
(3)在Export中导出结果,常用pdf和png格式。导出pdf文件可以根据自己需要进行AI修改,添加热图样品信息等。
- 发表于 2021-11-16 09:52
- 阅读 ( 7449 )
- 分类:科研作图
