GWAS分析哪个模型最优
GWAS关联分析课程推荐:https://bdtcd.xetslk.com/s/2KgXQq
前面我们介绍了GWAS分析中很多不同的模型GLM,MLM, CMLM,ECMLM, MLMM,SUPER,FarmCPU等详情见:《GWAS分析模型介绍》。那么我们在做GWAS分析的时候选择哪种模型分析更好呢?下面介绍一篇文献在大豆和玉米中测试了这些模型,并给出了这些模型优劣。

假阳性与假阴性控制比较
介绍模拟数据之前先介绍几个概念:
遗传力(Heritability H) 又称遗传率,指遗传方差在总方差(表型方差)中所占的比值。遗传力表明某一性状受到遗传控制的程度。它介于0到1之间,当遗传力为1时,表型变异完全由遗传因素决定,当遗传力为0时表型变异由环境因素决定。
数量性状位点(Quantitative trait locus, QTL/Q)就是一个性状由多个基因决定,每个基因对此性状都是微效的。
数量性状可以由稀有的大效应基因控制,也可能是由许多常见的微效基因控制,这两类基因用GWAS关联难度存在差异,因此作者模拟了不同遗传力(H)和不同QTL(Q )数量性状数据测试模型关联的效果:H分别模拟20%,60%,80% ;Q分别模拟 20个和40个。
8个模型分别对3个前人报道过的性状(A-B)和6个模拟性状(D-I)进行关联分析并绘制QQ图。CW trait (H = 80% 下图A) 关联结果看, ANOVA, GLM, and SUPER假阳性高;MLM, CMLM, and ECMLM这几个模型只是尾部少量SNP翘起说明假阳性减少但可能存在假阴性,因为尾部翘起的点较少。而FarmCPU 尾部高高翘起而且多说明这个模型假阳性和假阴性都控制的很好。随着遗传率H的降低(图A>B>C)只有FarmCPU 还能很好的控制假阳性和假阴性。再看模拟性状的结果(D-I)也有类似的结果。
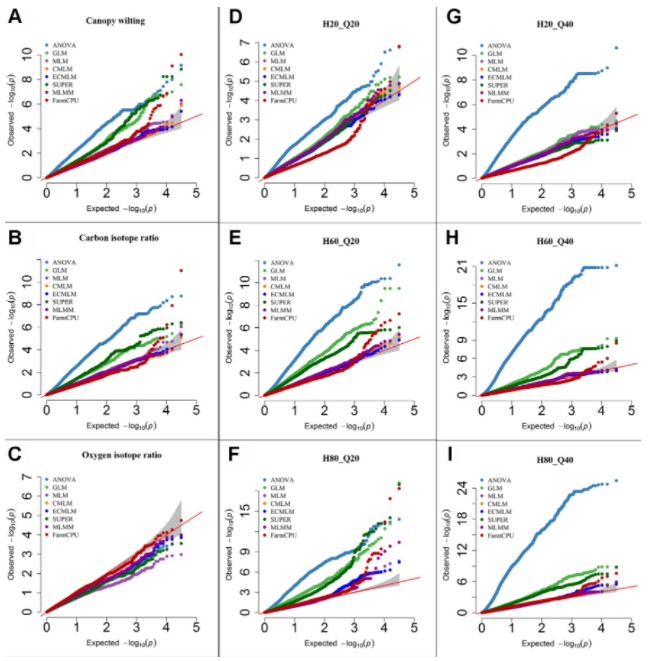
关联准确性
大豆中的花色性状受W1基因控制,这个基因的野生型为紫色花,突变型为白色花,位于大豆基因组13号染色体4552540-4557331bp处。从下图的关联结果中发现:FarmCPU, GLM, and ANOVA这结果模型关联到了W1基因所在位置附近的SNP。除了MLMLM模型其他模型关联到了,其他的一些模型关联到很多离这个基因较远距离的SNP;但是,只有FarmCPU模型关联了一个SNP正好位于W1基因上 4,559,799 bp。
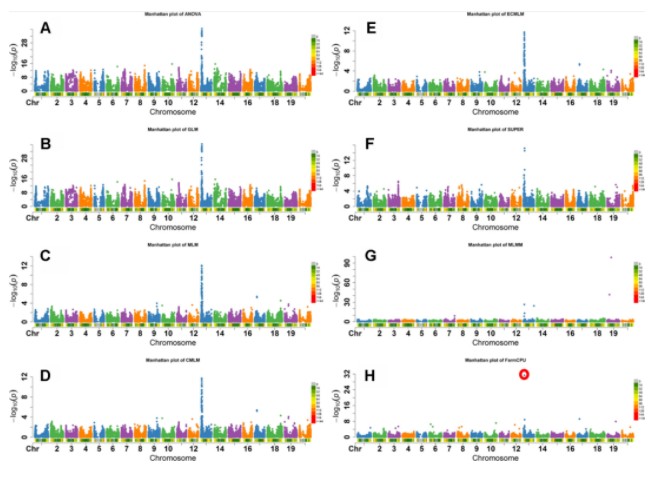
注:Manhattan plots of -Log10 (P) vs. chromosomal position of SNP markers associated with flower color in soybean from eight models including Analysis of Variance (ANOVA) (A), General Linear Model (GLM) (B), Mixed Linear Model (MLM) (C), Compressed MLM (CMLM) (D), Enriched Compressed MLM (ECMLM) (E), Settlement of MLM Under Progressively Exclusive Relationship (SUPER) (F), Multiple Loci Mixed linear Model (MLMM) (G), and Fixed and random model Circulating Probability Unification (FarmCPU) (H).
结论
综上所述, 8中模型中FarmCPU在关联准确性和控制假阳性和假阴性方面都优于其他模型。
参考文献:https://www.frontiersin.org/articles/10.3389/fpls.2019.01794/full
延伸阅读
- 发表于 2021-11-18 15:23
- 阅读 ( 5783 )
- 分类:GWAS
你可能感兴趣的文章
- AI 写代码之绘制GWAS 曼哈顿图 330 浏览
- TWAS分析的原理和工具 2287 浏览
- 如何从GWAS结果中获取表型解释率PVE——TASSEL、GEMMA篇 8911 浏览
- BLINK(C版本) 进行 GWAS分析的教程 4717 浏览
- 一行代码检查GWAS分析中表型数据的错误 1405 浏览
- 群体遗传进化与GWAS分析镜像更新2.0 1856 浏览
相关问题
- 全基因组关联分析GWAS教程中的beagle填充问题 1 回答
- GWAS分析的教程中06.impute的beagle填充出现问题 2 回答
- 老师您好,针对这个问题https://www.omicsclass.com/question/7725。你们提到:你这个数据里面是不是P值有0的情况,取了对数就无穷大了,导致出问题。但是,我发现里面没有P值为0的情况,那是不是脚本gwas_manhattan_plot.r有问题呢?需要修改gwas_manhattan_plot.r脚本的什么地方才可以解决顶部只能画半圆的问题呢?还有,我发现P-Value的原始文件有NaN的情况,是什么原因呢?是否对GWAS有影响呢? 1 回答
- GWAS 曼哈顿 Manhattan 图像问题 1 回答
- R语言GAPIT跑GWAS,ERROR求解:不能有负长度矢量,万分感谢!!! 1 回答
- R语言GAPIT跑GWAS错误 Error in ‘[.data.frame‘(GD,,!is.na(GI.MP[,3])):选择了未定义的列 0 回答
