11月8日见刊(IF=4.5)水稻U-box E3基因家族分析文献解析
今天我给大家带来一篇11月8日刚刚在线发表的水稻U-box E3家族的文章,发表在International Journal of Molecular Sciences期刊上,影响因子IF=4.556,我们就一起看看这篇文章都写了哪些内容,希望对您的思路有所帮助。
U-box E3 Ubiquitin Ligase简介
泛素蛋白酶体途径是己知的所有真核生物体内具有高度选择性的最为重要的蛋白质降解途径。真核细胞中泛素化修饰后的靶蛋白可能被降解、可能被转移到细胞或细胞外的特定部位,也有可能导致靶蛋白的功能发生变化,这主要取决于靶蛋白所加的泛素链的结构,以及泛素链的长短。泛素连接酶E3决定靶蛋白的特异性识别,能够将泛素分子连接到靶蛋白的某个赖氨酸上,在泛素途径中具有重要的作用。
文章结果内容
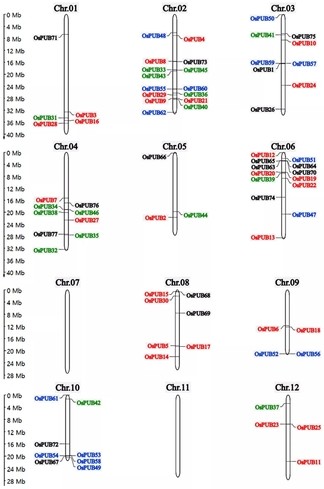
1. 水稻中U-box E3泛素连接酶(OsPUBs)染色体分布
2008年li-rong-zheng等已经报道了在水稻中鉴定的77个U-box E3泛素连接酶基因(OsPUBs),但基因家族分析内容涉及不是太多,详见原文:https://www.cell.com/molecular-plant/pdf/S1674-2052(14)60357-9.pdf。
所以作者直接使用上述论文中已鉴定好的77个OsPUBs基因信息,绘制染色体分布图:
2. 水稻中U-box E3泛素连接酶(OsPUBs)进化及基因结构分析
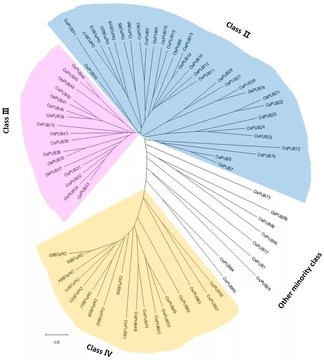
作者使用ClustalW做了多序列比对,并根据蛋白全长序列进一步构建进化树,并根据拟南芥该家族基因分组信息分组,将如下图。
77个OsPUBs基因结构如下图:
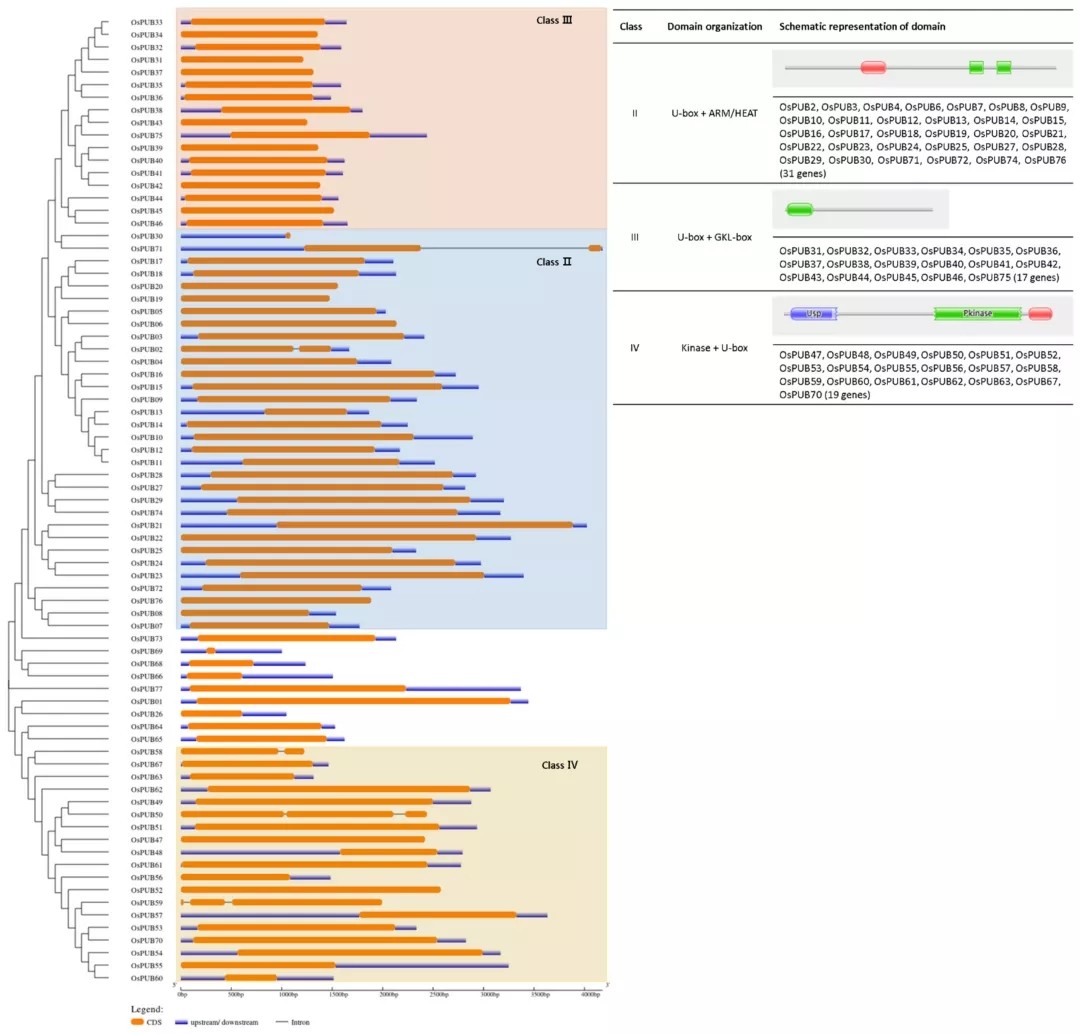 77个OsPUBs Motif分析,如下图:
77个OsPUBs Motif分析,如下图:
3. 水稻中U-box E3泛素连接酶(OsPUBs)在生物及非生物胁迫下的表达模式分析
为了阐明水稻中U-box E3泛素连接酶(OsPUBs)在干旱(非生物)和真菌侵染(生物)胁迫下的表达模式变化,作者分别作者转录组实验。
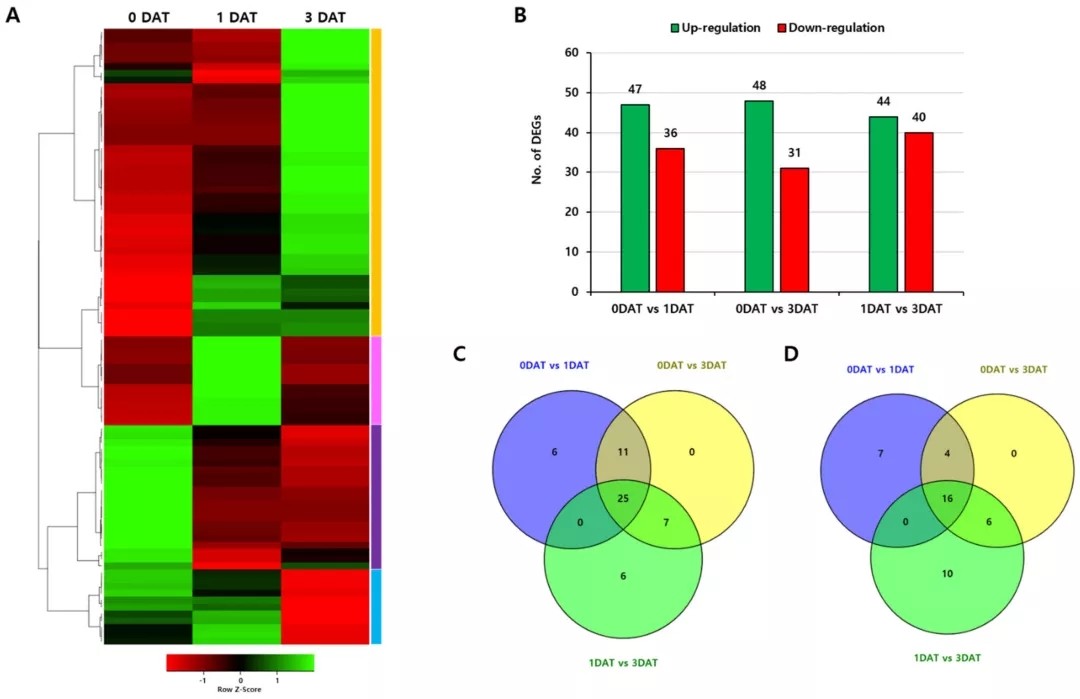 两种胁迫下水稻中U-box E3泛素连接酶(OsPUBs)表达模式变化如下表:
两种胁迫下水稻中U-box E3泛素连接酶(OsPUBs)表达模式变化如下表: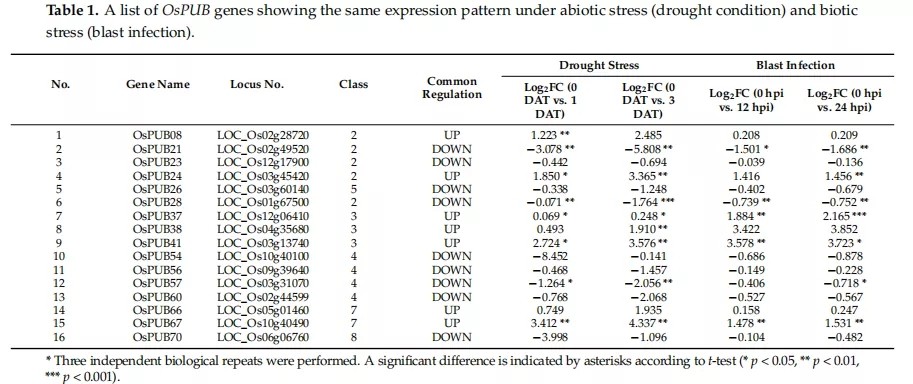
4. 水稻中U-box E3泛素连接酶(OsPUBs)顺式作用元件分析
明确了水稻中U-box E3泛素连接酶(OsPUBs)在胁迫下的表达模式变化,作者进一步分析了OsPUBs基因上游顺式作用元件,以期阐释OsPUBs基因的表达的调控机制,并选择了18类元件在5种作物(水稻、玉米、大麦、小麦、马铃薯)中进行的详细比较,如下图:
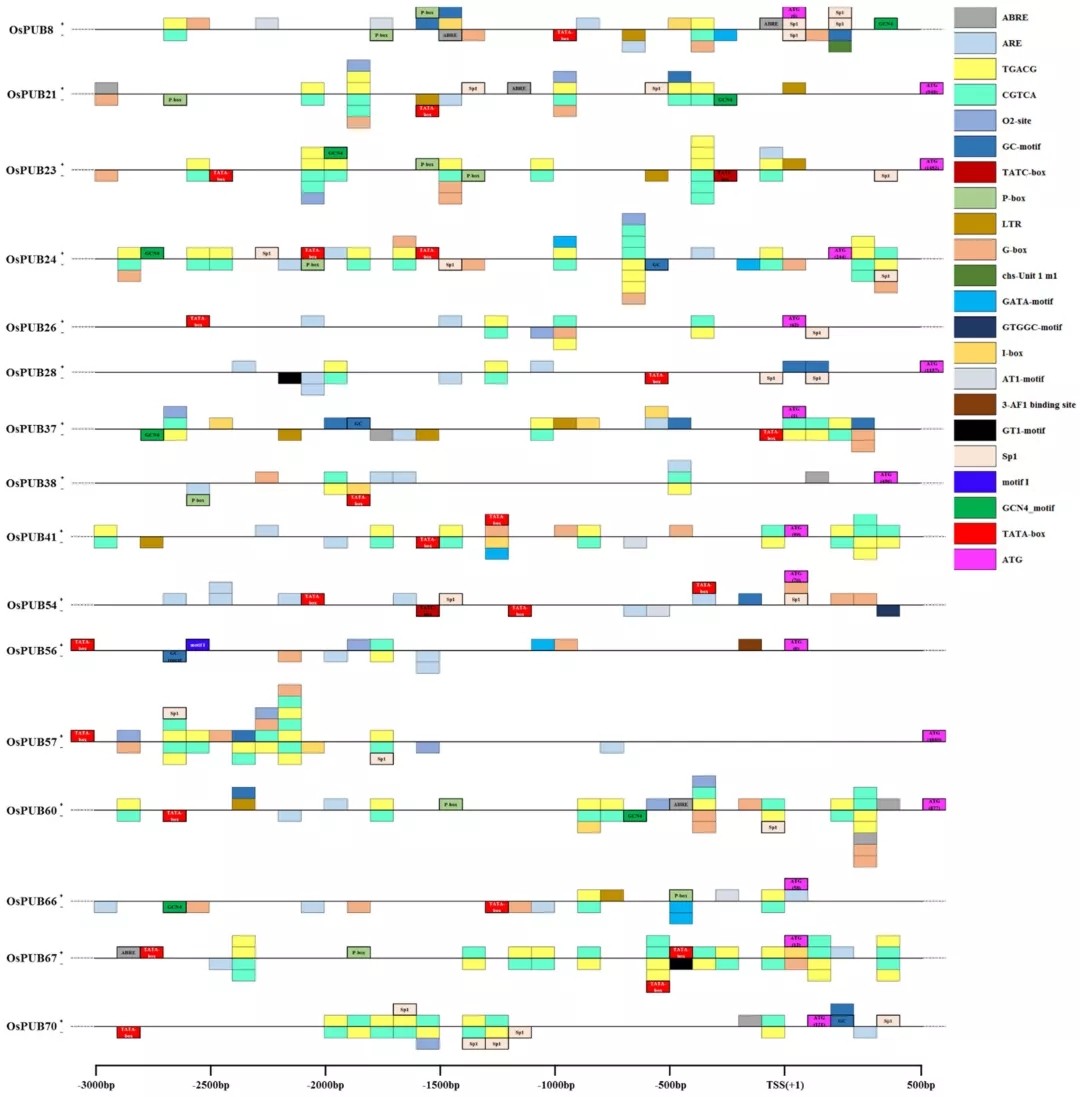
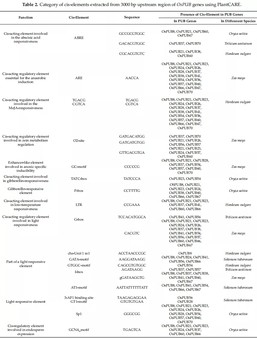
5. 水稻中U-box E3泛素连接酶(OsPUBs)在生物及非生物胁迫下的表达的qRT-PCR验证
作者从77个OsPUBs基因中选择16个基因,用qRT-PCR方法验证了在干旱和真菌侵染下的表达模式变化,如下图:
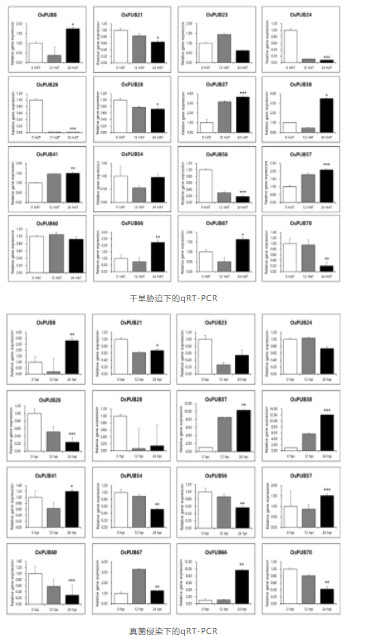
总结
以上就是该篇文章全部结果内容,无非还是基因家族分析的内容,但是该文章在其他人已经报道过该家族基因的情况下,又发了一篇,真是硬核!有胆识!不过也从侧面反映了基因家族分析类文章在审稿人的眼中认可度是真高啊!
看到这里,你是不是也动心了呢?趁着好发赶紧行动起来吧!
1. 想学习基因家族实操分析请点击学习:最新版课程学习链接:基因家族分析实操课程
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,又能发文章,又能学生信技能,一举两得的SCI好思路;基因家族分析课程已更新分析内容,最新版课程学习链接:基因家族分析实操课程
2. 转录组数据结果理解不深入?图表看不懂?这就是你转录组数据不会挖掘、文章不会撰写的原因,让我带你一起深入了解转录组数据结果,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?多学点数据处理技能:学习链接:转录组标准分析后的数据挖掘
5.微生物多样性分析很简单,但是分析内容项目并不少,理解起来有困难?看我深入浅出讲给你听,学习链接: 微生物16S/ITS/18S分析原理及结果解读
6. 学生物的必学生信技能:Docker安装与使用、linux系统入门、Perl语言入门到精通、Python生信入门到精通、perl语言高级编程
7. 生信绘图、科研绘图技能:Cytoscape与网络图绘制、微生物OTU网络图绘制(Cytoscape)、R语言基础与绘图、R语言绘图基础(ggplot2)
8.生物信息实战技能,0基础也可以学会,内含脚本及demo数据:RNAseq有参转录组自主分析、基因组重测序自主分析、微生物多样性自主分析
9. 更多学习内容:linux、perl、R语言画图,更多免费课程请扫描下方二维码进入组学大讲堂网校学习:
- 发表于 2022-01-19 22:02
- 阅读 ( 3092 )
- 分类:文献解读
你可能感兴趣的文章
- 2022年基因家族第1篇—大型动物基因家族分析怎么写?! 5167 浏览
- 2区SCI赏析—木麻黄中MYB基因家族鉴定及其在盐胁迫中的响应 5061 浏览
- 第三期《基因家族分析实操》直播课开始报名了! 2771 浏览
- 《基因家族分析实操》直播培训(第二期)火热报名中! 4316 浏览
- 2020年-基因家族分析文章怎么发! 8017 浏览
相关问题
- 基因家族成员系统发育树 0 回答
- 获取基因与mRNA的对应关系,注意文件中的位置mRNA的位置; #perl script/mRNAid_to_geneid.pl Arabidopsis_thaliana.TAIR10.41.gff3 mRNA2geneID.txt 1 回答
- 果实发育时期的转录组热图中,有几个基因在各个时期均表现低FPKM值,请问是转录组数据有误吗? 2 回答
- 合并提取后的domain序列之后,linux系统中的clustalw不能读出蛋白信息 3 回答
- 基因家族分析课程get_fa_by_id.pl脚本问题 1 回答
- 基因家族分析——有几个保守结构域,对应的有几个pfm号 1 回答

