蛋白信号肽预测—SignalP使用详解
信号肽是是引导新合成的蛋白质向分泌通路转移的短肽链,长度约5-30个氨基酸;对应的mRNA序列,在起始密码子后,有一段编码疏水性氨基酸序列的RNA区域,该氨基酸序列就被称为信号肽序列,它负责把蛋白质引导到细胞含不同膜结构的亚细胞器内。
今天我给大家介绍一款鉴定信号肽的在线软件,简单易用。
SignaIP简介
SignaIP是目前应用最广泛信号肽在线预测软件,目前SignaIP目前已经更新到5.0版本,不过小编不太喜欢最新版本的结果展示形式,仍在使用SignaIP4.1版本,结果信息展示更精准明了。
SignaIP4.1地址:http://www.cbs.dtu.dk/services/SignalP-4.1/ 。SignaIP4.1页面如下:

SignaIP的使用
SignaIP使用很简单,首先是上传需要分析的蛋白序列,如下图,待分析的蛋白序列上传有两种方式:可在粘贴框中单条序列上传;也可以可以点击选择文件直接上传FASTA文件,每次上传最多提交2000个序列和200000个氨基酸,每条序列不超过6000个氨基酸。
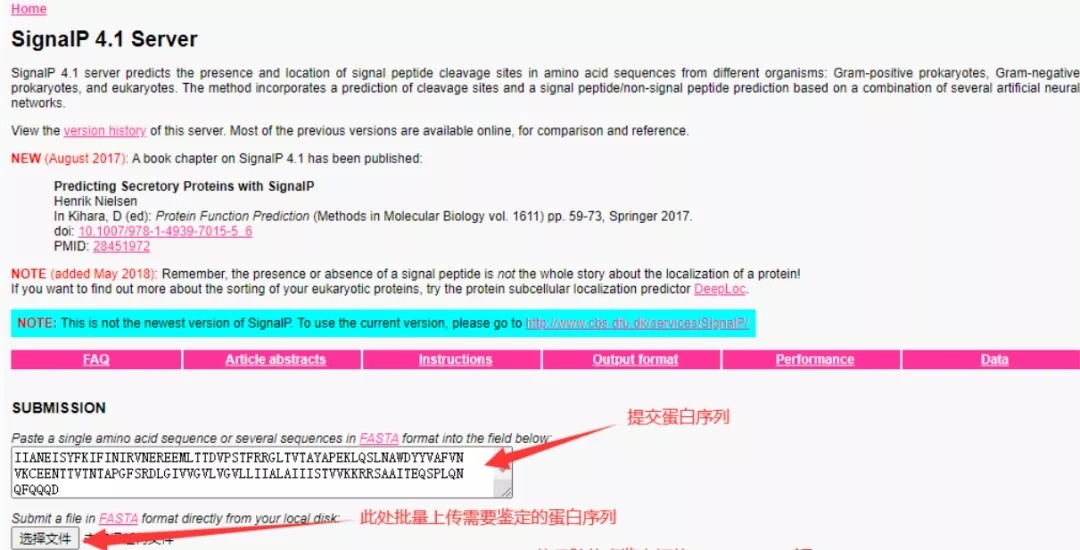
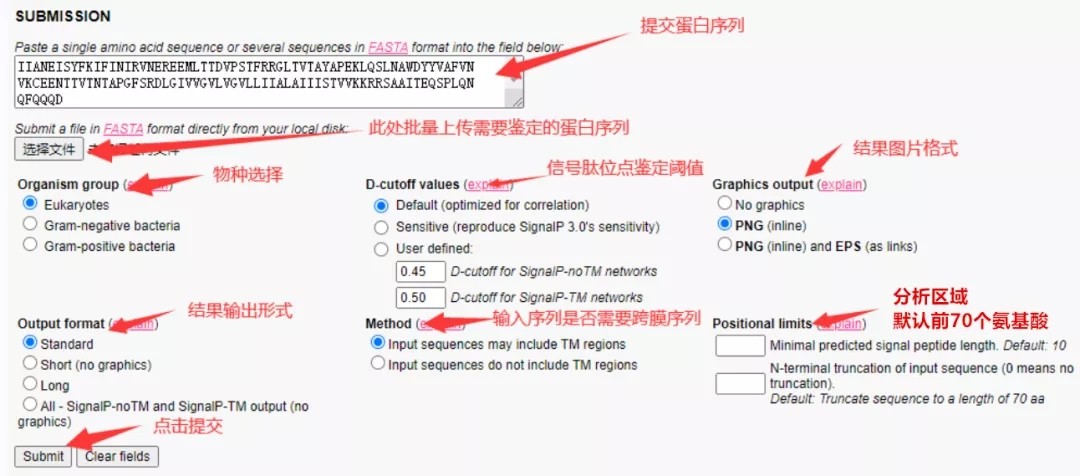
提交完蛋白序列,接下来进行一些简单设置,如下图,由于示例基因来源于动物,所以物种选择真核生物;D-cutoffvalues一般默认即可;结果图片格式可以选择第三个PNG and EPS(矢量格式);输出格式一般选择Standard 和Long;Method选项是区分蛋白序列中是否存在跨膜区域(TM regions),假如明确知道无跨膜区域,就选择不包括跨膜区域预测会更准确,其余选择第一个;分析区域可以自行设置,默认是前70个氨基酸,一般保持默认即可。
设置完成后,点击Submit即开始分析。
SignaIP结果解读
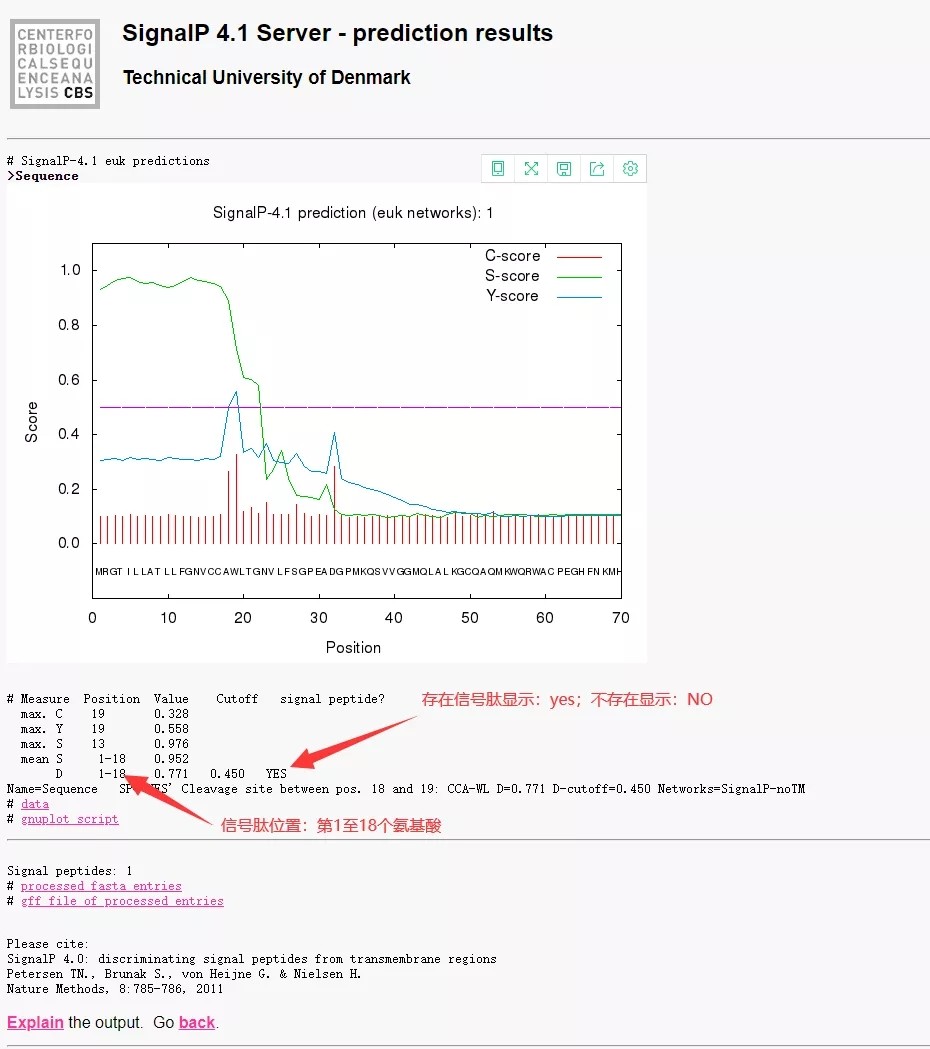
下面即是SignaIP分析结果,其中有3个比较重要的打分介绍一下:
C-score (raw cleavage site score):用来区分是否为剪切位点,最高峰值为剪切位点后的第一个氨基酸(即成熟蛋白的第一个氨基酸残基);
S-score (signal peptide score):用来区分相应位置是否为信号肽区域;
Y-score (combined cleavage site score):C-score和S-score的几何平均数,用于避免多个高分C-score值对结果的影响;
在分泌蛋白的预测结果中,NN法Signal peptide列中结果为yes或者NO,并根据C值、S值和Y值等给出潜在的剪切位点;图表右上角处有C值、S值和Y值的曲线颜色指示,图表中有各值的变化趋势曲线,如下图。
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,又能发文章,又能学生信技能,一举两得的SCI好思路;基因家族分析课程已更新分析内容,最新版课程学习链接:基因家族分析实操课程
2. 转录组数据结果理解不深入?图表看不懂?这就是你转录组数据不会挖掘、文章不会撰写的原因,让我带你一起深入了解转录组数据结果,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?多学点数据处理技能:学习链接:转录组标准分析后的数据挖掘
5.微生物多样性分析很简单,但是分析内容项目并不少,理解起来有困难?看我深入浅出讲给你听,学习链接: 微生物16S/ITS/18S分析原理及结果解读
6. 学生物的必学生信技能:Docker安装与使用、linux系统入门、Perl语言入门到精通、Python生信入门到精通、perl语言高级编程
7. 生信绘图、科研绘图技能:Cytoscape与网络图绘制、微生物OTU网络图绘制(Cytoscape)、R语言基础与绘图、R语言绘图基础(ggplot2)
8.生物信息实战技能,0基础也可以学会,内含脚本及demo数据:RNAseq有参转录组自主分析、基因组重测序自主分析、微生物多样性自主分析
9. 更多学习内容:linux、perl、R语言画图,更多免费课程请扫描下方二维码进入组学大讲堂网校学习:
- 发表于 2022-01-19 22:17
- 阅读 ( 49510 )
- 分类:软件工具

