如何鉴定长距离运输的mRNA?
上一期给大家介绍了一篇如何鉴定嫁接中长距离运输mRNA的方法:嫁接中长距离运输的mRNA是如何鉴定的?今天给大家介绍已经发表的第2种鉴定长距离运输的mRNA的方法,两者方法类似,但也各有不同,该方法引入了SNP用以排除假阳性reads。今天着重介绍下大家都非常关心的实验设计以及分析思路。
文章简介
该篇文章是中国农业大学高丽红教授在International Journal of Molecular Sciences杂志发表题为“Identification of Long-Distance Transmissible mRNA between Scion and Rootstock in Cucurbit Seedling Heterografts”的研究论文,该文章也是通过重测序矫正基因组与转录组reads比对方式相结合去除假阳性来鉴定黄瓜-南瓜嫁接体中mRNA 的长距离运输。

实验设计
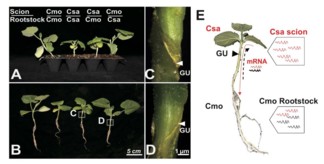
嫁接组合设置:作者共设四个嫁接组合,如下图,同源嫁接2个:黄瓜Csa/黄瓜Csa,南瓜Cmo/南瓜Cmo;异源嫁接2个:黄瓜Csa/南瓜Cmo,南瓜Cmo/黄瓜Csa。
取样:1. 分别取南瓜和黄瓜的叶片和根,混合提取DNA,用于重测序建库测序。2. 每嫁接组合,9株植株作为一个生物学重复,共3个生物学重复,均取接穗的第一子叶(包括叶柄)以及砧木的根混合提取总RNA,分别用于转录组测序。
分析思路
基因组重建:由于作者所有黄瓜和南瓜材料与参考基因对应材料之间存在差异,所以作者利用重测序数据(120x)替换原参考基因上的SNP、Indel等变异信息,重新构建优化了基因组。
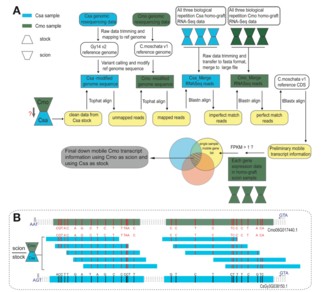
转运mRNA识别:如下图,以南瓜接穗黄瓜砧木为列,识别有接穗南瓜向下运输至黄瓜砧木的mRNA,分析过程较为复杂,我们分条来说:
(1)首先用黄瓜砧木根的转录组数据和重建的黄瓜参考基因进行比对,取未必对上的reads再与重建后的南瓜基因组进行比对,比对上的reads用于后续分析;
(2)取步骤1中比对到南瓜基因上的reads再与来自于黄瓜同源嫁接接穗和根转录组合并而来的reads进行blastn比对,不完美匹配的reads(至少存在2个及以上SNP)再次和重建后的南瓜基因组进行比对,比对上的reads用于后续分析;
(3)经过以上多次比对,在步骤2中的再次和南瓜基因组匹配的reads被认为是来自于接穗南瓜向下转运的mRNA,最后再将这些reads与南瓜CDS序列进行blast比对得到转运mRNA基因ID。
两种方法比较
综合来看,两种方法思路类似,但也各有千秋,在基因组重建过程中,第一篇方法中多了物种特异基因组装过程这一步,而本篇文章在RNA数据比对时引入SNP概念也是一种方法创新。
经常遇到小伙伴问:哪种方法更好呢?我想两种方法取其优点结合一下才更好,有对此类长距离运输信使分子鉴定感兴趣的小伙伴欢迎添加微信:llcheng1314 咨询。
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,又能发文章,又能学生信技能,一举两得的SCI好思路;基因家族分析课程已更新分析内容,最新版课程学习链接:基因家族分析实操课程
2. 转录组数据结果理解不深入?图表看不懂?这就是你转录组数据不会挖掘、文章不会撰写的原因,让我带你一起深入了解转录组数据结果,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?多学点数据处理技能:学习链接:转录组标准分析后的数据挖掘
5.微生物多样性分析很简单,但是分析内容项目并不少,理解起来有困难?看我深入浅出讲给你听,学习链接: 微生物16S/ITS/18S分析原理及结果解读
6. 学生物的必学生信技能:Docker安装与使用、linux系统入门、Perl语言入门到精通、Python生信入门到精通、perl语言高级编程
7. 生信绘图、科研绘图技能:Cytoscape与网络图绘制、微生物OTU网络图绘制(Cytoscape)、R语言基础与绘图、R语言绘图基础(ggplot2)
8.生物信息实战技能,0基础也可以学会,内含脚本及demo数据:RNAseq有参转录组自主分析、基因组重测序自主分析、微生物多样性自主分析
9. 更多学习内容:linux、perl、R语言画图,更多免费课程请扫描下方二维码进入组学大讲堂网校学习:
- 发表于 2022-03-25 20:43
- 阅读 ( 2295 )
- 分类:转录组

