快速高效KM生存曲线绘制-脚本使用
Kaplan-Meier法是现如今生信分析最常用的方法之一,先通过计算出活过一定时期的患者再活过下一时期的概率(即生存概率),然后将逐个生存概率相乘,得到相应时段的生存率。基于这一方法,绘制出KM生存曲线能够直观展现分类变量(如治疗A vs 治疗B,男 vs 女等)对患者生存造成的影响。
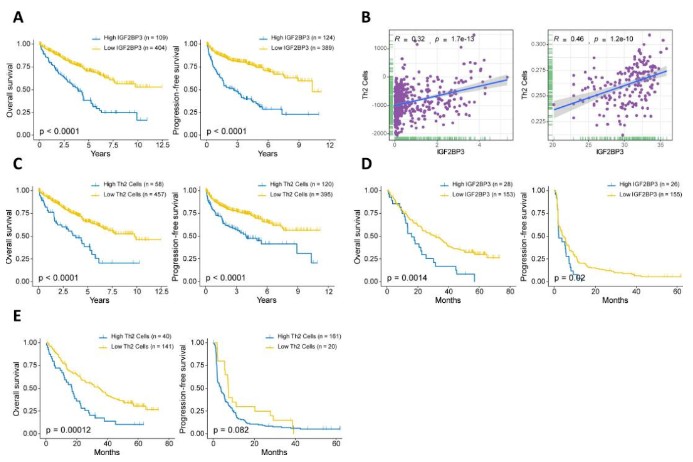
例如:2021年发表在The Journal for ImmunoTherapy of Cancer上的一篇影响因子为13.75的文献为例,该文献中运用KM生存曲线生动形象地展现了不同变量分别对患者生存的影响。
作者根据基因表达量和细胞浸润程度的高低对样本进行分组,以总生存期(OS)绘制KM生存曲线,随着时间的推移,生存率从1逐渐下降,但可以看出黄色生存曲线始终高于蓝色生存曲线,即说明其代表的低表达组/低浸润组的生存率更高。同时图中还添加了基于Log-rank检验比较两组生存曲线得到的P值,通过P值可以判断两组生存曲线之间是否有统计学差异,一般认为P值小于0.05即表示两组间有显著的生存差异。
为了使大家更简便快捷地绘制出精美的KM生存曲线,这里我们给大家提供一个绘制KM生存曲线的R脚本,这个脚本只需要准备好相应的输入文件,再进行简单的命令行操作即可绘制出KM生存曲线,判断该变量对患者生存是否有显著影响。
使用命令示例如下
#输入变量为连续变量时,取连续变量的中位值对样本进行分组,绘制KM生存曲线
Rscript /share/work/fangs/scripts/tcga/km_plot.r -m metadata_group.tsv \
-t TIME -s EVENT --median_cut -g IRPS -n IRPS_group -o kmplot -p IRPS -H 6 -W 6
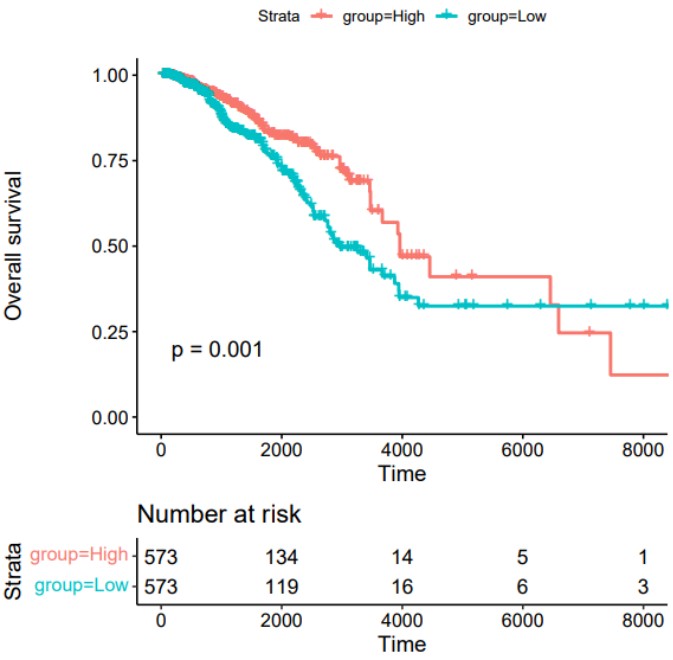
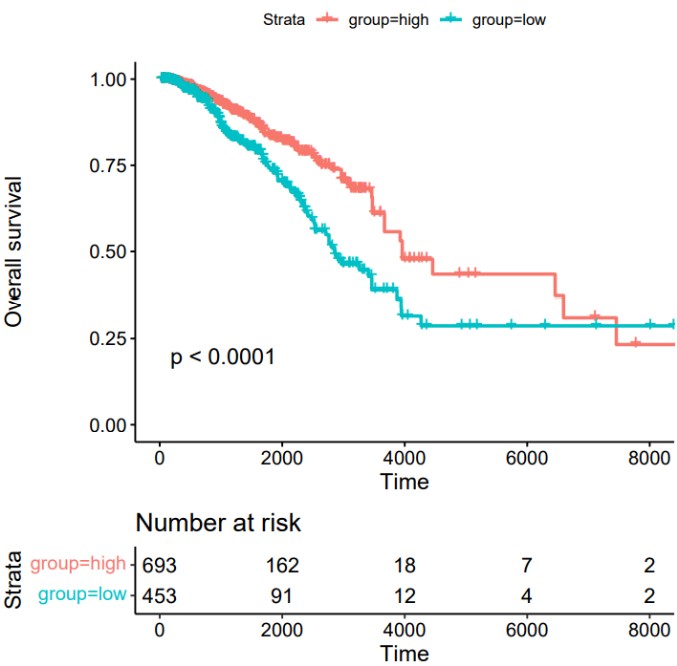
#输入变量为连续变量时,通过survminer包的surv_cutpoint函数取连续变量的最佳截断值对样本进行分组,绘制KM生存曲线
Rscript /share/work/fangs/scripts/tcga/km_plot.r -m metadata_group.tsv \
-t TIME -s EVENT --surv_cut -g IRPS -n IRPS_group -o kmplot -p IRPS -H 6 -W 6
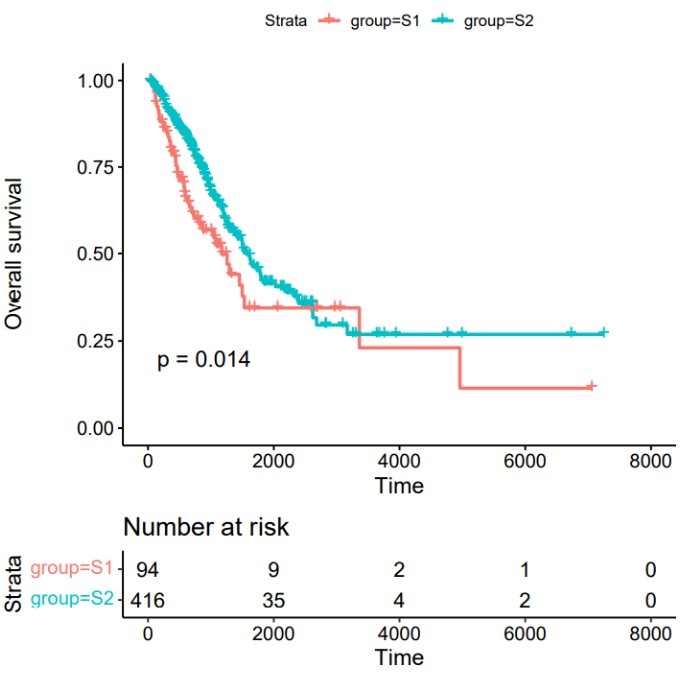
#输入变量为分类变量时,可直接绘制KM生存曲线
Rscript /share/work/fangs/scripts/tcga/km_plot.r -m metadata_group.tsv \
-t TIME -s EVENT -g Subtype -o kmplot -p DDR_Subtype -H 6 -W 6
输入文件准备
这个脚本所必需的输入文件只有一个,通过-m参数指定(metadata_group.tsv),文件中每一行为一个样本,列必须包含可对样本进行分组的分类变量或连续变量以及样本的生存数据,如下列表格所示:
| barcode | TIME | EVENT | IRPS | Subtype |
| TCGA-E2-A158-11A-22R-A12D-07 | 450 | 0 | 17.53 | S1 |
| TCGA-BH-A0DD-11A-23R-A12P-07 | 2486 | 0 | 16.79 | S2 |
| TCGA-BH-A1EO-11A-31R-A137-07 | 2798 | 1 | 19.29 | S2 |
更多脚本参数设置及说明
初次使用脚本时,可以通过-h参数获得以下帮助信息。
Rscript km_plot.r -h
usage: km_plot.r [-h] -m metadata [--surv_cut] [--median_cut] [-l] [-t time]
[-s status] [-g group] [-n group_name] [-y ylab] [-H HEIGHT]
[-W WIDTH] [-o outdir] [-p prefix]
KM plot
optional arguments:
-h, --help show this help message and exit
-m metadata, --metadata metadata
input the path to the clinical information file
containing survival data[required]
--surv_cut Whether to group by surv_cutpoint
function[optional,default:False]
--median_cut Whether to group by median[optional,default:False]
-l, --log2 Whether to do the log2 transformation of a continuous
variable[default FALSE]
-t time, --time time Specifies the column name for the
lifetime[optional,default:OS_Time]
-s status, --status status
Specifies the survival event column
name[optional,default:OS_Status]
-g group, --group group
Specifies the column name that contains grouping
information[optional,default:m6acluster]
-n name, --name name
The column name of the sample grouping information
after the samples are grouped according to continuous
variables[optional,default:group]
-y ylab, --ylab ylab Image y axis name[optional,default:Overall survival]
-H HEIGHT, --height HEIGHT
the height of dumbbell plot[optional,default 6]
-W WIDTH, --width WIDTH
the width of dumbbell plot[optional,default 8]
-o outdir, --outdir outdir
output file directory[optional,default cwd]
-p prefix, --prefix prefix
out file name prefix[optional,default m6a]
根据自身需要调整必要的参数:
-t 输入列名,指定样本的生存时间;
-s 输入列名,指定样本的生存状态(死亡为1,存活为0);
-g 输入列名,指定根据哪一个变量对样本进行分组,仅可分为两组;
-y 输入KM生存曲线中y轴的名称,一般根据样本生存时间的类型进行调整,如以样本总生存期(OS)绘制则可命名为Overall survival。
--median_cut和--surv_cut以及-n这三个参数比较特殊,当针对的变量为分类变量时,这三个参数可不添加,直接根据分类变量对样本进行分组,此时仅生成KM生存曲线图。而当针对的变量为连续变量(如某个基因的表达量)时,就必须指定--median_cut和--surv_cut两个参数其中之一,同时还必须指定-n参数。指定--median_cut是对连续变量取中位值从而将样本分组;--surv_cut则是对连续变量通过survminer包的surv_cutpoint函数取最佳截断值从而将样本分组;-n参数是根据连续变量将样本分组后,指定包含该分组信息的列名,此时除了生成KM生存曲线图,还会在metadata_group.tsv的基础上加入新的样本分组信息并输出。
其他参数说明
--log2 加上该参数即要对连续变量进行log2转化;
-H 指定输出图片的高度,默认高为6英寸;
-W 指定输出图片的宽度,默认宽为8英寸;
-o 指定结果输出的路径,默认为当前路径;
-p 指定输出文件名字的前缀。
脚本获取方法
在“生信博士“公众号回复kmplot即可获得网盘链接以及提取码。
如何使用命令行的方法分析数据
可能有的人没有用过命令的形式分析数据, 可以学习下面的课程入门一下:

- 发表于 2022-06-10 14:16
- 阅读 ( 4570 )
- 分类:R
你可能感兴趣的文章
- 不同的生存曲线含义 OS DFS 6096 浏览
- Cox与KM生存分析及结果解读 38135 浏览
- cox回归PH假设检验 12032 浏览
- KM-plotter在线生存分析 14785 浏览
- GEPIA--TCGA癌症数据挖掘网站 12269 浏览
相关问题
- 生存分析 0 回答
- 老师,想请教一下TCGA基因表达数据的问题,我从xena.ucsc网页上下载了基因表达数据TCGA-CESC.htseq_counts.tsv;然后发现该数据中只有Ensembl格式的基因ID ,没有SYMBOL格式的。所以接下来进行基因ID格式转换,却发现同一个SYMBOL ID对应的多个Ensembl格式的ID,想问下老师,这种情况该怎么处理?同一个SYMBOL ID所对应的多个Ensembl格式ID的基因表达数据应该留下哪一个? 1 回答
- 老师,请教您一个生存分析的问题,为什么phenotype的删失数据天数一列中会出现alive time=0的情况,是病人还没开始实验观察就去世了吗? 0 回答
- 生存分析:老师,我看到了这个生存分析的代码在利用索引定义一个新的向量时先定义了一个,想请教一下您他为什么刚开始要定义一个纯0向量呢,先谢谢您啦 1 回答
- 计算每一个单因素cox分析,lapply函数,好久都没有反应,怎么看进度啊,要等多久啊? 1 回答
- 你好,我想问下大佬做生存分析,怎样用ROC曲线寻找cut-off值? 0 回答
