进化树美化操作详解- iTOL
绘制进化树,是科研人的基础技能之一,而一张好看的进化树图,不仅能直观得展现基因或物种间的发育进化关系,更能增加文章的观赏性,赢得审稿人的青睐。今天给大家介绍的是一款非常实用且操作简便的在线进化树美化工具—iTOL软件。接下来通过实操来展示一下iTOL进行进化树美化的基础操作。
一、数据准备
在进化树绘制之前要进行进化树构建,进化树构建分为两步:
多序列比对:准备一组氨基酸序列或蛋白质序列,多序列比对常用软件为muscle、mafft和clustalW,或者直接选择MEGA软件进行比对。
比对结果建树:常用建树方法有基于距离和基于特征/性状两种,我们选择基于距离的邻接法(Neighber-joining)构建进化树,将比对结果直接导入MEGA软件上进行操作,bootstrap选择1000。
二、基础操作
1. 进入iTOL网站(https://itol.embl.de/)注册账户,点击Tree upload即可上传已经构建成功的.nwk格式树文件。
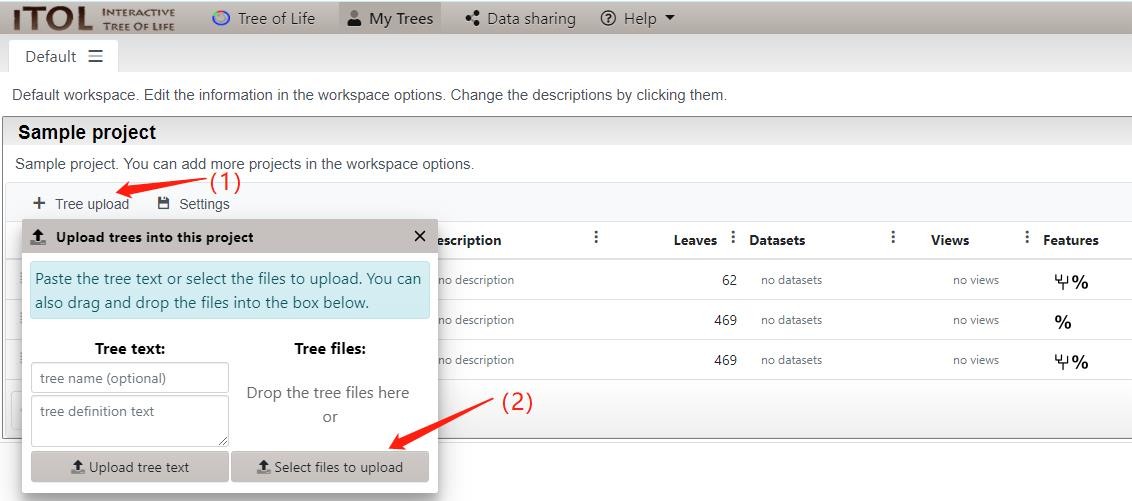
2. 点击上传的树文件进入操作界面,我们主要通过右上角的控制面板进行基本操作。
(1)在Basic面板中,有Mode options、Label options、Branch options三大板块,Basic面板主要负责修改树的形状、方向,叶标签的字体、字号、位置,还有进化枝的粗细、颜色等基础设置。
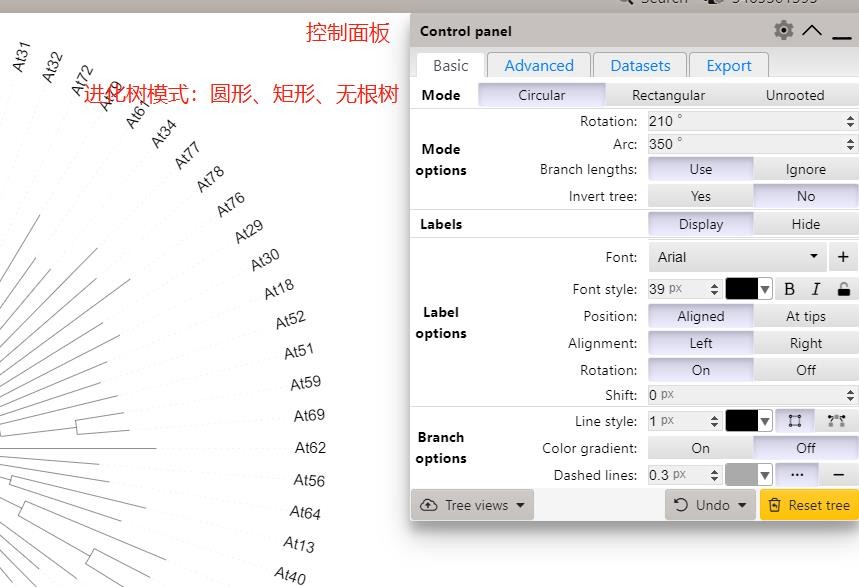
Mode:进化树模式选择,可以绘制开口圆形、矩形、无根树。这边以开口圆形进化树为例。
Rotation:旋转进化树,调整圆形开口方向。
Arc:调整圆形开口角度大小。
Branch lengths:是否展示进化枝的枝长。
Invert tree:是否将延伸方向由根枝叶转换成叶在内侧,进化枝在外侧。
Labels:是否展示叶标签(leaf)。
Font:叶标签的字体,点击右侧加号也可以导入新字体。
Font style:调整标签字号、颜色、加粗、倾斜。
Position:叶标签之间对齐or标签放在枝顶端与枝对齐。
Alignment:叶标签字母靠左对齐or靠右对齐。
Rotation:选择“On”使左侧标签旋转180°便于阅读。
Shift:标签与进化枝之间距离。
Line style:调整进化枝粗细、颜色、clade的棱角选择。
Color gradient:控制进化枝颜色是否渐变。
Dashed lines:进化枝与标签之间连接线。
(2)Advanced面板主要负责展示bootstrap(置信度)的特征。
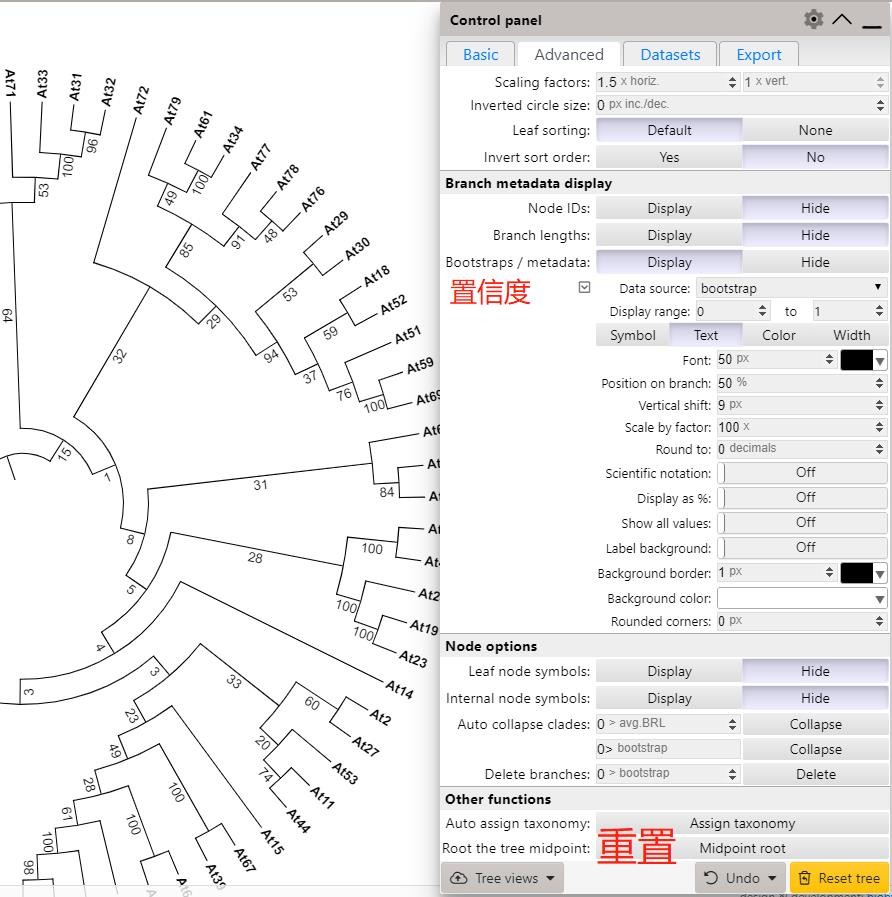
如图所示,Bootstraps/metadata参数设为Display。
Display range:Bootstrap显示范围,过低的置信度值可以不显示。
Symbol、Text、Color、Width:bootstrap展示方式,常用文本形式展示bootstrap。
Text下方为其参数,常用Font(字体)和Scale by fater(数值倍数)两个参数。
最下方的Undo和Reset tree都是重置设置,Undo是重置和撤销branch、label及其它美化样式设置,Reset是直接恢复原始进化树样式。
三、全局美化
前文是一些基础操作设置,接下来就该是真正的进化树美化了。
iTOL的进化树美化需要通过配置文件来完成,无论是参数类型的,例如给不同分支划分颜色、不同物种基因添加标记;还是数据表类型的,例如给进化树增加热图、基因结构图、表达丰度图等……
处理好的配置文件需要在Datasets面板处上传,配置文件模板可以通过官方下载,点击最上方Help进入Help pages页面。在.txt模板右侧展示了该模板的美化结果,根据自己需要下载对应的模板或参数。
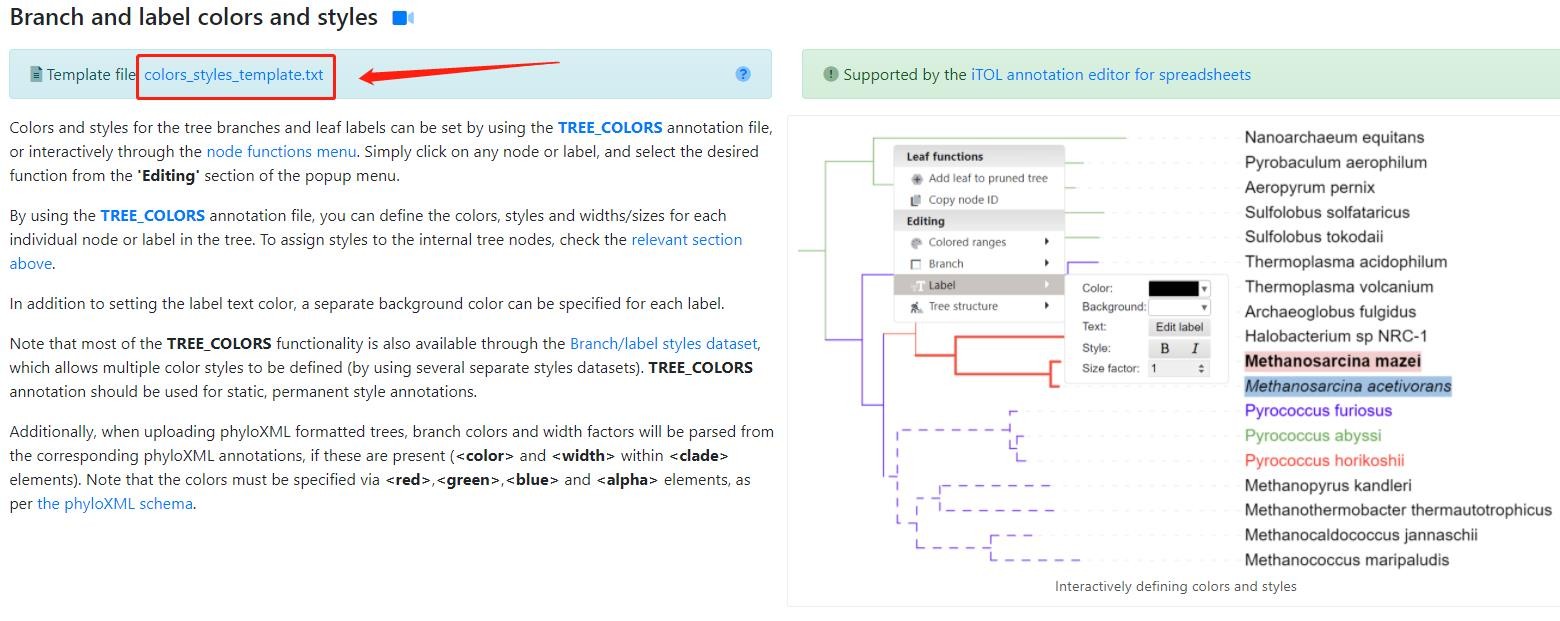 除了通过上传配置文件之外,iTOL还提供局部的leaf、branch、clade修改。
除了通过上传配置文件之外,iTOL还提供局部的leaf、branch、clade修改。
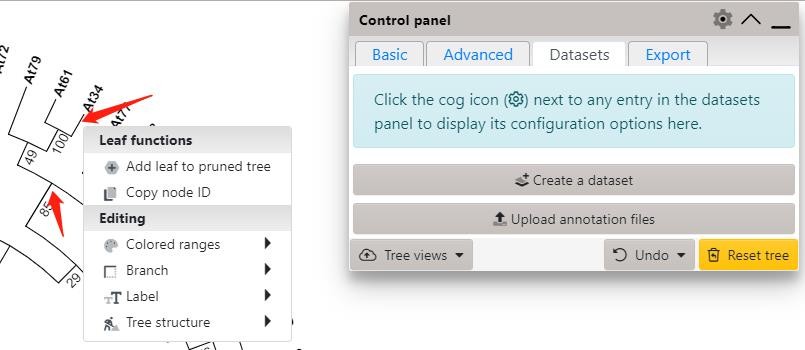
将鼠标光标悬停到修改处上方,鼠标键左击后会出现Clade functions/Leaf functions字样的浮动面板,之后可以在Editing下方参数中修改局部的Branch或Label信息。
接下来放上一些官方展示的进化树美化图:
四、结果导出
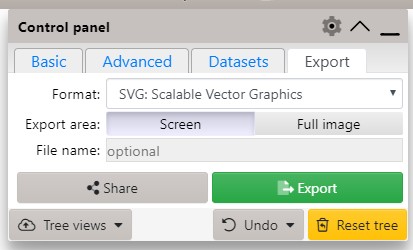
如果没有付费订阅iTOL,美化完成后需要将结果及时导出,因为iTOL只会保存账号下的原始树文件,二次登录美化参数会丢失。
 好了,今天就介绍到这里,感兴趣的去试试吧!
好了,今天就介绍到这里,感兴趣的去试试吧!- 发表于 2022-10-12 15:53
- 阅读 ( 24044 )
- 分类:软件工具
你可能感兴趣的文章
- itol添加圆环分组和节点标签时位置调整 4985 浏览
