火山图--ggplot2
利用ggplot2进行火山图的绘制
ggplot2包绘制火山图
加载R包以及案例数据temp1
library('ggplot2')
head(temp1,10)
-log10FDR logFC Regulate
64170 1.971938 -2.2884388 Down
6674 1.864104 -1.2460804 Down
7401 1.864104 1.2950939 Up
57217 1.864104 -1.2676311 Down
50613 1.801074 1.6640064 Up
3433 1.801074 4.2696415 Up
54830 1.801074 -1.9012811 Down
54886 1.801074 -1.5446208 Down
6355 1.801074 1.4273926 Up
338324 1.801074 0.9592775 Normal
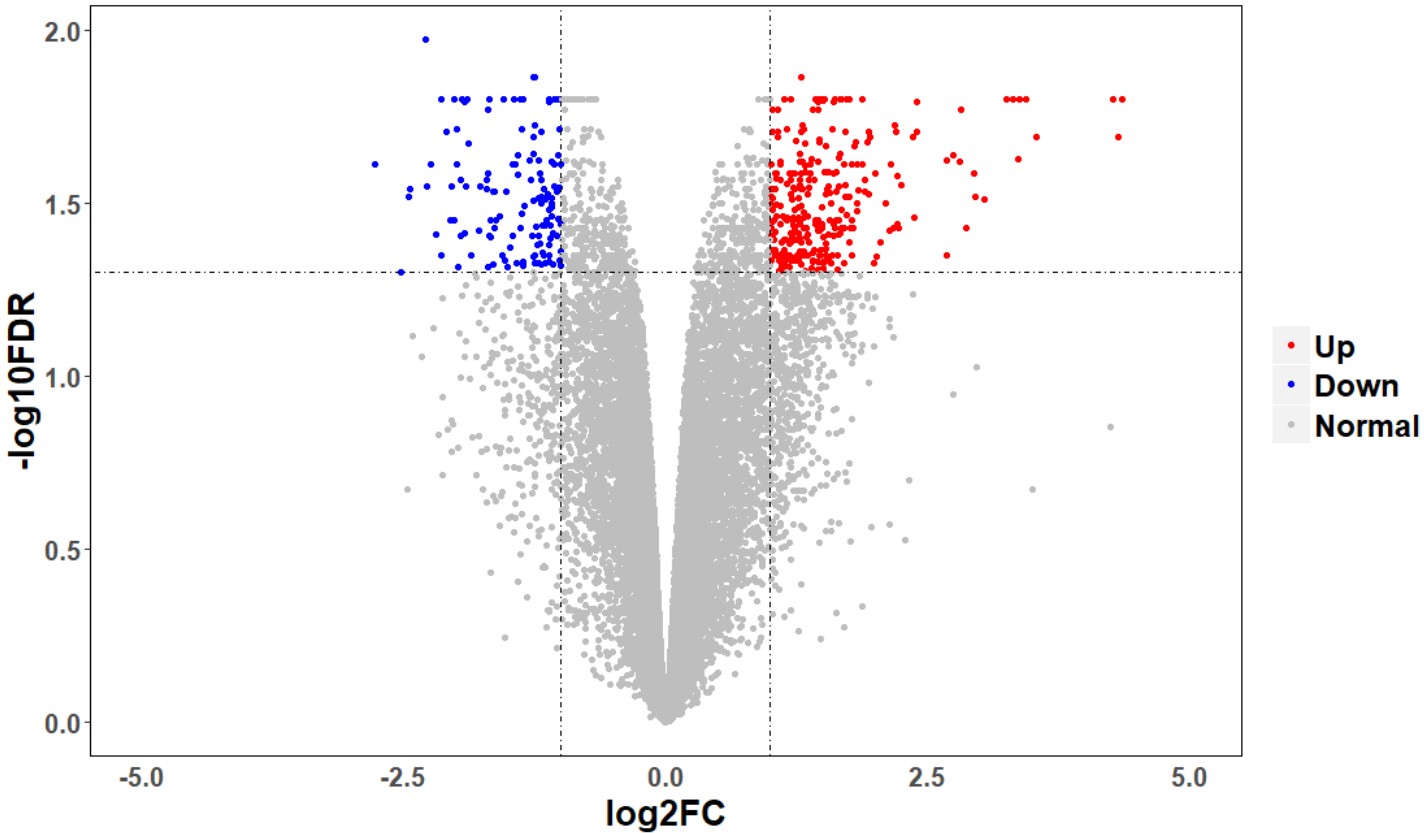
数据解释: 数据框temp1,行名为GeneID,第一列为-log10FDR,数据愈大,该基因变化为显著,第二列为logFC,实际表示log2FC,为差异变化倍数,第三列为上下调信息,基于第一列和第二列的信息的阈值进行筛选获得,需要给这一列信息进行设置,设置为有序因子,方便后续图片颜色设置
temp1$Regulate=factor(temp1$Regulate, levels=c("Up","Down","Normal"), order=T)
绘图,绘制成点图
P_volcano=ggplot(temp1,aes(x=temp1$logFC,y=temp1[,"-log10FDR"]))+
geom_point(aes(color=temp1$Regulate))+ #设置点的颜色
scale_color_manual(values =c("Up" = "red", "Down" = "blue", "Normal" = "grey"))+
labs(x="log2FC",y="-log10FDR")+ #增加阈值线:分别对应FDR=0.05,|log2FC|=1
geom_hline(yintercept=-log10(0.05),linetype=4)+
geom_vline(xintercept=c(-1,1),linetype=4)+
xlim(-5,5)+
theme(plot.title = element_text(size = 25,face = "bold", vjust = 0.5, hjust = 0.5),
legend.title = element_blank(),
legend.text = element_text(size = 18, face = "bold"),
legend.position = 'right',
legend.key.size=unit(0.8,'cm'),
axis.ticks.x=element_blank(),
axis.text.x=element_text(size = 15,face = "bold", vjust = 0.5, hjust = 0.5),
axis.text.y=element_text(size = 15,face = "bold", vjust = 0.5, hjust = 0.5),
axis.title.x = element_text(size = 20,face = "bold", vjust = 0.5, hjust = 0.5),
axis.title.y = element_text(size = 20,face = "bold", vjust = 0.5, hjust = 0.5),
panel.background = element_rect(fill = "transparent",colour = "black"),
panel.grid.minor = element_blank(),
panel.grid.major = element_blank(),
plot.background = element_rect(fill = "transparent",colour = "black"))
P_volcano
结果如下:
如果想提升自己的绘图技能,我们推荐:R语言绘图基础(ggplot2)
更多生物信息课程:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程、基因家族文献思路解读
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘、转录组文献解读
5. 微生物16S/ITS/18S分析原理及结果解读、OTU网络图绘制、cytoscape与网络图绘制课程
6. 生物信息入门到精通必修基础课:linux系统使用、perl入门到精通、perl语言高级、R语言入门、R语言画图
7. 医学相关数据挖掘课程,不用做实验也能发文章:TCGA-差异基因分析、GEO芯片数据挖掘、GEO芯片数据标准化、GSEA富集分析课程、TCGA临床数据生存分析、TCGA-转录因子分析、TCGA-ceRNA调控网络分析
8.其他,二代测序转录组数据自主分析、NCBI数据上传、二代测序数据解读
- 发表于 2018-06-15 09:59
- 阅读 ( 7965 )
- 分类:R
你可能感兴趣的文章
- 多组学联合分析—桑基图 518 浏览
- 一文教你绘制GO富集分析弦图,简单又好看 501 浏览
- R脚本绘制多层环形饼图 3302 浏览
- corrplot包绘制相关性图,简单又好看! 3438 浏览
- 已知集合交集数量绘制韦恩图 3965 浏览
- 不均等坐标系绘制 2803 浏览
相关问题
- linux 下 R 命令怎么后台运行 2 回答
- ggplot2-气泡图-geom_point几何对象函数中点的颜色/大小映射的问题 1 回答
- rJava 安装包错 1 回答
- R包安装 1 回答
- R语言绘制中国地图,每个省份出现多边形 1 回答
- R 更新后报错 0 回答
0 条评论
请先 登录 后评论
