O2PLS-DA分析
两组学数据的正交偏最小二乘法判别分析(2-way orthogonal Partial Least Square with Discriminant Analysis )
O2PLS 模型用于两个数据组间的整合分析,包括系统生物学组学间关联、分子调控机制-表型间关联等各种大数据组的内在联系都可通过此模型进行整合分析。该模型一方面可反映不同数据组间的整体影响,另一方面可直接体现不同变量在模型中的权重(权重越大,意味着该变量的变化对另一个组学的扰动更剧烈),从而更加精准地发现关键调节现象。O2PLS 为非监督建模,可客观描述两数据组间是否存在关联趋势,尽可能从源头上避免假阳性关联。由于 O2PLS 模型可灵活进行组学数据挖掘,发现不同层面的调节信息,从而有助于建立系统生物学调节网络。
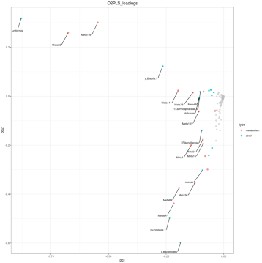
选取所有的差异基因、差异代谢物建立 O2PLS 模型,通过载荷图初步判断不同数据组中相关性和权重都比较高的变量,筛选出影响另一组学的重要变量。图中每个点到原点的距离高度代表基因或者代谢物和另外一个组学相关性的大小,图中标示出对另一个组学影响较大的前 10 个基因/代谢物。
(载荷图)
O2PLS模型关联分析需具备以下数据:
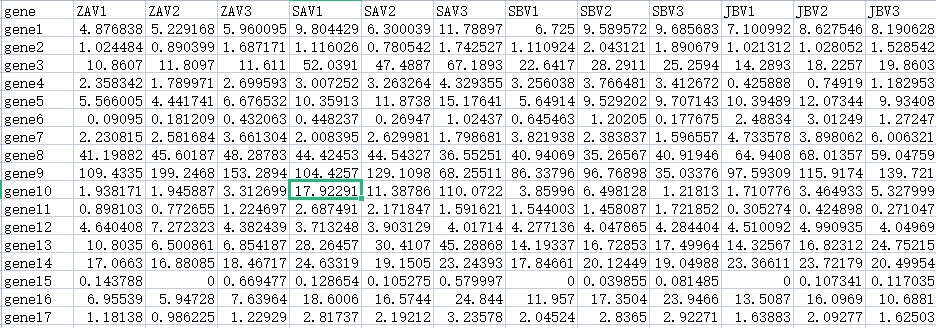
组学数据A:代谢组、转录组、菌群分布等各种定量的多元变量数据组;
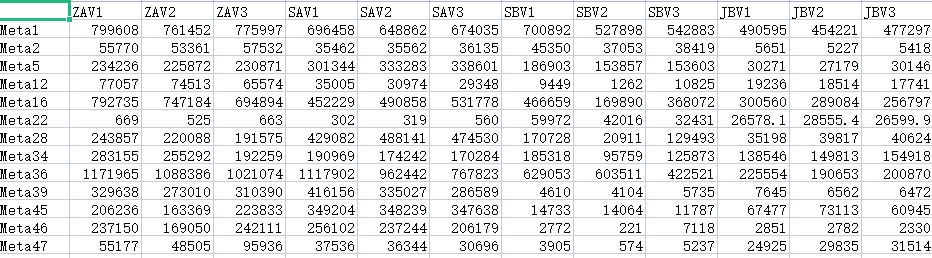
组学数据B:与A数据相同样本来源的另一类具有潜在关联性的组学数据(样本必须一一对应,如同一病人的血清样本和尿液样本、同一细胞的脂肪酸组数据和脂质组数据等)。
(例组学数据A)
 (例组学数据B)
(例组学数据B)
O2PLS模型分析可区分为两类:
A. 同一样本,不同角度研究(来自同样实验对象的血清代谢组和尿液代谢组数据,考察系统性代谢现象;或针对正常和疾病人群血清样本,进行代谢组和转录组考察):
通过是否可建立O2PLS模型,判断数据组间是否存在关联性;
通过得分图判断结合两组数据是否可反映样本宏观特征(聚类、分组);
通过载荷图判断不同数据组中相关性和权重都比较高的变量。
B. 通过两数据组在不同条件下的调节是否可建立O2PLS模型,判断数据组在不同条件下的调节间是否存在关联性:
对不同实验条件下的各组样本分别建立O2PLS模型;
考察不同组间的SUS2图(整合两个O2PLS模型的载荷图),发现各组间随处理条件变化而改变的变量(代谢物、基因、菌群等指标)。
判别分析法(discriminant analysis)
在已知的分类之下,一旦遇到有新的样本时,可以利用此法选定一判别标准,以判定如何该将新样本放置于那个族群中
- 发表于 2022-12-19 14:20
- 阅读 ( 4242 )
- 分类:其他
