基因家族分析课程更新-批量进行kaks计算
在我们的基因家族视频课程中,演示的计算kaks方法为手动计算,当我们要计算的复制基因对太多时,手动计算就太费时间了,这里我们更新了课程,可以批量提供基因对列表(串联重复和大片段复制),就可以快速批量的进行kaks计算,其中用到的重要工具为Para AT,文献如下:

ParaAT工具介绍
ParaAT是中科院基因组所的张章教授课题组开发的工具,该工具可以批量并行的做基因对的比对,并把比对的结果转换成KaKs_Calculator需要的axt格式文件,对于大批量的基因对之间的kaks计算十分有用。
蛋白序列比对(可选比对软件 clustalw2 | t_coffee | mafft | muscle)。根据蛋白比对结果回译成codon对应的核酸比对结果(Back-translated nucleotide alignments guided by amino acid alignments are more reliable and accurate than direct nucleotide alignments)。该方法尤其对于序列差异较大的基因对,比对更准确,可以避免由于比对问题导致KaKs_Calculator计算不出结果。
下载安装和数据准备
ParaAT2.0下载
ParaAT下载地址为:https://ngdc.cncb.ac.cn/tools/paraat
“ParaAT.pl”是运行的脚本,下载解压后就可以直接使用。可以把解压后的路径加入环境变量,或者用脚本所在的绝对路径来运行。
输入文件准备:
复制基因对列表,每一行位一对复制基因,中间用tab分割开即可test.homologs (软件自带的测试数据)
NP_000005 NP_783327
NP_000006 NP_032699
NP_000008 NP_031409
NP_000009 NP_059062
NP_000010 NP_659033
NP_000012 NP_032969
NP_000013 NP_031424
NP_000014 NP_033187
NP_000015 NP_031446
NP_000016 NP_038490
对应基因的蛋白质序列和cds序列文件,fasta 格式:test.cds;test.pep
运行命令
ParaAT.pl -h test.homologs -n test.cds -a test.pep -p proc -m muscle -f axt -g -k -o result_dir #proc文件必须与输出位置在同一个目录下,不然会报错
#合并文件
cat result_dir/*axt >all_aln.axt
#kaks 计算
KaKs_Calculator -i all_aln.axt -o all_kaks_results.txt -m YN
参数说明:
-h, 指定同源基因列表文件
-n, 指定核酸序列文件
-a, 指定蛋白序列文件
-p, 指定多线程文件
-m, 指定比对工具
-g, 去除比对有gap的密码子
-k, 用KaKs_Calculator 计算kaks值
-o, 输出结果的目录
-f, 输出比对文件的格式
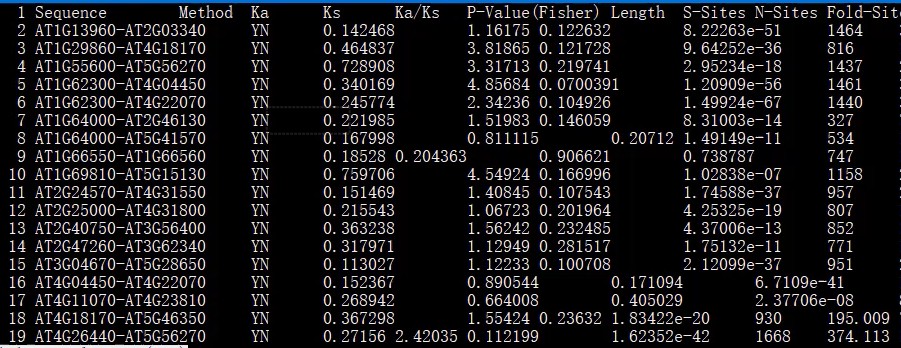
结果展示:
基因家族课程更新:
已经更新了这部分内容:https://zzw.xet.tech/s/1BAqPp
或者扫码观看:
Reference
Zhang, Z., Xiao, J., Wu, J., Zhang, H., Liu, G., Wang, X. and Dai, L. (2012) ParaAT: A parallel tool for constructing multiple protein-coding DNA alignments, Biochem Biophys Res Commun, 419(4):779-7
- 发表于 2023-01-07 18:24
- 阅读 ( 5219 )
- 分类:基因家族分析
你可能感兴趣的文章
- 计算Kaks时批量提取多对基因的序列 4996 浏览
- kaks和dnds是什么? 25568 浏览
- Ka/Ks与进化选择压力 10291 浏览
- 计算串联重复基因KaKs的时候,aln文件一定要通过脚本转换为axt文件么?这两种文件格式是否可以直接通过修改扩展名来实现? 4859 浏览
- Ka/Ks与进化选择压力 15543 浏览
