叶绿体基因组四个区边界作图——irscope:本地化及使用教程(2023.02)
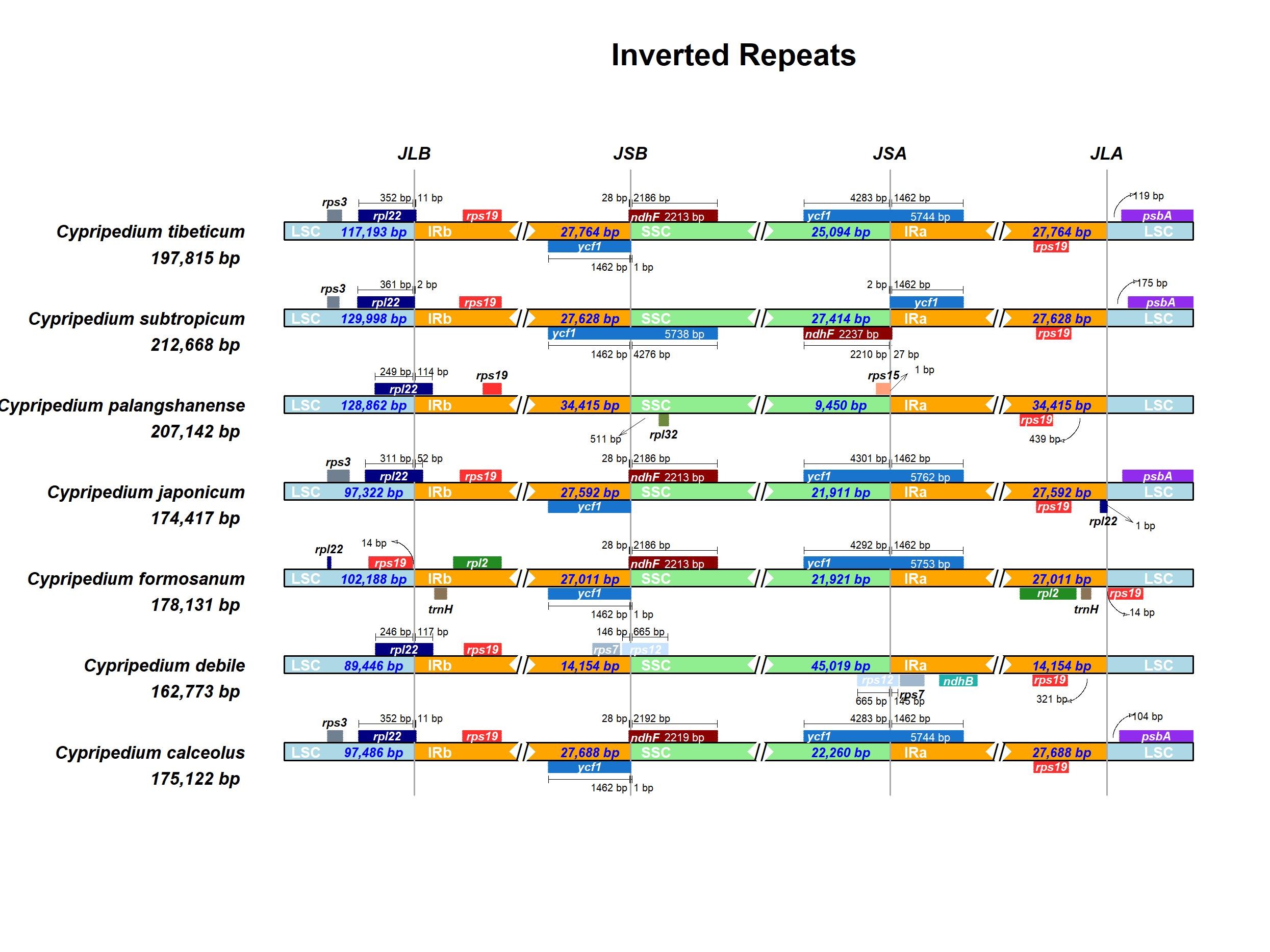
大部分植物的叶绿体基因组具有典型的四分体结构,即由LSC、SSC以及两个IR区域构成,IR和SSC区域的收缩与扩张是常见的进化事件。通过IRscope工具可以将多个物种叶绿体的区域边界进行可视化对比,从而对该科/属的叶绿体的结构有一个更加宏观的认知。
一、获取工具
irscope是一个在线工具,网址为:https://irscope.shinyapps.io/irapp/
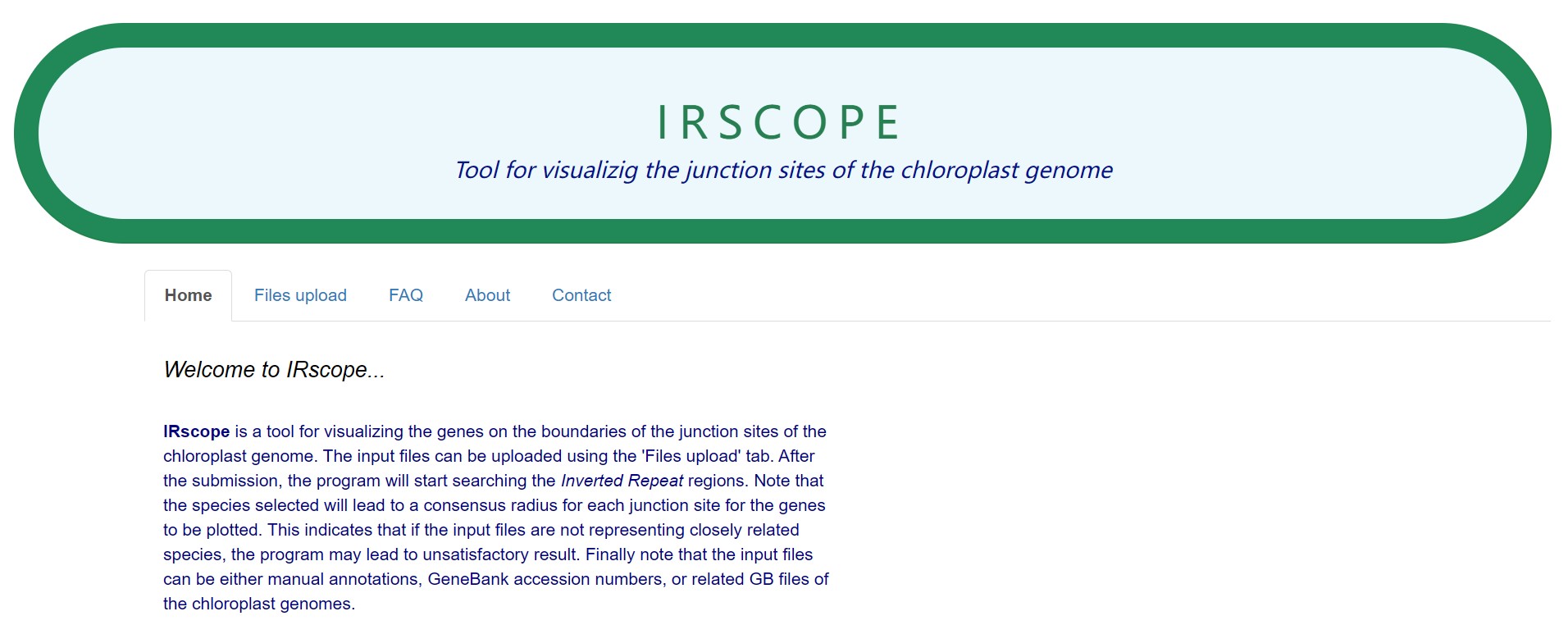
但是在线工具很不稳定,会经常会出现进网站很慢,或者运行时总是出错的情况,比如这样
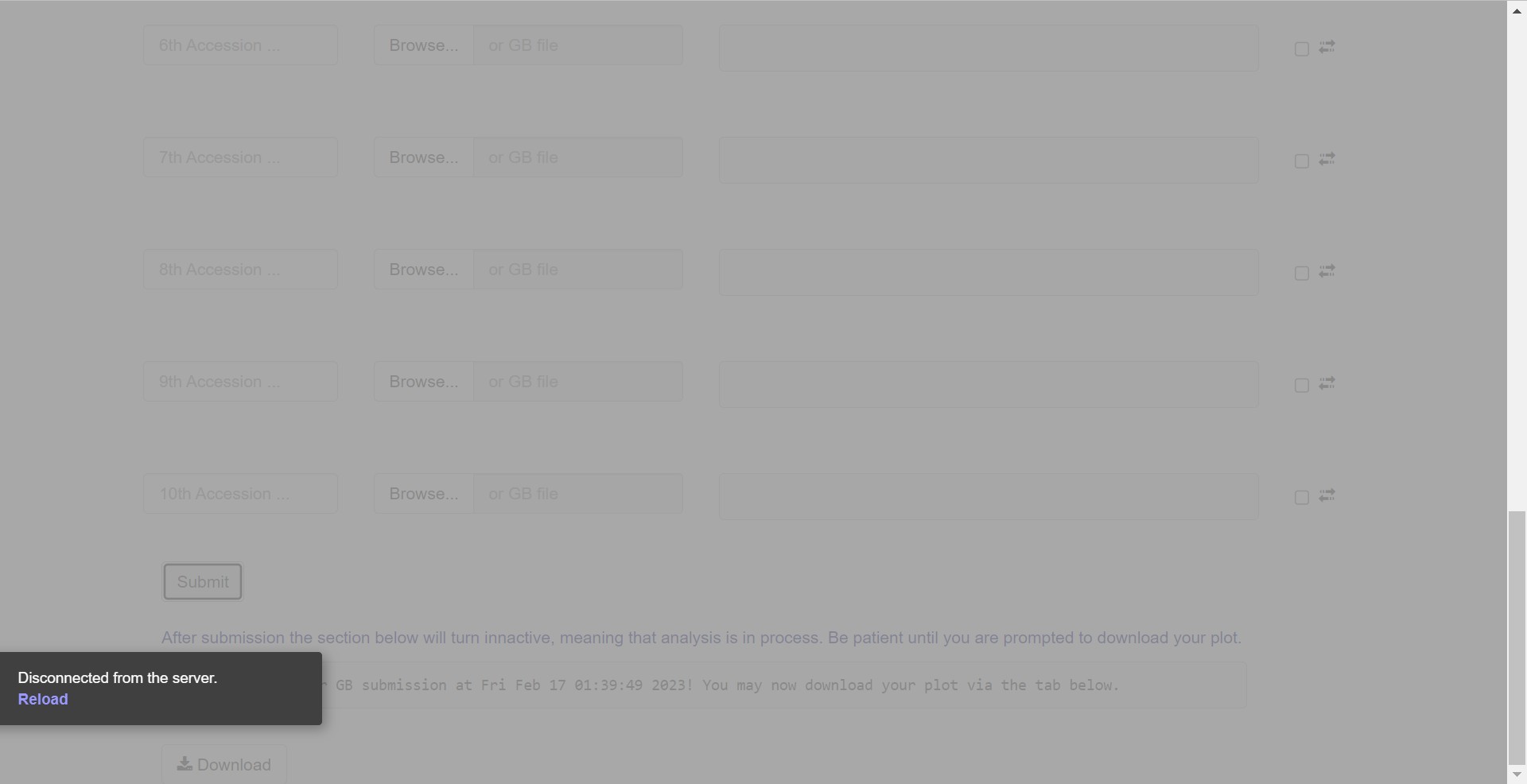
所以我们可以利用R语言将irscope本地化,生成本地网站,运行就不会卡了。
二、利用R语言将IRscope本地化
所需软件:R及Rstudio,网上有很多详细的安装教程,在此就不赘述了。
可能由于网站更新等缘故,本地化过程稍有不同,此记录为2023年2月进行IRscope本地化的过程。
1.下载IRscope源代码
首先进入github:https://github.com/AmiryousefiLab/IRscope
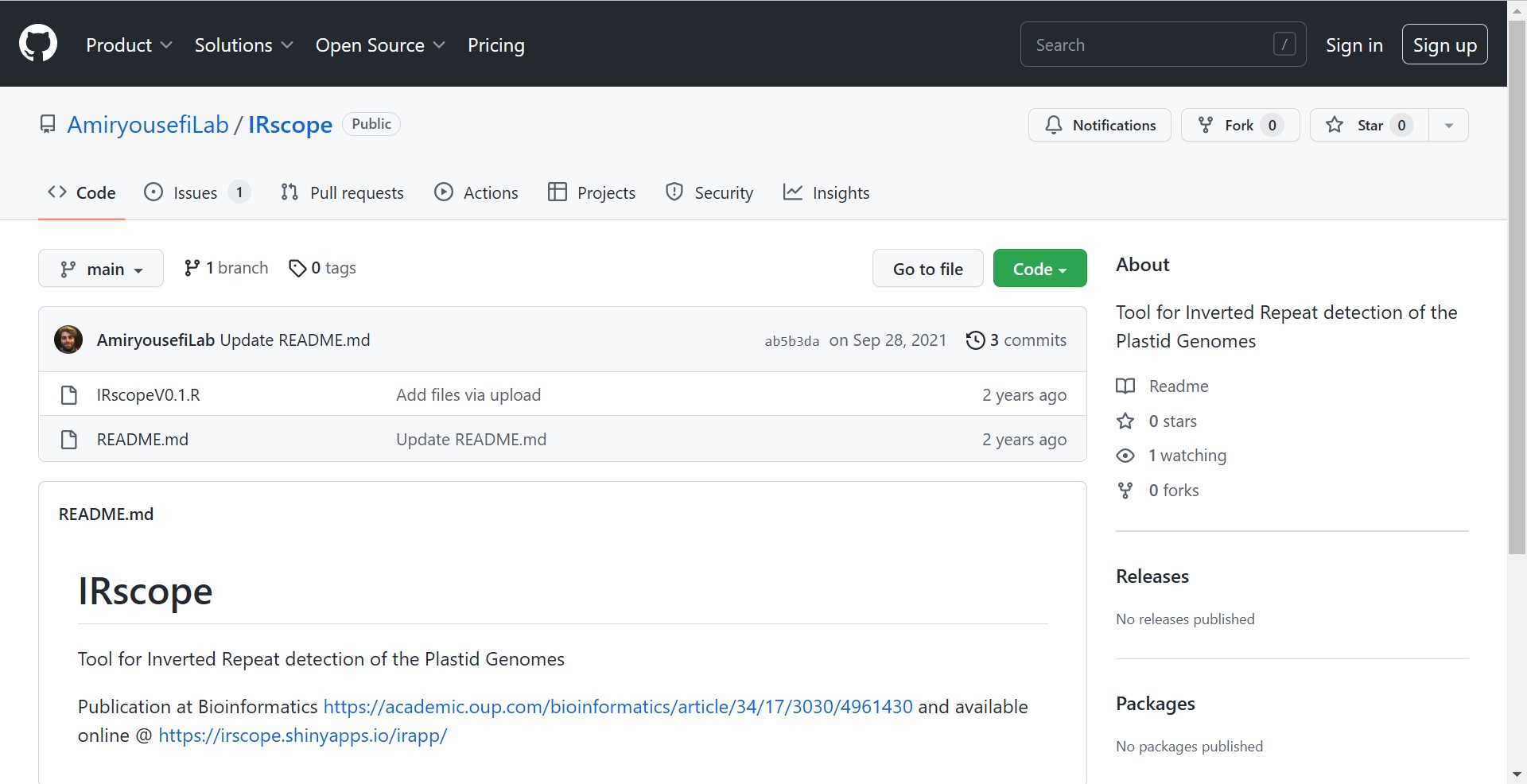
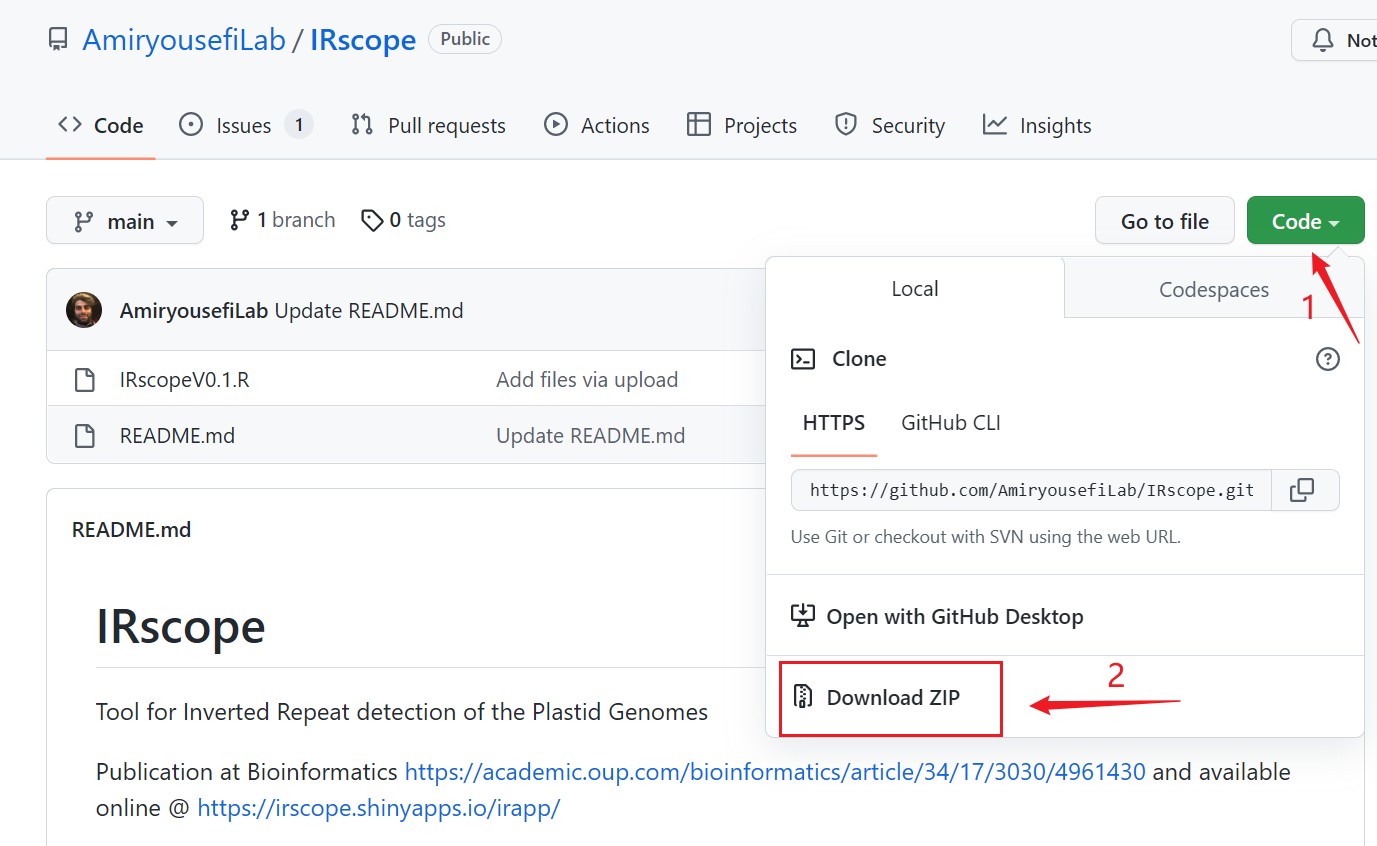
点击code → Download ZIP,即可下载原代码,为方便大家下载,我把获取到的代码压缩包放在这里了→ IRscope-main.zip
下载解压后,目录下有两个文件,IRscopeV0.1.R 是我们接下来要用到的文件。
2.使用Rstudio进行IRscope本地化
打开Rstudio,点击File→Open File,打开IRscopeV0.1.R文件
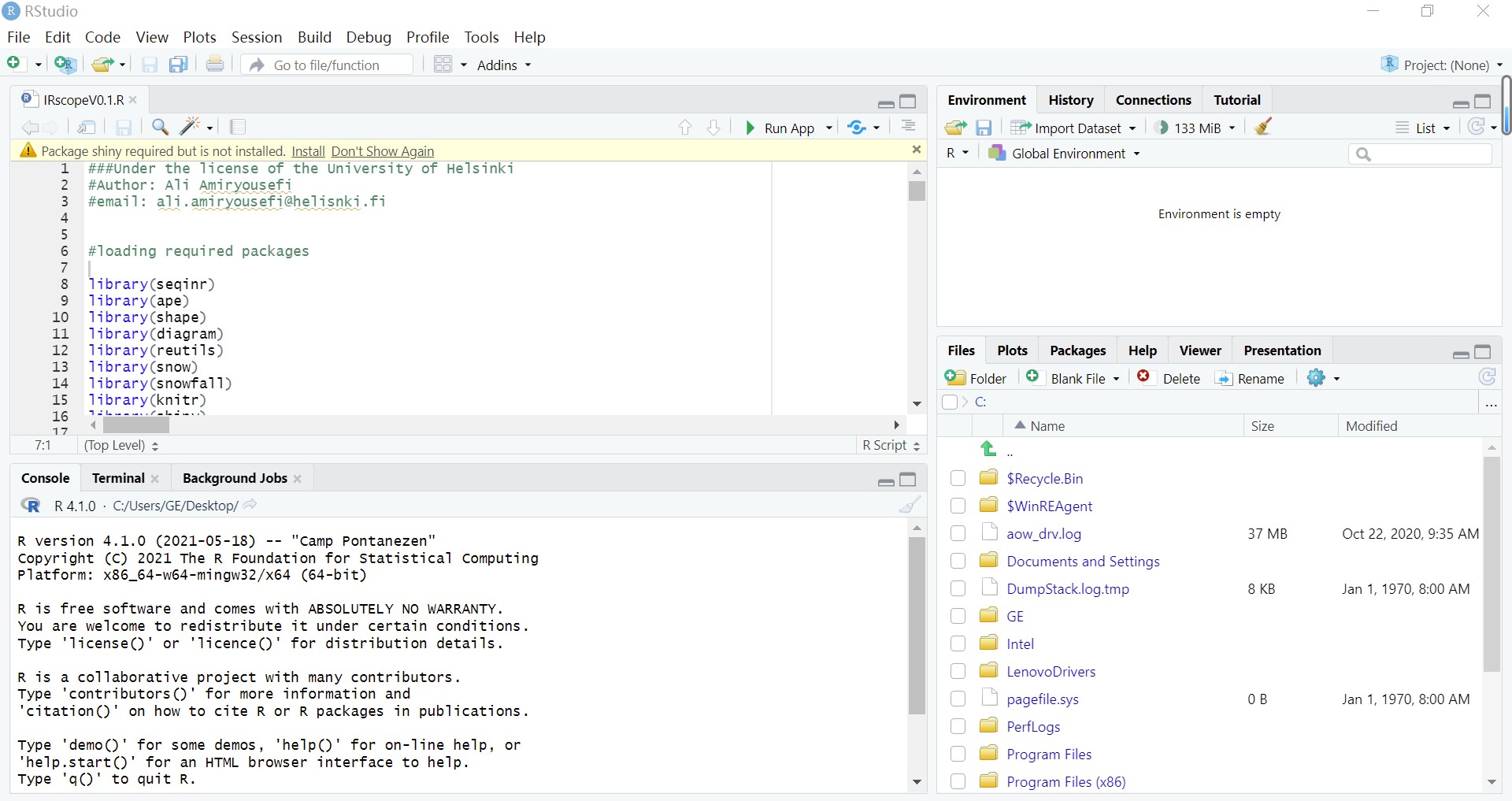
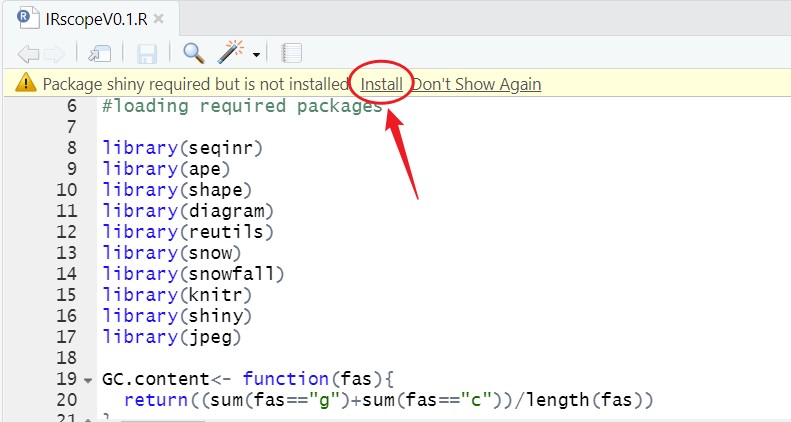
如果之前没有安装过随需要的安装包,需要先进行安装,有两种方法:一是Rstudio会自动识别我们没有安装的包,直接点击图中的Install,会自动安装;二是使用安装命令自己安装,命令为install.packages("包的名字"),例如install.packages("seqinr")。
如果不知道自己是否安装了对应的安装包,可以将光标置于library+对应安装包处,按ctrl+enter检查是否有该安装包
点击run app,下面开始运行
会出现以下报错信息
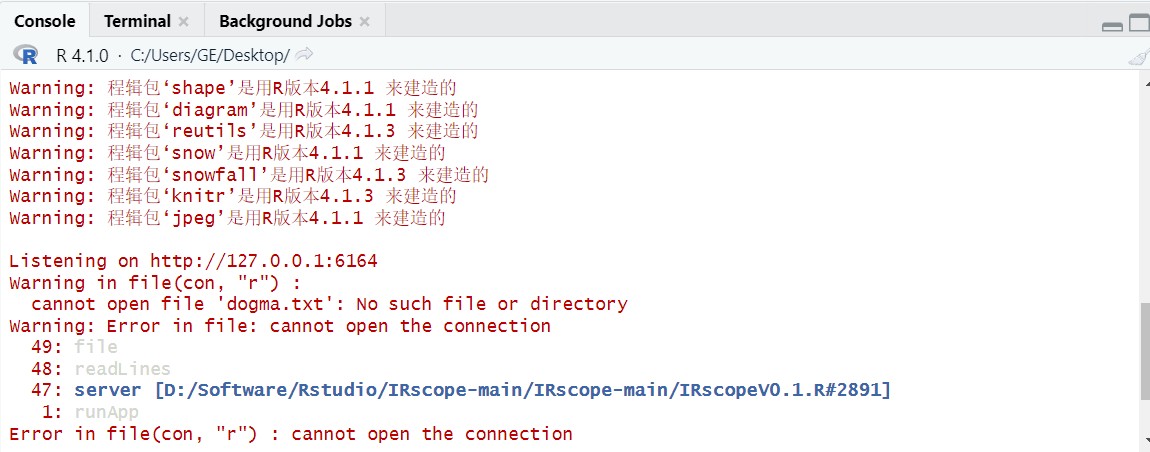
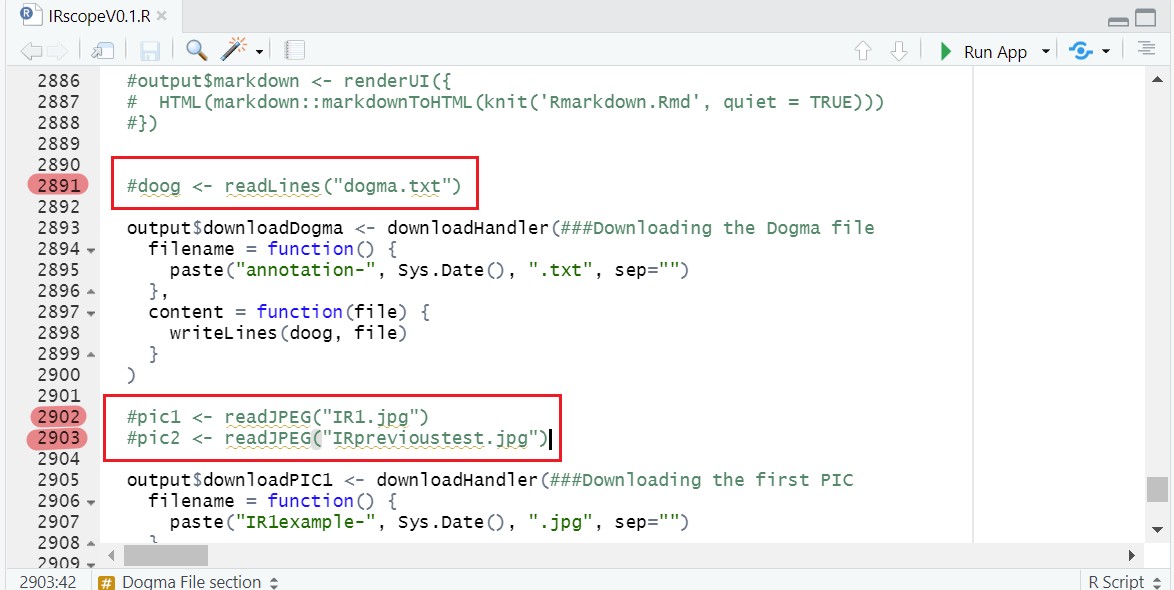
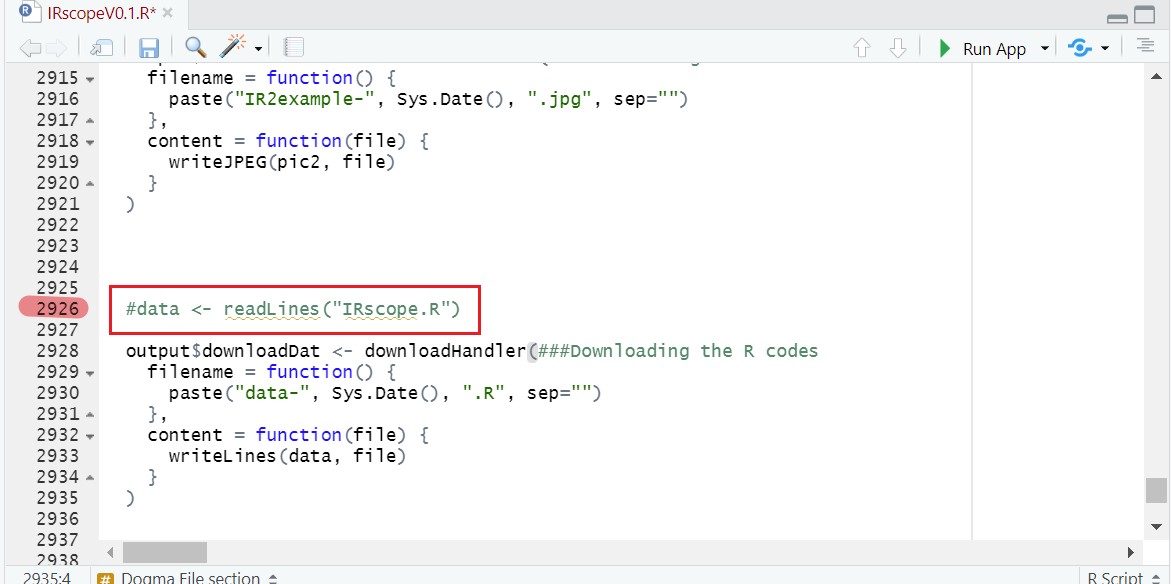
在以下几行变量前输入“#”注释掉,第2891行的doog变量,第2902-2903行的pic1和pic2,第2926行的data(具体行数可能稍有偏差,找到正确的变量即可)
运行结束后弹出IRscope界面,就可以开始使用啦~
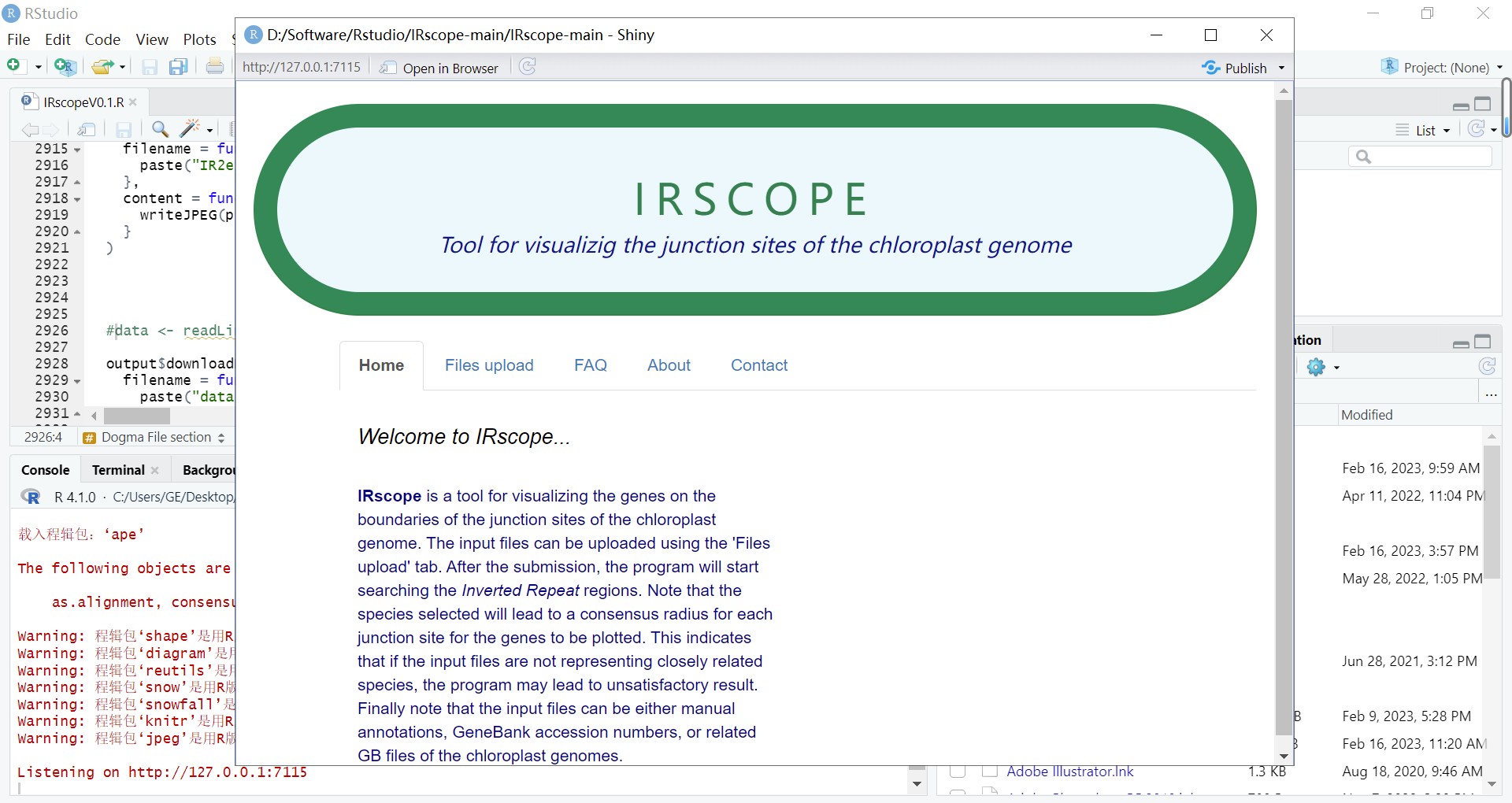
如果弹出页面是灰色的,点击Open in Browser即可在浏览器中打开

出现IRscope界面就可以使用了
三、irscope的使用
以本地化软件为例,展示该软件的使用
点击files upload
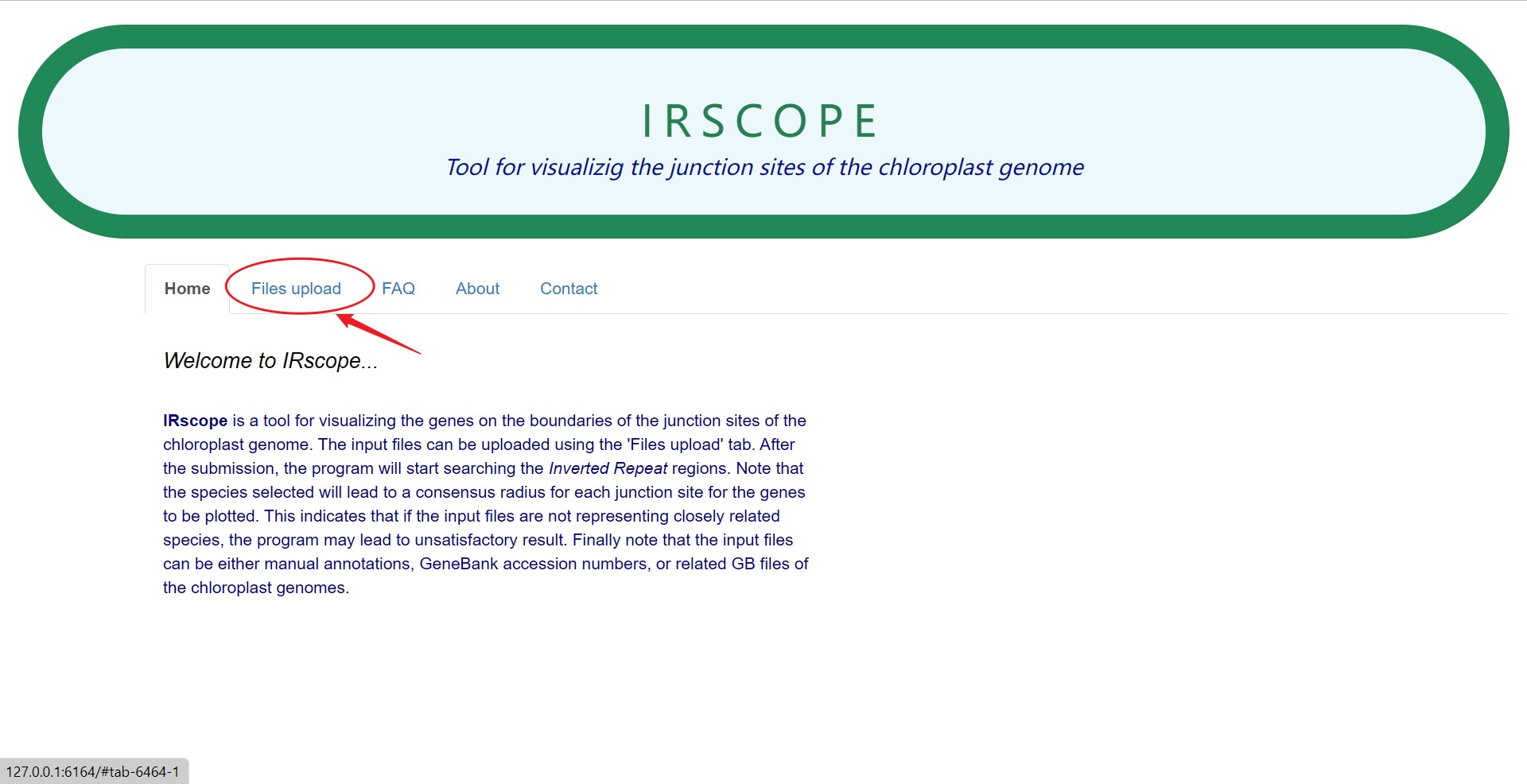
上传文件有两种方式。一是输入accession number,IRscope会自动在ncbi上下载对应物种的文件;二是自己直接上传目标物种的gb文件,一次最多上传10个。
第一种方法速度特别慢,总是报错,建议大家直接自行上传gb文件。
ssc选项是可以将ssc区反向,适用于组装时将ssc区方向反了的,一般不选。
点击Browse,上传gb文件
此处使用了文献中的7个杓兰属的物种的gb文件,文献链接为: https://doi.org/10.3389/fpls.2022.911702
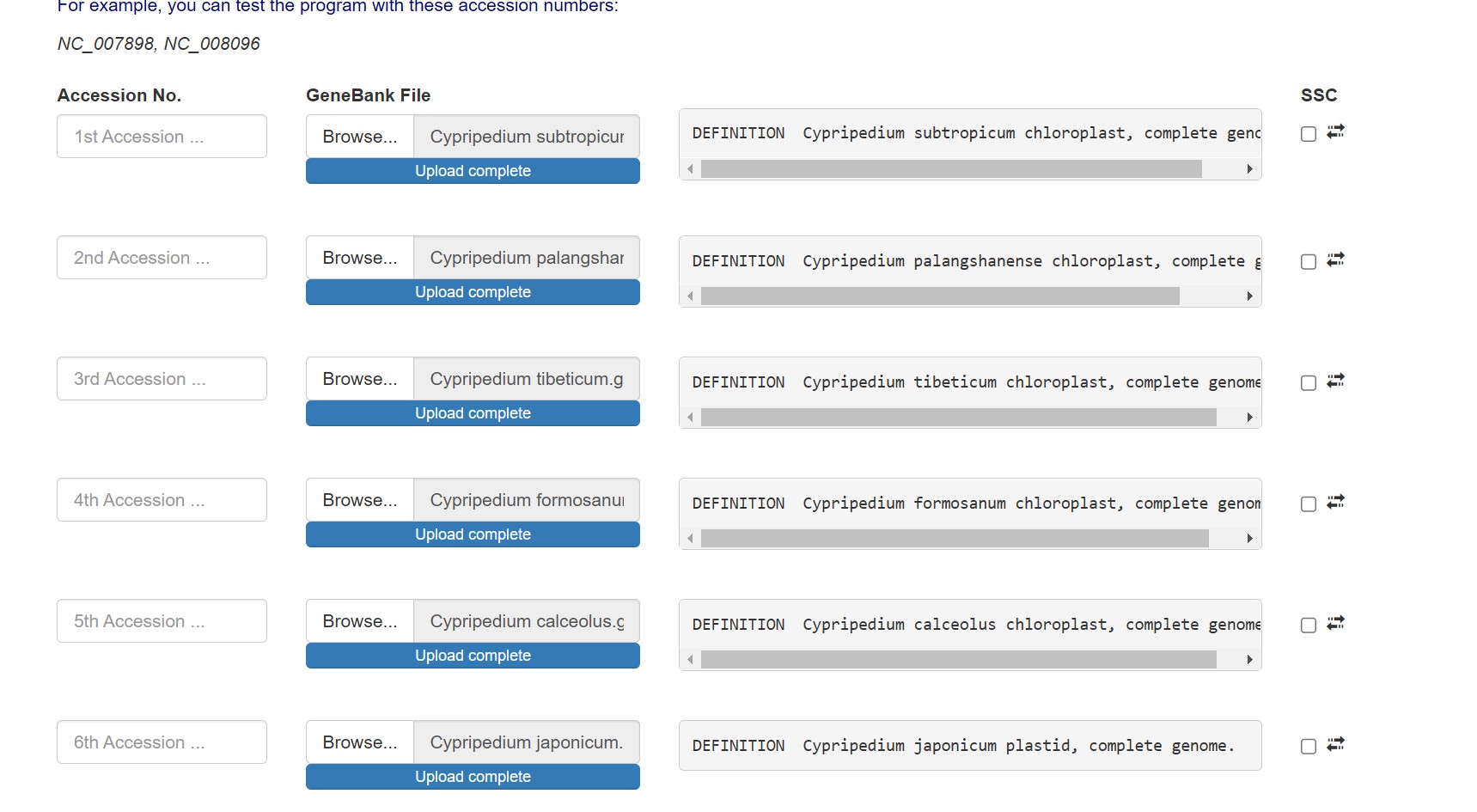
点击submit提交
耐心等待,这时候Rstudio会显示进度,上传了几条序列,就会提示几次“Stopping cluster”,直到所有gb文件运行完。
回到网站,出现这样一行字提示之后,就可以下载了,这时候点击download
下载后打开文件就可以得到这样的图啦
参考:https://www.bilibili.com/video/BV1gV411k7Bs/?from=search&seid=5741158078848451253&spm_id_from=333.337.0.0&vd_source=ccfe6b6e7b1b5b1a790a358d48c6d36e
https://blog.csdn.net/weixin_62735625/article/details/122869720
- 发表于 2023-02-15 15:53
- 阅读 ( 5061 )
- 分类:软件工具
你可能感兴趣的文章
- 叶绿体或线粒体基因组重复序列分类及鉴定 5520 浏览
- 叶绿体/线粒体基因组的串联重复序列鉴定——TRF 2991 浏览
- 《叶绿体基因组分析实操》课程更新 2311 浏览
- 叶绿体/线粒体散在重复序列注释——REPuter 5488 浏览
- NCBI下载物种叶绿体或线粒体基因组fasta,gff3,gtf,gb文件等 10796 浏览
