全基因组加倍/复制介绍(whole genome duplication, WGD)
什么是全基因组复制?
全基因加倍/复制(whole genome duplication, WGD)或者多倍化(polyploidy)事件是指某物种通过染色体倍增的方式导致多套染色体共存于同一细胞核中,形成稳定遗传的新物种的现象。根据多倍化后染色体的演化状态,可将多倍体分为两类:古多倍体(paleo-polyploids)和新多倍体(neo-polyploids)。古多倍体物种是指经历多倍化事件的多倍体祖先,通过多组亲本染色体组的改组、丢失和重排而重新二倍化,本身以二倍体的形式存在。新多倍体是指在多倍化后仍保留父母本的染色体组的多倍体物种。多倍化不仅普遍存在于高等植物中,而且还广泛存在于低等植物,例如裸子植物,蕨类和硅藻中。与植物相比,动物的多倍化现象相对稀少,但仍有上百个已知的昆虫和脊椎动物为多倍体,且多倍体化的脊椎动物主要为两栖动物、鱼类和昆虫。全基因组复制使基因组内的所有序列都发生重复,重复为生物进化提供了原始的遗传材料,使物种基因组快速重组,丢失大量基因,增加结构变异,促进生物的多样化和物种形成,给予了物种进化新的可能性。例如,WGDs促使形成了>25,000种鱼类、>350,000种开花植物。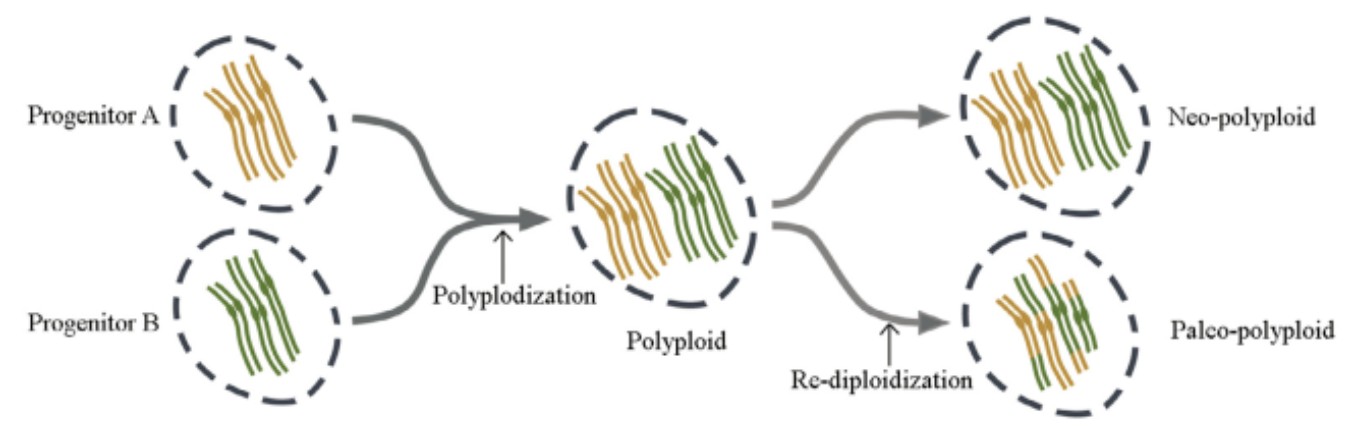
植物中的全基因组复制
多倍体植物广泛存在于自然界中,如日常生活中的马铃薯、小麦、棉花等。研究发现多倍化在有花植物进化过程中十分频繁,在现存的被子植物和种子植物分化之前,都分别发生过加倍事件,可能对花和种子的产生有重要贡献 。随着测序技术的飞速发展,越来越多植物基因组被组装出来,许多多倍化事件也被鉴定出来,基因组多倍化已成为植物学研究领域中的热点。例如:在 被子植物和种子植物 开始大规模分化前,其祖先各发生了一次 古多倍化 事件;在 单子叶和双子叶植物 中,其分别经历了 早期的多倍化:Gamma(γ)三倍化以及Tau(τ)事件 ;在 被子植物 中,那些物种丰富的科都有过一样的故事:如菊科、十字花科、葫芦科、豆科、禾本科、以及兰科等。拟南芥发生了三次多倍话事件:three rounds of WGDs (γ-β-α)葡萄在发生了γ事件之后再没有发生多倍化,长用来作为参考物种。不同证据表明,全基因组复制事件发生于植物演化的不同时期。但巧合的是,有大量的重复事件发生于白垩纪-第三纪大灭绝事件(Cretaceous–Paleogene,K-Pg),也被称为K-Pg线,即传闻让所有恐龙不复存在的那次生物大灭绝事件。这也暗示着,全基因组复制事件可能帮助物种适应当时恶劣的气候环境,使得他们于灭绝事件中存活下来。但对于此,一切都仅仅是猜测,尚未有明确的遗传学证据证明。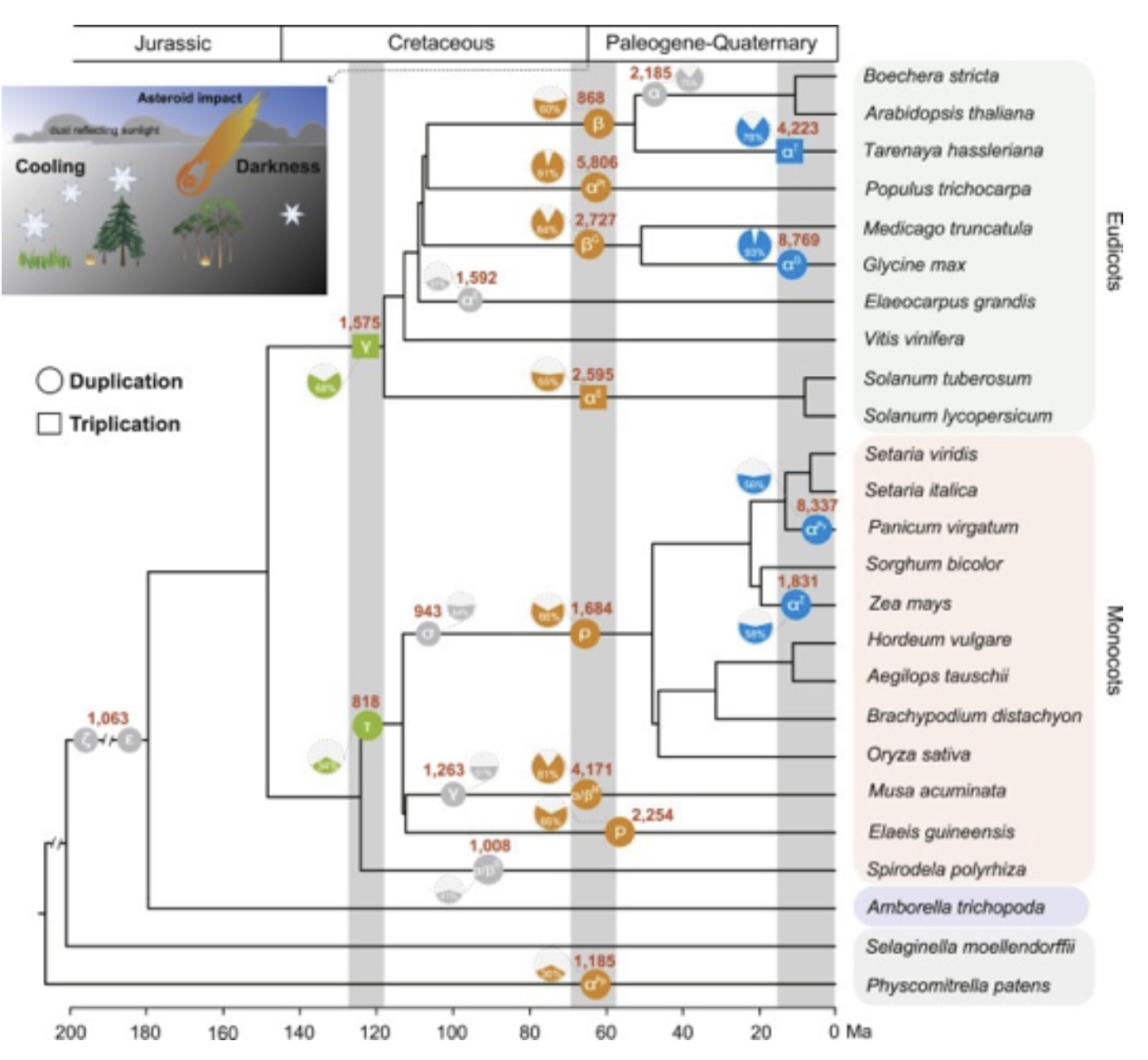
如何鉴定全基因组复制?
鉴定全基因组复制的方法一般可以通过以下两种:
(1)共线性分析
可以通过基因(基因组)的共线性(synteny)进行识别,方法比较直观。其方法是全基因组范围比较两个物种的基因(基因组)的序列,并将同源序列的位置绘制成点状图,如果能在点状图中发现比较明显的长片段,并且这样的长片段比较多,便可以推测是由于大尺度的全基因组复制以后保留下来的痕迹,也就是全基因组发生了复制。同样,对于单个物种而言,我们也可以绘制基因组内部的共线性的点状图,如果发现同一个物种的基因(基因组)的区间可以匹配到多个不同的区间中,这就暗示了该物种经历过基因组的加倍事件。但对于经历过多次全基因组加倍事件的物种来说,后来的加倍事件会加速上一次加倍事件的基因丢失,造成上一次加倍事件的痕迹越来越不明显,这也给共线性分析带来干扰。基因组共线性是基因组加倍比较直接的证据,通过比较两个基因组的序列并将共线性的区域作图展示,可以直观发现全基因组加倍的痕迹。如下图(左)马尾树的基因组circos图中,可以明显染色体间大片段的共线性,表明该物种近期发生了全基因组复制。在一串红基因组中,通过基因组自身的比对展示如下图(右),对角线为物种自身的基因和其本身的共线性。其余的点为基因组其他位置的旁系同源基因对。图中绿色圆圈标注的位置,及其他连续点的位置为基因组加倍事件留下的痕迹。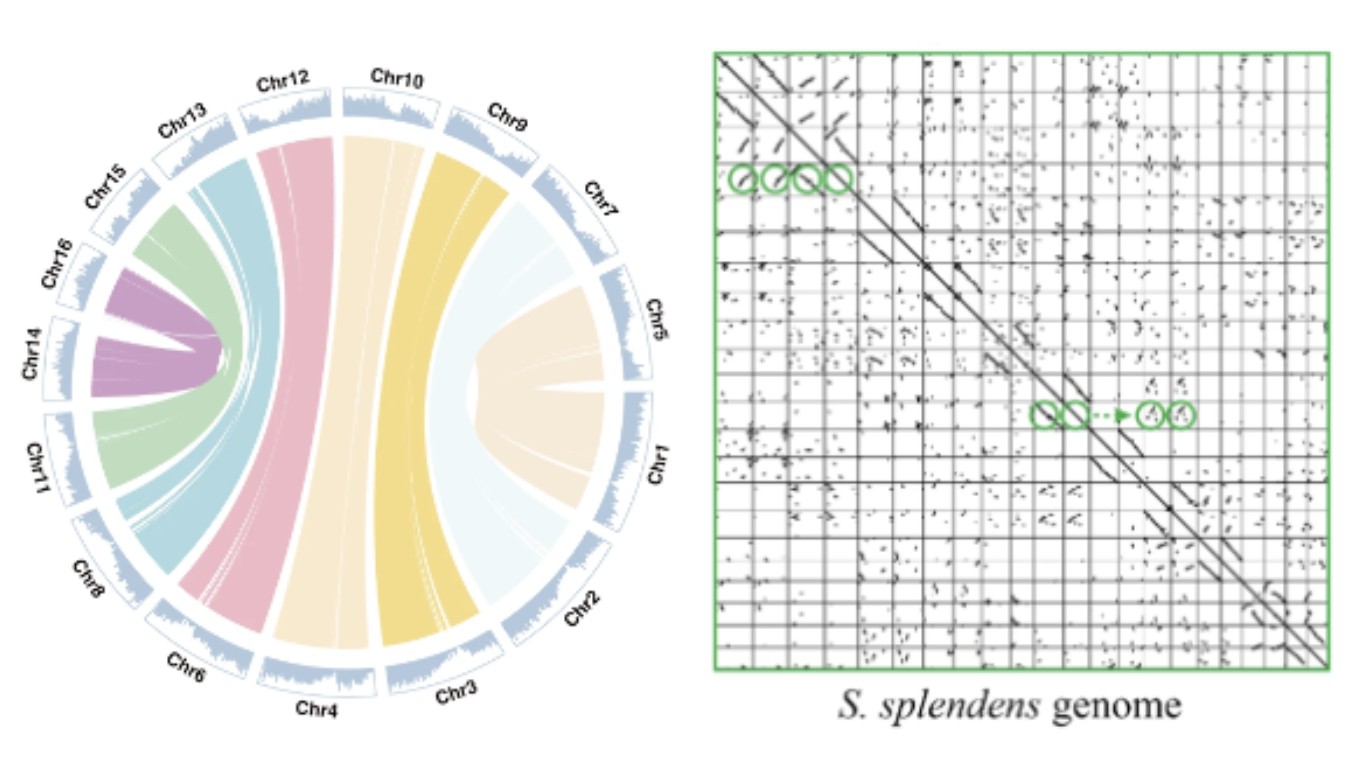
如果物种经历过多次全基因组加倍事件,近期的加倍事件会加速早期加倍事件的基因丢失,早期的加倍事件痕迹往往越来越不明显,共线性直观上不明显,这就需要我们探索其他方式来挖掘加倍事件,这就用到了4DTv和Ks的信息。下面我们对这两种方式来进行简单的介绍。
(2)同义突变率 Ks 方法/4dTV 法
同义突变指突变并不影响氨基酸序列,进而不会影响蛋白结构与功能,Ks值表示发生非同义替换的位点数量/非同义替换位点数。假设这个基因没有受到自然选择压力,那么根据中性选择理论,密码子同义位点的变化是完全随机的,并随时间推移累积。如果物种发生了全基因组加倍事件,会产生大量的同源基因会产生大量的同源基因,这些基因在进化过程中积累的同义突变接近,计算得到的Ks值也接近,因此在某一个Ks值处会形成一个峰(ks peak)。如果这处Ks值的基因数目足够多,就会形成比较尖的峰值,可以认为在进化过程中该处发生过全基因组加倍事件。要进行 Ks 分析,首先要找到同源基因对,在不同的物种里面(比如葡萄-拟南芥),是找最近的直系同源基因(ortholog),而在一个物种内部(比如葡萄-拟葡萄),则是找最近的旁系同源基因(paralog)。通过计算这些基因的 Ks 值,我们就可以绘制出不同 Ks 值对应的基因对数量的分布图。旁系同源基因对的 Ks 分布峰值对应全基因组复制事件,直系同源基因对的峰值对应物种的分化事件,借助于物种分化事件对应的时间,可以推出全基因组复制事件发生的时间。例如,在一串红(Salvia splendens)基因组文章中,选取了柚木(Tectona grandis )、葡萄(grape)来做ks分析。结果如下图,从Ks峰图和进化树可以看出:(1)葡萄和一串红在Ks值约 1.6 处核心双子叶植物共有的全基因组三倍化事件;
(2)柚木在Ks值约Ks = 0.6–0.8 (WGD-3)处发生了单独的全基因组复制。
(3)一串红分别在Ks值0.08 (WGD-1) 和 0.26 (WGD-2)处发生了两次近期全基因组复制。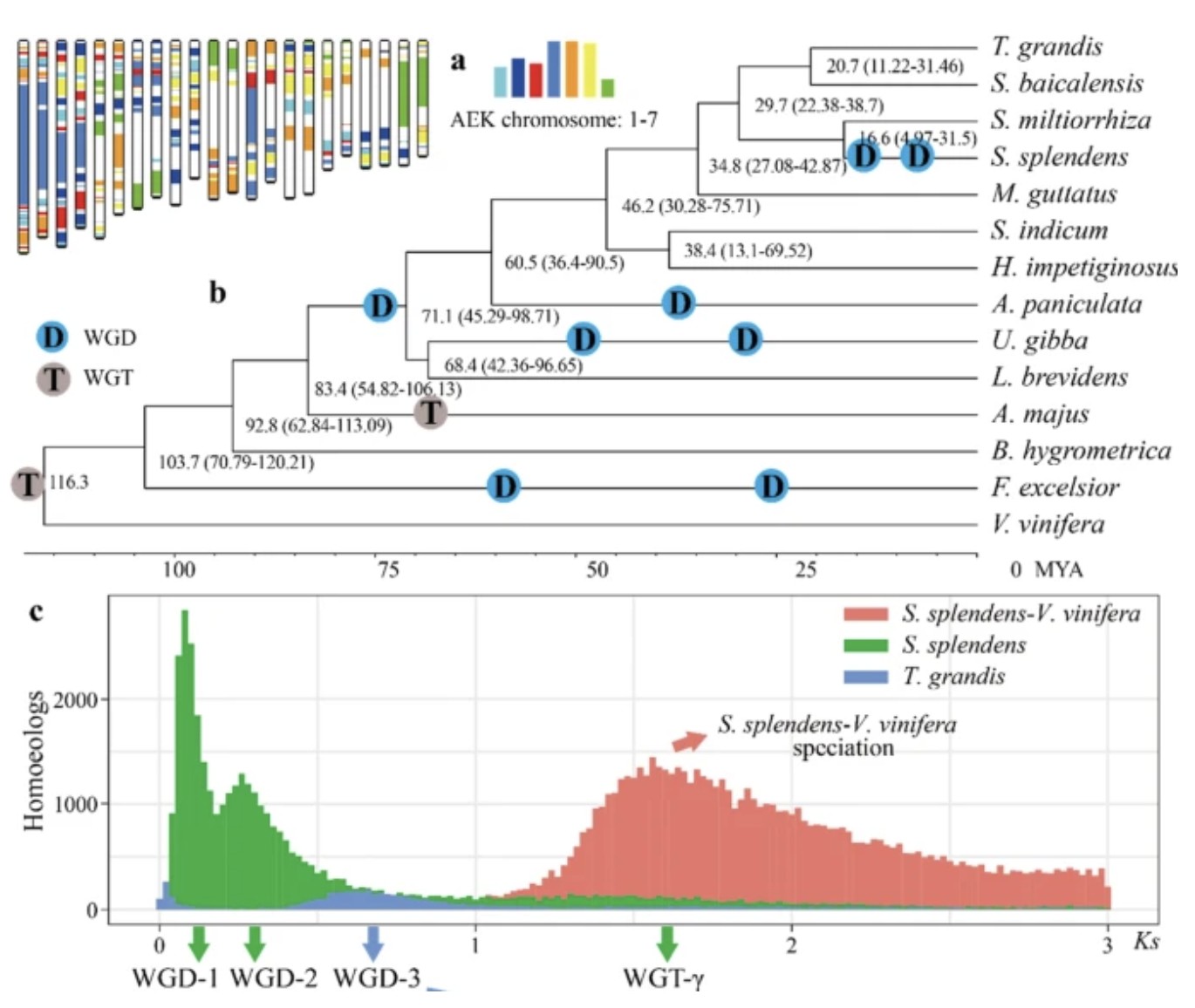
当然 Ks 值也有一些不可避免的限制,比如很难应用于比较古老的基因组加倍事件的识别,这是因为随着时间的推移,基因丢失越多,发生的变化也要越大,形成的Ks峰越扁平,对于寻找古老的基因组加倍事件造成困难。
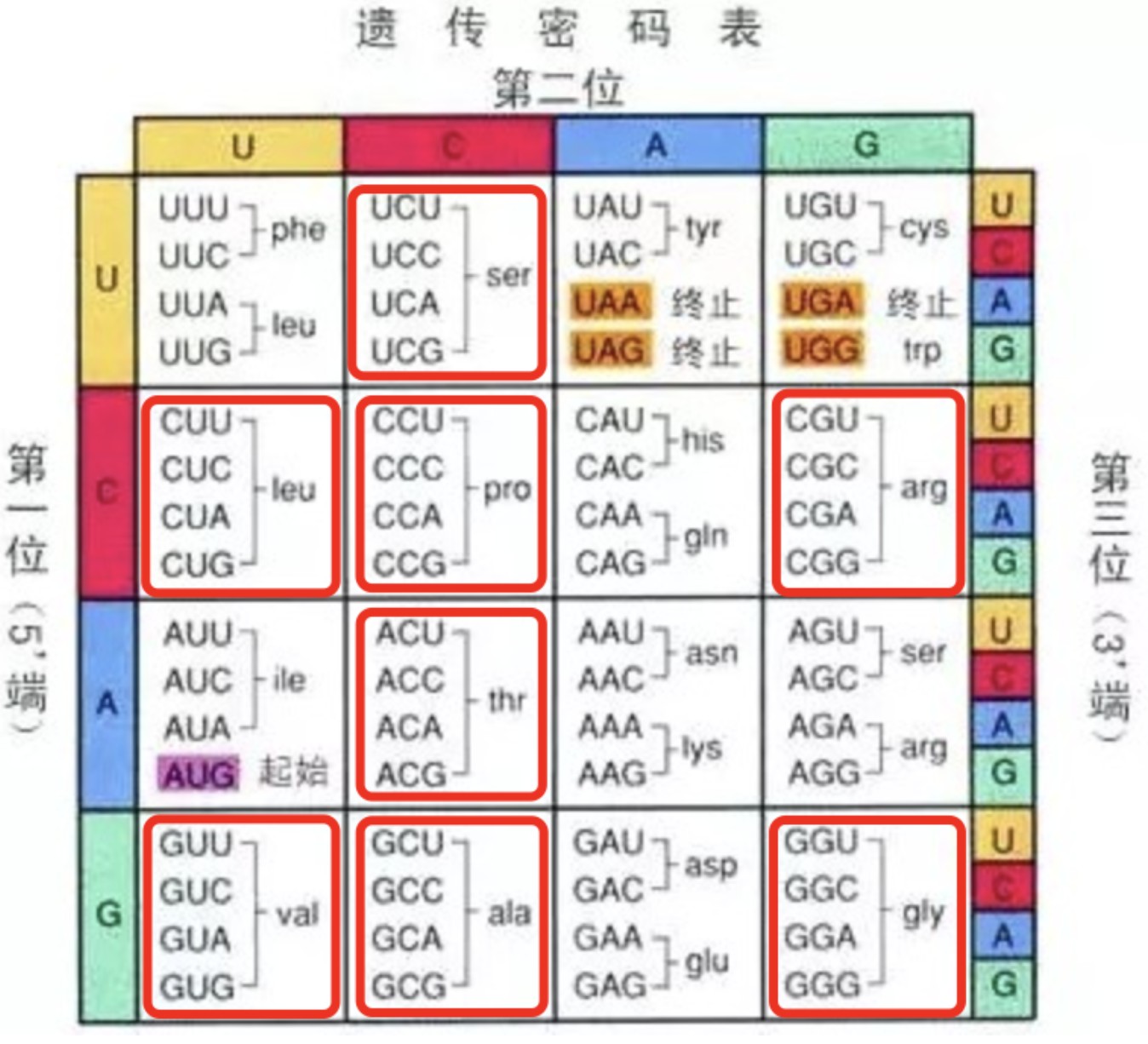
4DTv与Ks有异曲同工之处。4DTV( four-fold synonymous (degenerative) third-codon transversion) 表示4D位点(fourfold degenerate site)上发生颠换(嘌呤突变为嘧啶或者嘧啶突变为嘌呤)的位点替换率。什么事4D位点呢? 一个遗传密码子通常由三个核苷酸构成,从左到右依次为第一个位点、第二个位点、第三个位点。如果密码子的某个位点上无论是哪种核苷酸,均编码同样的氨基酸,则称这个位点为 4 倍简并位点。例如甘氨酸密码子(GGA, GGG, GGC, GGU)的第三个位点就是一个 4 倍简并位点。按照密码子表,目前只有某些密码子的第三个位点才可能是 4 倍简并位点。
例如,中国本土罂粟(CHM)(Papaver somniferum,Pso)基因组文章中用 到4DTv 分析, 同时引入其他物种做比较,包括:假泽兰 M. cordata (Mco), 和 毛茛 A. coerulea (Aco). 通过4DTv值分布发现, M. cordata, P. somniferum, and A. coerulea 的旁系同源基因的4DTv峰值几乎挨在一起,说明他们经历共同的加倍事件。另外 Pso_Pso在0-0.06处有一个单独的峰,说明罂粟经历了近期的独有加倍事件。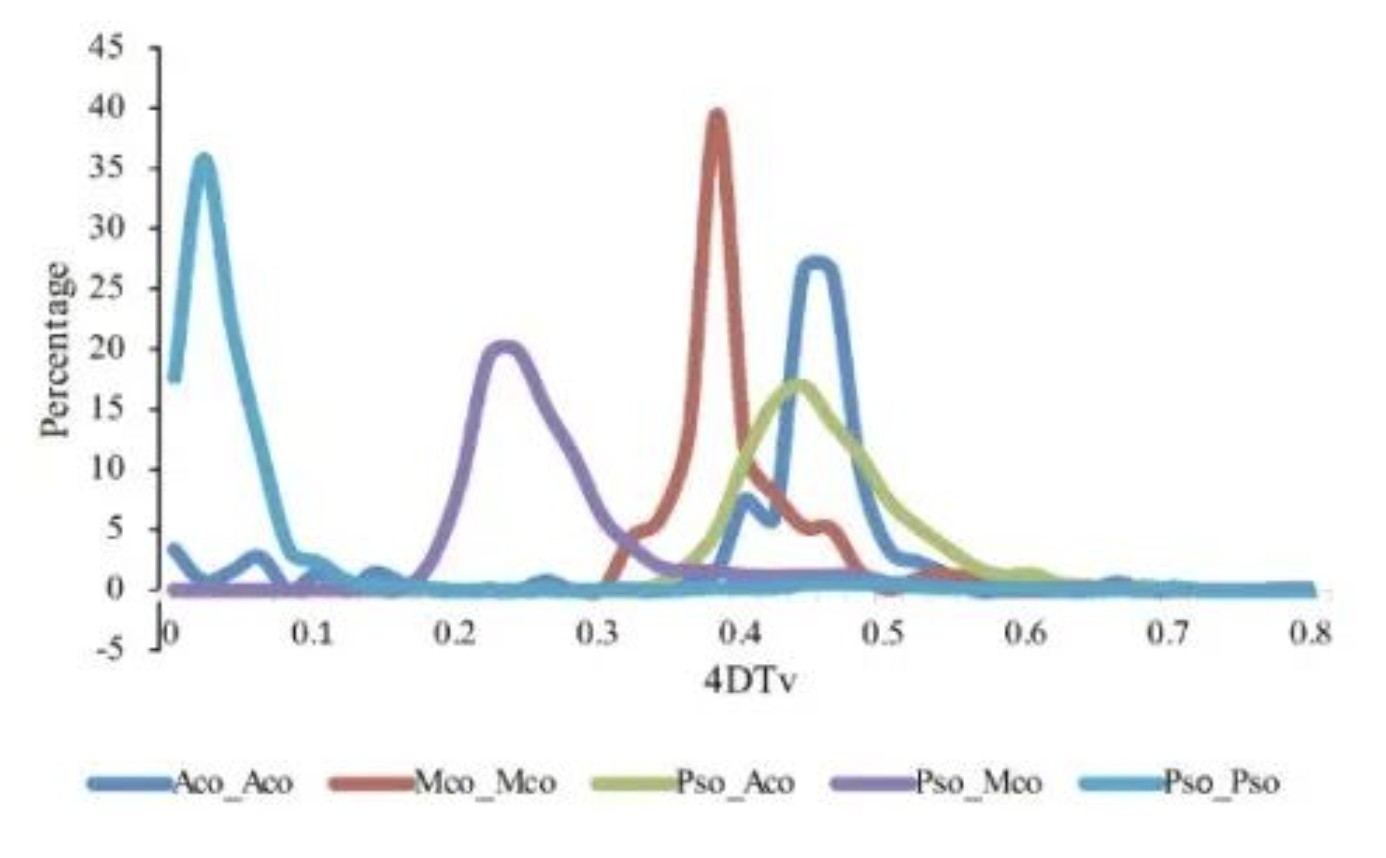
参考文献1. Kang Zhang, Xiaowu Wang, and Feng Cheng. Plant Polyploidy: Origin, Evolution, and Its Influence on Crop Domestication,Horticultural Plant Journal,20192. Van de Peer Y, Maere S, Meyer A. The evolutionary significance of ancient genome duplications[J]. Nature Reviews Genetics, 2009, 10(10): 725-732.3. Badouin, H., Gouzy, J., Grassa, C.J., Murat, F., Staton, S.E., Cottret, L., Lelandais-Briere, C., Owens, G.L., Carrere, S., Mayjonade, B., et al. (2017). The sunflower genome provides insights into oil metabolism, flowering and Asterid evolution. Nature 546, 148-152.4. Daccord, N., Celton, J.M., Linsmith, G., Becker, C., Choisne, N., Schijlen, E., van de Geest, H., Bianco, L., Micheletti, D., Velasco, R., et al. (2017). High-quality de novo assembly of the apple genome and methylome dynamics of early fruit development. Nat Genet 49, 1099-1106.5. Guo, L., Winzer, T., Yang, X., Li, Y., Ning, Z., He, Z., Teodor, R., Lu, Y., Bowser, T.A., Graham, I.A., et al. (2018). The opium poppy genome and morphinan production. Science 362, 343-347.Jiao, Y. (2018). Double the Genome, Double the Fun: Genome Duplications in Angiosperms. Mol Plant 11, 357-358.6. Jiao, Y., Wickett, N.J., Ayyampalayam, S., Chanderbali, A.S., Landherr, L., Ralph, P.E., Tomsho, L.P., Hu, Y., Liang, H., Soltis, P.S., et al. (2011). Ancestral polyploidy in seed plants and angiosperms. Nature 473, 97-100.7. Qin, C., Yu, C., Shen, Y., Fang, X., Chen, L., Min, J., Cheng, J., Zhao, S., Xu, M., Luo, Y., et al. (2014). Whole-genome sequencing of cultivated and wild peppers provides insights into Capsicum domestication and specialization. Proc Natl Acad Sci U S A 111, 5135-5140.8. Tang, H., Wang, X., Bowers, J.E., Ming, R., Alam, M., and Paterson, A.H. (2008). Unraveling ancient hexaploidy through multiply-aligned angiosperm gene maps. Genome Res 18, 1944-1954.
Wu, S., Han, B., and Jiao, Y. (2020). Genetic Contribution of Paleopolyploidy to Adaptive Evolution in Angiosperms. Mol Plant 13, 59-71.9. Wu S, Han B, Jiao Y. Genetic contribution of paleopolyploidy to adaptive evolution in angiosperms[J]. Molecular Plant, 2019.10. Ren R, Wang H, Guo C, et al. Widespread whole genome duplications contribute to genome complexity and species diversity in angiosperms[J]. Molecular plant, 2018, 11(3): 414-428.11. Jiao Y. Double the genome, double the fun: genome duplications in angiosperms[J]. Molecular plant, 2018, 11(3): 357-358.12. Van de Peer Y, Mizrachi E, Marchal K. The evolutionary significance of polyploidy[J]. Nature Reviews Genetics, 2017, 18(7): 411.13. Ding, YM., Pang, XX., Cao, Y. et al. Genome structure-based Juglandaceae phylogenies contradict alignment-based phylogenies and substitution rates vary with DNA repair genes. Nat Commun 14, 617 (2023). https://doi.org/10.1038/s41467-023-36247-z14. Jia, KH., Liu, H., Zhang, RG. et al. Chromosome-scale assembly and evolution of the tetraploid Salvia splendens (Lamiaceae) genome. Hortic Res 8, 177 (2021). https://doi.org/10.1038/s41438-021-00614-y15. Pei, L., Wang, B., Ye, J. et al. Genome and transcriptome of Papaver somniferum Chinese landrace CHM indicates that massive genome expansion contributes to high benzylisoquinoline alkaloid biosynthesis. Hortic Res 8, 5 (2021). https://doi.org/10.1038/s41438-020-00435-5
- 发表于 2023-03-02 17:24
- 阅读 ( 13872 )
- 分类:基因组学
你可能感兴趣的文章
相关问题
- 请问已知4dtv值,如何计算具体的分歧时间呢? 0 回答
- ks计算物种共同祖先WGD事件的问题 2 回答
