如何按照影响因子筛选文章--PubMed文献搜索技巧
无论您是学生还是老师,只要做科研,搜索文献就是一项必不可少的基本技能。
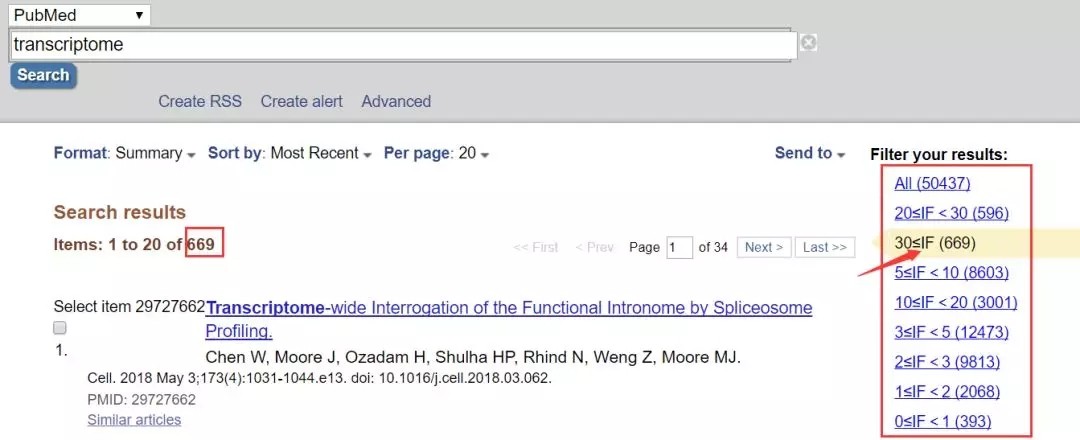
在搜索文献时,我们常常会用到PubMed数据库。通过关键词搜索文献是最简单也最常用的方法,比如搜索和"transcriptome"相关的文章,搜索结果是50437篇文章。如此多的文章,我们该如何找到我们想要的呢?
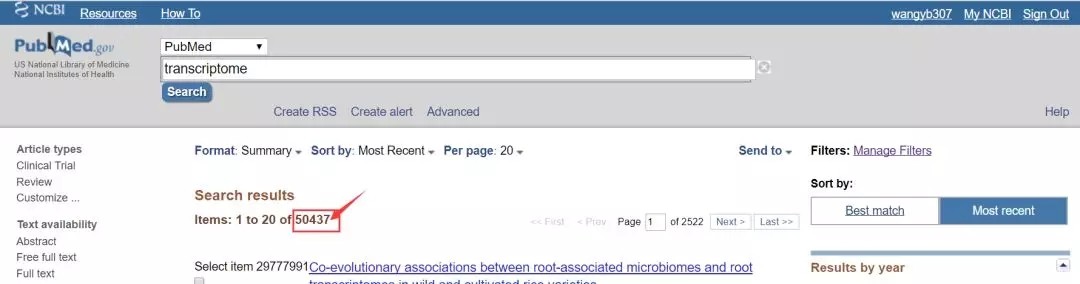
即使我们按照年份进行筛选,比如只看2018年发表的文章,也有3032篇。
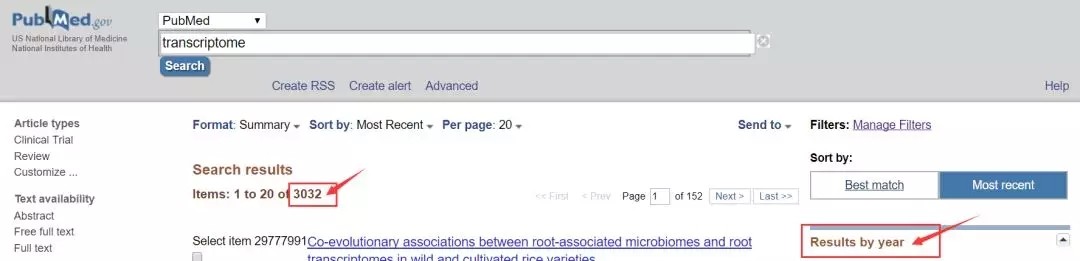 通常我们不是特别在意文章具体是哪一年发的,而是更关心这文章发的期刊是否权威,毕竟做科研要紧跟大牛的脚步
通常我们不是特别在意文章具体是哪一年发的,而是更关心这文章发的期刊是否权威,毕竟做科研要紧跟大牛的脚步![]() 。那么我们该如何按照自己的需要筛选影响因子在目标范围内的文章呢?
。那么我们该如何按照自己的需要筛选影响因子在目标范围内的文章呢?
接下来,小编就给大家分享PubMed数据库的一个高级功能来解决这个问题(文末提供制作好的过滤器下载)。
PubMed影响因子过滤器
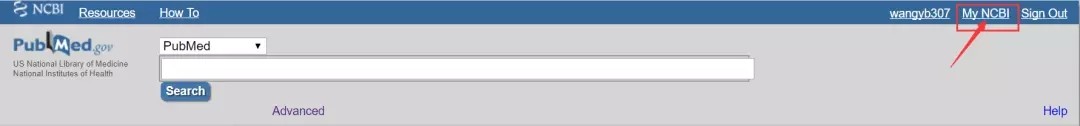
首先,打开PubMed网站:https://www.ncbi.nlm.nih.gov/pubmed,按下图说明点击“Signin to NCBI”,输入账户密码,登录NCBI。
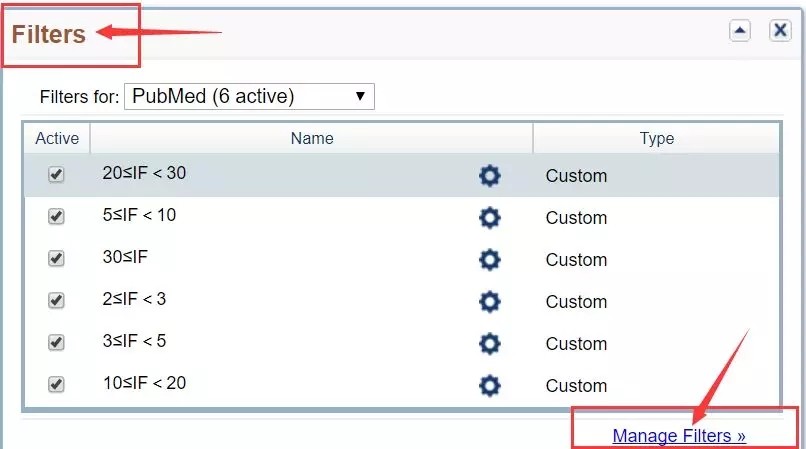
然后,点击右上角的"My NCBI",进去之后找到"Filters"选项,点击"Manage Filters"按钮进行"custom filter"的创建。
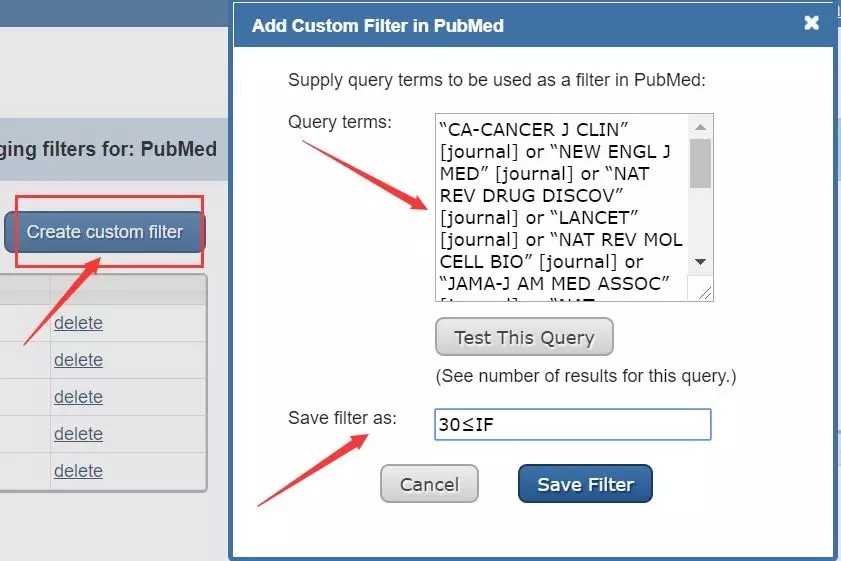
点击"Create custom filter"按钮,在"Query terms"框内按照特定的格式输入目标影响因子范围内的期刊列表(下文有列表制作方法),并保存为相应的名称即可。如下图:输入筛选好的影响因子大于30的期刊列表,命名为"30≤IF",其他影响因子范围内的期刊也按照相同的方法进行输入并保存。
 接下来,创建好"custom filter"之后,将过滤器前的"Active"选项勾上,这样该过滤器就可以正常使用了。
接下来,创建好"custom filter"之后,将过滤器前的"Active"选项勾上,这样该过滤器就可以正常使用了。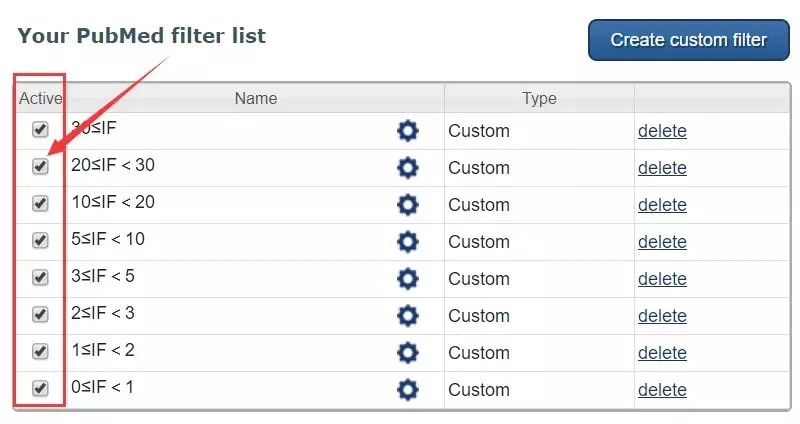
在这里,我们试一下设置好的过滤器。我们还是搜索关键词“transcriptome ”,得到的文章总数依然是5万多篇,然后按照过滤器中"30≤IF"的指标进行过滤,结果是669篇相关的高分文章。这说明我们的影响因子筛选过滤器发挥作用了。
 看到这里大家就会发现,整个过程中,最重要的就是如何制作指定影响因子范围的期刊列表(Query terms)(文末提供整理好的列表下载),下面进行难点讲解:
看到这里大家就会发现,整个过程中,最重要的就是如何制作指定影响因子范围的期刊列表(Query terms)(文末提供整理好的列表下载),下面进行难点讲解:
第一、准备好包含期刊名称和影响因子的列表,并按照影响因子高低进行排序;
第二、直接复制列表中影响因子在一定范围内的期刊名称(注意:最好是简称),并新建一个word文档,然后右键选择性粘贴为文本(这里以影响因子大于30的为例);
第三、Ctrl+H打开Word的替换功能,把^p(换行符)全部替换为" [journal] or ";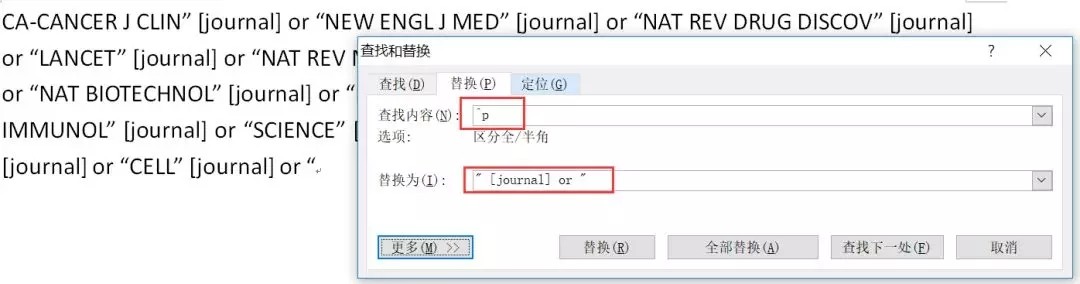
第四、在替换好的文档开头加上一个单引号",并把文档末尾的or "删掉。这样,30≤IF的期刊列表就制作好了,将该段文本黏贴进 "custom filter"里的"Query terms"即可。
以上就是按照影响因子搜索文章的全过程。
为了方便大家,小编已经把生命科学类的期刊(包括农学、医学、生物学等)按照30≤IF、20≤IF<30、10≤IF<20、5≤IF<10、3≤IF<5、2≤IF<3、1≤IF<2、0≤IF<1整理好了对应的过滤器。
如果您需要相关文件,可以在公众号对话框输入:过滤器+姓名+您的邮箱,即可获得下载链接。
最后,如果您感觉小编写的还不错,也为了让更多的小伙伴学会这个操作,请转发本文至微信朋友圈,这是对小编工作最好的鼓励!
更多生物信息课程:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程、基因家族文献思路解读
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘、转录组文献解读
6. NCBI数据上传
- 发表于 2018-06-20 10:18
- 阅读 ( 23684 )
- 分类:软件工具
