解决基因组当中蛋白质序列ID和gff中ID不一致的问题
解决基因组当中蛋白质序列ID和gff中ID不一致的问题
经常有小伙伴做基因家族分析时遇到gff里面的mRNA ID与蛋白或者cds 序列 ID不一致的问题: 导致程序报错
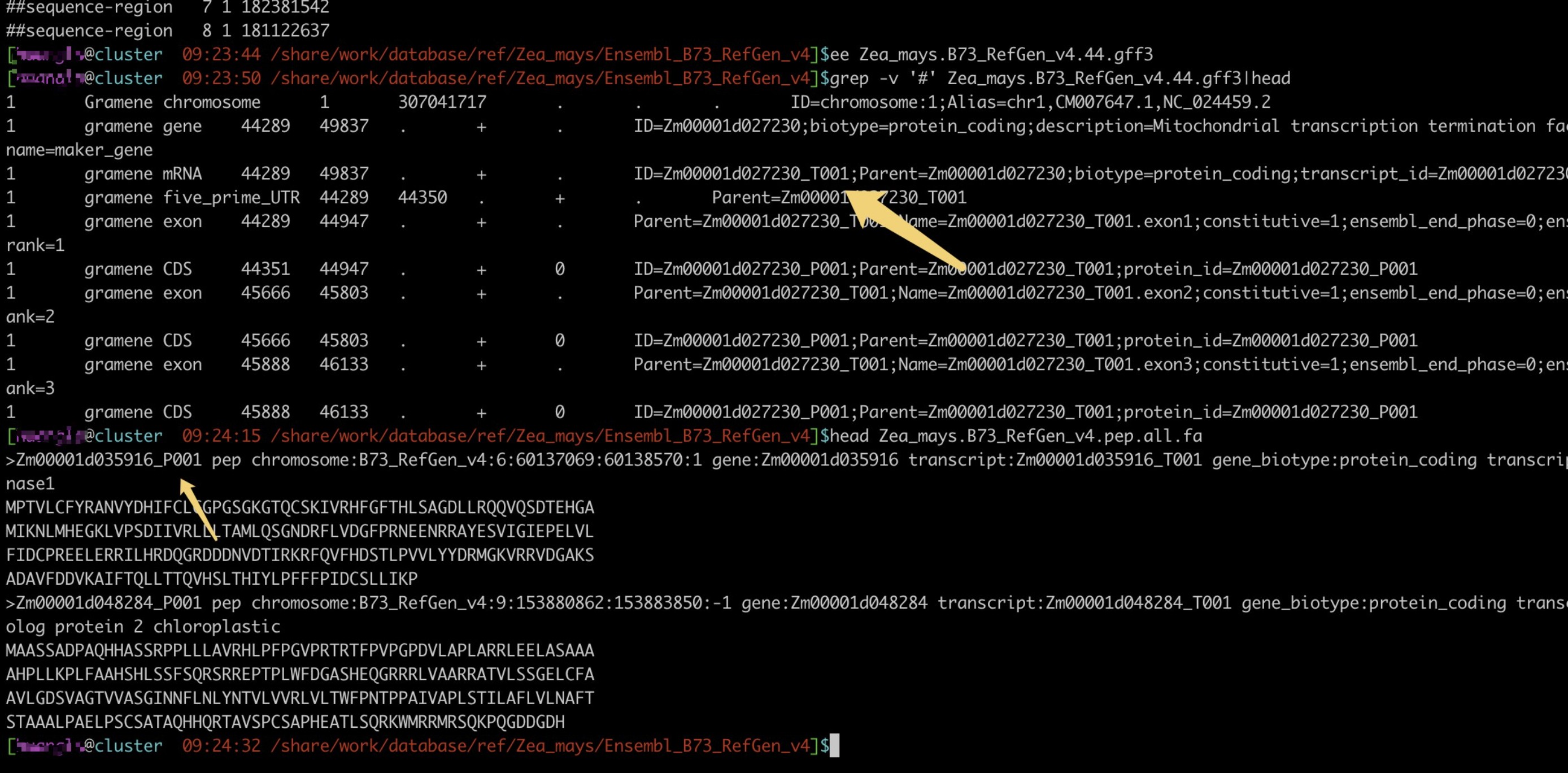
例如下面玉米的GFF里面的mRNA ID和基因组基因蛋白序列的ID不一致的问题;
一劳永逸的彻底解决办法是,自己根据GFF信息,从基因组里面提取基因的蛋白序列和cds序列;这里给出代码:
# 设置文件路径和物种拉丁名,并运行
species=Zea_mays
genome=Zea_mays.B73_RefGen_v4.dna.toplevel.fa
gff=Zea_mays.B73_RefGen_v4.44.gff3 #以下代码不用修改,可直接运行
#保留蛋白编码基因
agat_sp_filter_feature_by_attribute_value.pl --gff $gff --attribute biotype --value protein_coding -t '!' -o $species.protein_coding.gff3
#保留最长转录本
agat_sp_keep_longest_isoform.pl --gff $species.protein_coding.gff3 -o $species.longest_isoform.gff3
#Ensembl 下载的基因组 会在基因和mRNA ID前面加gene: 和 transcript:这里给去掉
sed -i 's/gene://' $species.longest_isoform.gff3
sed -i 's/transcript://' $species.longest_isoform.gff3
#提取cds序列
/share/work/biosoft/TransDecoder/latest/util/gff3_file_to_proteins.pl --gff3 $species.longest_isoform.gff3 --fasta $genome --seqType CDS >$species.cds.fa
#提取pep序列
/share/work/biosoft/TransDecoder/latest/util/gff3_file_to_proteins.pl --gff3 $species.longest_isoform.gff3 --fasta $genome --seqType prot >$species.pep.fa
以上代码可以在基因家族分析镜像 2.0中运行: omicsclass/gene-family:v2.0
注意,以上命令会将基因组读入内存,因此电脑内存不足的可能会报错,有条件的可以购买我们的大内存云服务器:https://study.omicsclass.com/
- 发表于 2023-04-06 09:38
- 阅读 ( 7263 )
- 分类:基因家族分析
你可能感兴趣的文章
- GFF或GTF格式转bed 2733 浏览
- gbff 格式注释文件转换成gff3注释文件格式 15750 浏览
- 根据GFF文件提取基因组中基因的蛋白,CDS,CDNA序列 4346 浏览
- 人类miRNA-mRNA 与 miRNA-lncRNA 靶向数据库总结 3571 浏览
- GFF文件格式不标准,第三列只有GENE处理方法 3797 浏览
相关问题
- 搜集的gff3文件;第一列不同,有的是chr,有的是id;这个影响吗? 2 回答
- 老师,我在做饭基因家族分析,我搜集的基因组的gff3文件,第一列有的是chr有的是基因id,这个会影响后续结果嘛? 2 回答
- 老师,为什么我的cds和pep文件导不出来呀?导出来报错,并且文件内容无。最后一张照片时我退出重进后的历史命令 2 回答
- 基因家族分析2.0,做基因结构分析时报错,完全按照提供指令做的 1 回答
- 老师,我这个是桑树的fasta和gff文件,不是从官网下载的。假如各个网站都找不到我想要的物种的基因组文件,只有手头这个文件,我要怎么做才能和教程的代码连接上呀?或者说碰到这种目标物种查不到的情况可以怎么做呢? 1 回答
- 老师,下面是报错、染色体信息、gff文件中的染色体和命令行 2 回答
2 条评论
请先 登录 后评论