GEO数据芯片数据挖掘-在线工具分析GDS数据
继安利GEO数据库和芯片数据的差异分析方法(欢迎来戳:No.1, No.2)之后,小编又来卖安利了。这次主要针对GEO DataSet 数据(GDS数据)教大家如何在线进行数据分析。
毕竟不是所有人都会编程敲代码,小白人生谁来解救?所以,小编就开始搬砖了,教大家利用GDS数据对应的Data Analysis Tools完成数据的基本分析,简单几步轻松易学~
GEO DataSet背景介绍
GEO数据库主要接收基因表达数据。实验研究项目中一般包含多个样本基因表达数据,研究人员进行数据上传之后,网站管理人员会根据实验样本的处理,数据来源等对数据进行整理,将能够进行生物学比较和统计的样品数据归纳整理到一起,形成唯一的DataSet record(GDS accession)。在这个DataSet中,所有的样品数据来源于同一种平台,数据背景处理和标准化方法都一致。
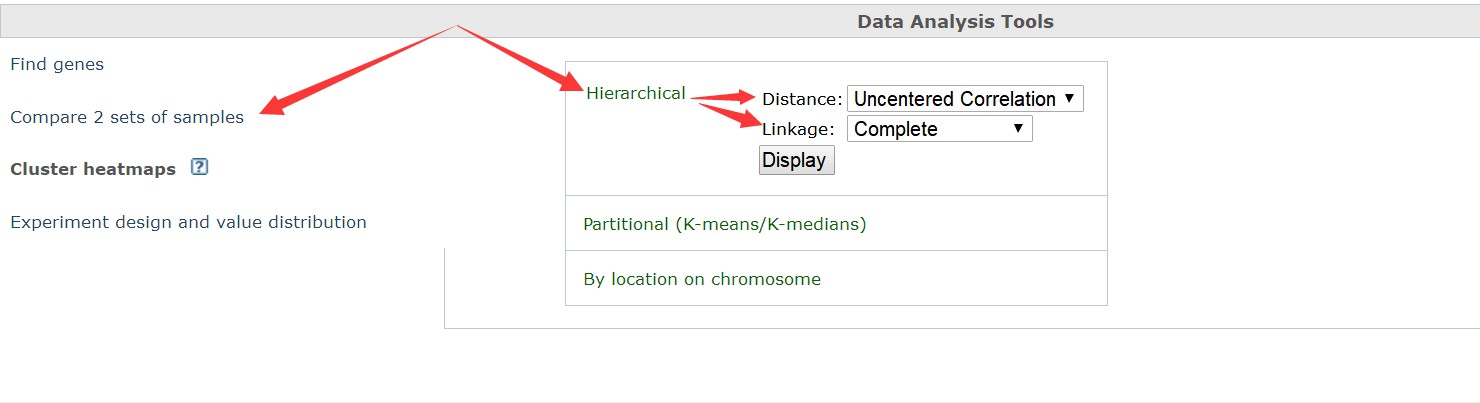
而针对每一个GDS数据,都可以通过网站在线工具:Data Analysis Tools 实现一些基本分析,包括差异分析、聚类分析等(见下图)。小编将以GDS6010对应都18个样品芯片数据为例给大家演示分析过程。
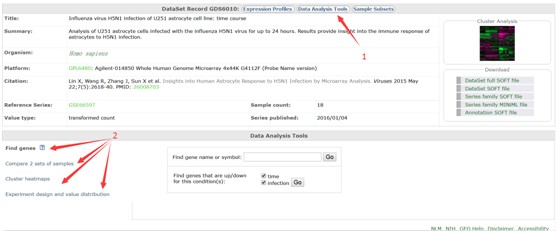
GDS数据搜索
由GEO主页进入GDSbrowser(GDS数据搜索网址: https://www.ncbi.nlm.nih.gov/sites/GDSbrowser)页面,输入登录号:GDS6010(下图),点击Search就可以进入数据页面,搜索结果如上图内容。
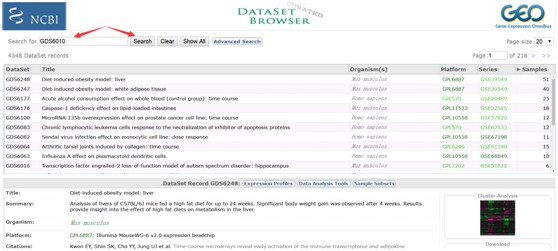
差异分析
点击分析页面左下角Compare 2 sets of samples,第一步,选择检验方法和阈值(见下图A);第二步,选择差异对比组,点击Select which Samples to put in Group A and Group B,出现样品处理信息,据此设定A组样品和B组样品(见下图B);第三步,针对A组和B组,进行差异分析(见下图C)
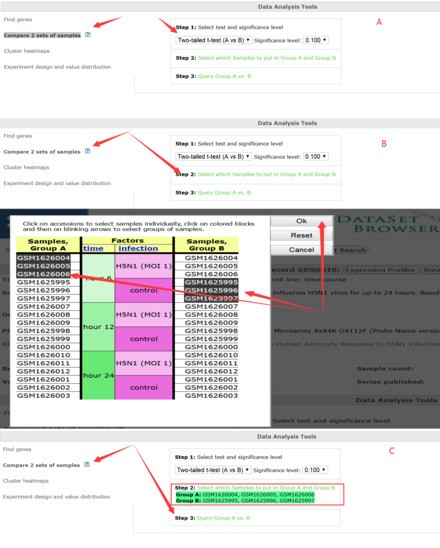
例如:针对Two-tailed t-test,阈值为0.01,对照组6h vs处理组6h,可以筛选到4140个探针表达谱(结果如下图),其缺点是,只能筛选出对应的探针单个表达谱,不方便进行批量下载数据。
聚类分析
聚类分析包括多种分析方法,以层次聚类为例,选择具体的聚类方法(如下图),进行聚类,如,Distance: Euclidean,Linkage:Average/UPGMA。
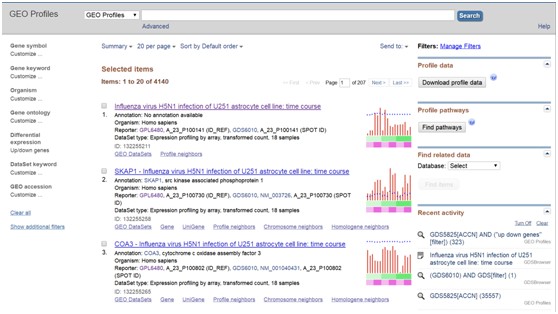
聚类分析的热图结果如下:
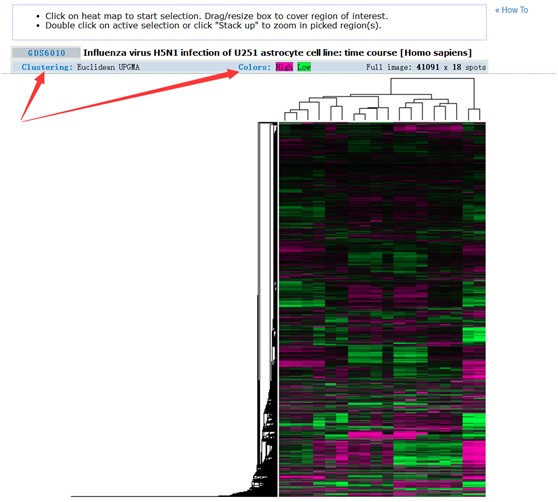
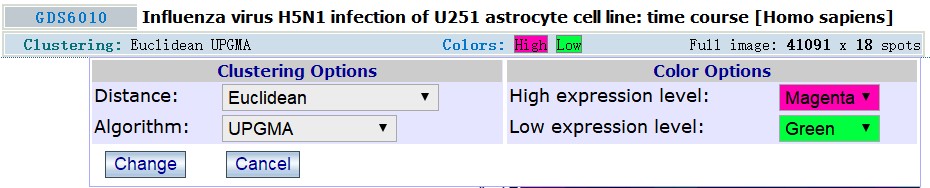
结果为所有探针数据对应聚类热图,图片中以颜色在不同样品中的变化对应表达数据的变化,而在结果显示页面,点击Clustering或者Colors可以通过修改具体的层次聚类方和图片颜色设置(如下图)。
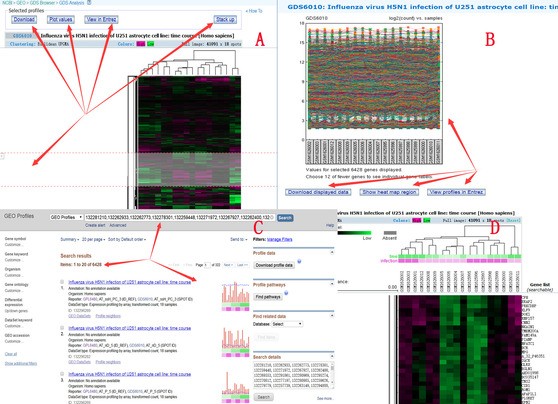
选定表达谱热图的一部分区间后(点击热图,出现选择框,可以调整选择框大小),页面弹出下载等相关设置,可以针对选定区域,进行所有表达数据下载,绘图,单基因表达谱搜索,以及显示选定区域的独立热图(如下图A、B、C、D)。
数据分布
通过Data Analysis Tools 可以查看数据整体的分布情况,例如点击Experiment design and value distribution,可以出现数据box分布图,如下:
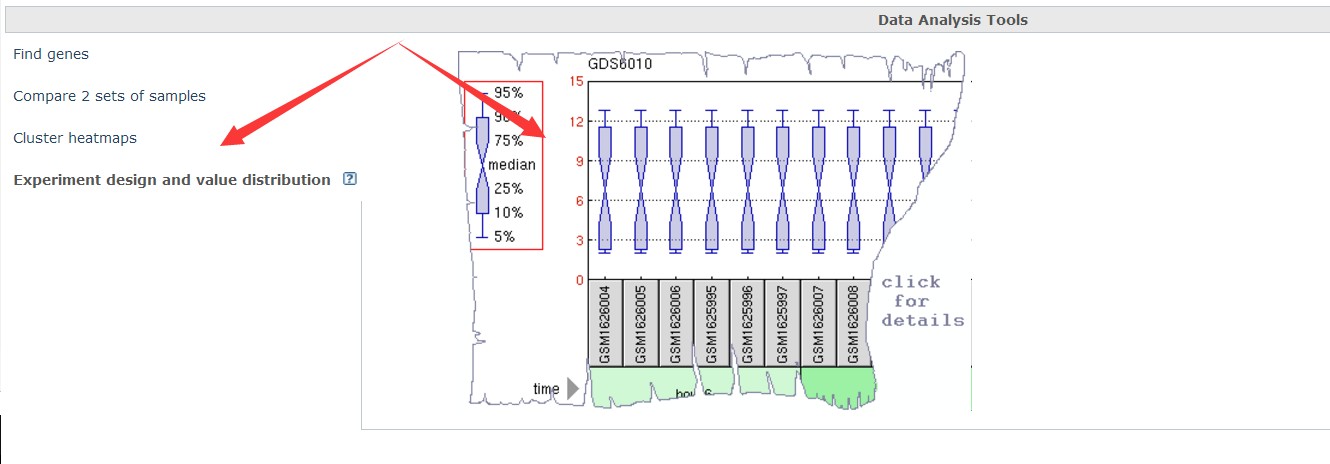
单击图片位置,可以查看整个GDS所有样品的数据分布情况,同时查看样品到处理信息(如下图)。
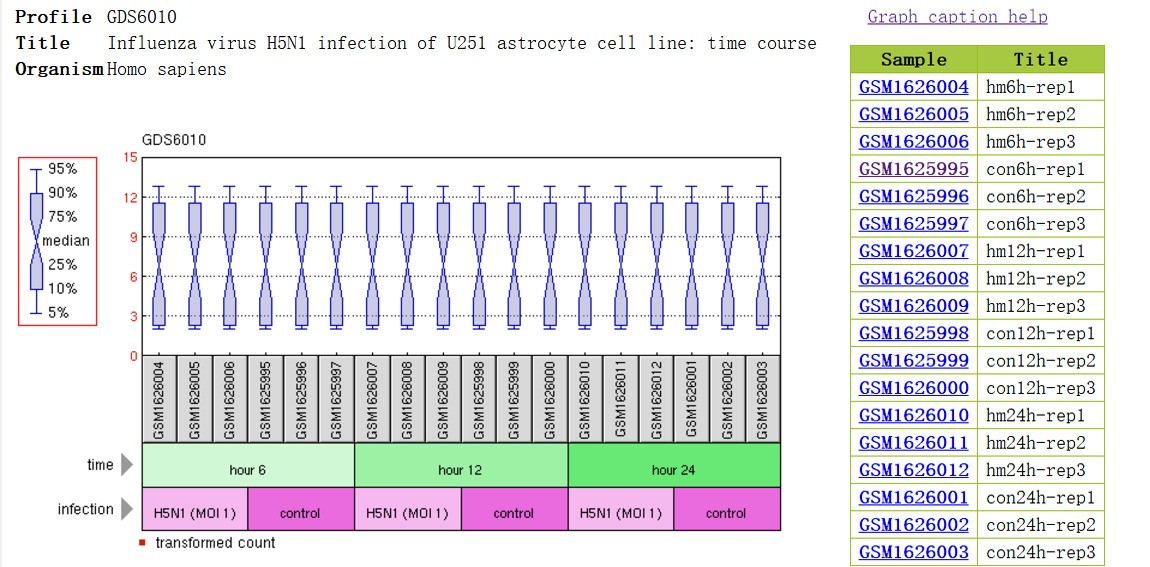
总结
针对GDS数据,利用在线工具可以进行比较基本的分析和数据筛选,虽然差异比较结果展示、批量下载存在困难等等,但是操作过程相对简单,适用于没有编程基础的科研人员学习,方便大家对数据内容进行初步掌握。
推荐课程:GEO芯片数据挖掘、GEO芯片数据标准化、R语言入门、R语言画图
更多生物信息课程:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程、基因家族文献思路解读
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘、转录组文献解读
5. 微生物16S/ITS/18S分析原理及结果解读、OTU网络图绘制、cytoscape与网络图绘制课程
6. 生物信息入门到精通必修基础课:linux系统使用、perl入门到精通、perl语言高级、R语言入门、R语言画图
7. 医学相关数据挖掘课程,不用做实验也能发文章:TCGA-差异基因分析、GEO芯片数据挖掘、GEO芯片数据标准化、GSEA富集分析课程、TCGA临床数据生存分析、TCGA-转录因子分析、TCGA-ceRNA调控网络分析
8.其他,二代测序转录组数据自主分析、NCBI数据上传、二代测序数据解读
- 发表于 2018-06-21 17:31
- 阅读 ( 10818 )
- 分类:软件工具
你可能感兴趣的文章
- 用AI写代码批量整理GEO数据库中的单细胞数据 1901 浏览
- 单细胞转录组数据挖掘流程记录-头颈癌(HNSCC)(GSE181919) 2496 浏览
- 细胞焦亡、铁死亡及铜死亡等基因集 7789 浏览
- 记录一个数据下载:GSE12417 2477 浏览
- 生信挖掘GEO+TCGA 文献速览 2590 浏览
- IF=3.47|非肿瘤-眼部老年黄斑变性ceRNA文章思路 3288 浏览
相关问题
- 代码报错 2 回答
- 老师您好,我这个是双通道芯片,我把read.maimages的所有source都试了一遍,还是不能把原始文件读进来 1 回答
- 老师您好,我在进行GEO分析时,打开分组文件group_file时出现报错,请问我该如何解决? 1 回答
- 请问老师,GEO数据分析时,运行gset = getGEO(GSE, GSEMatrix =TRUE, AnnotGPL=FALSE,destdir=workdir)时,遇到如图中报错,该如何处理呢? 1 回答
- Error in (function (classes, fdef, mtable) : unable to find an inherited method for function ‘bg.correct’ for signature ‘"HTAFeatureSet", "character"’ 0 回答
- 安捷伦双色通道 “RawData=read.maimages(files=files,source="genepix") “报错” 2 回答
