jcvi微共线区域(Microsynteny)可视化
如何使用jcvi通过调整相关文件参数展示局部共线性情况
使用jcvi进行局部区域(感兴趣基因)共线性绘图需要准备三个文件:
- 记录各物种基因位置信息的bed文件
- 包含感兴趣基因同源区块信息的block文件
- 包含绘图参数的layout.csv文件
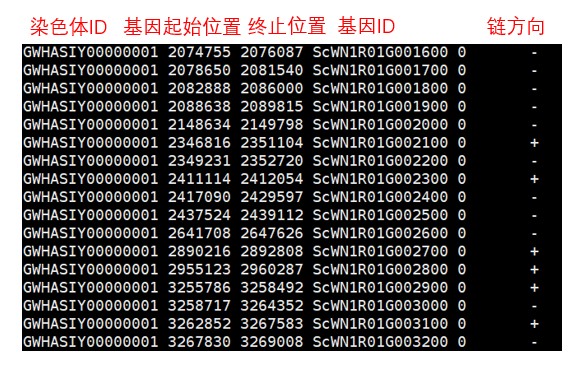
bed文件格式:(各列对应信息如下)
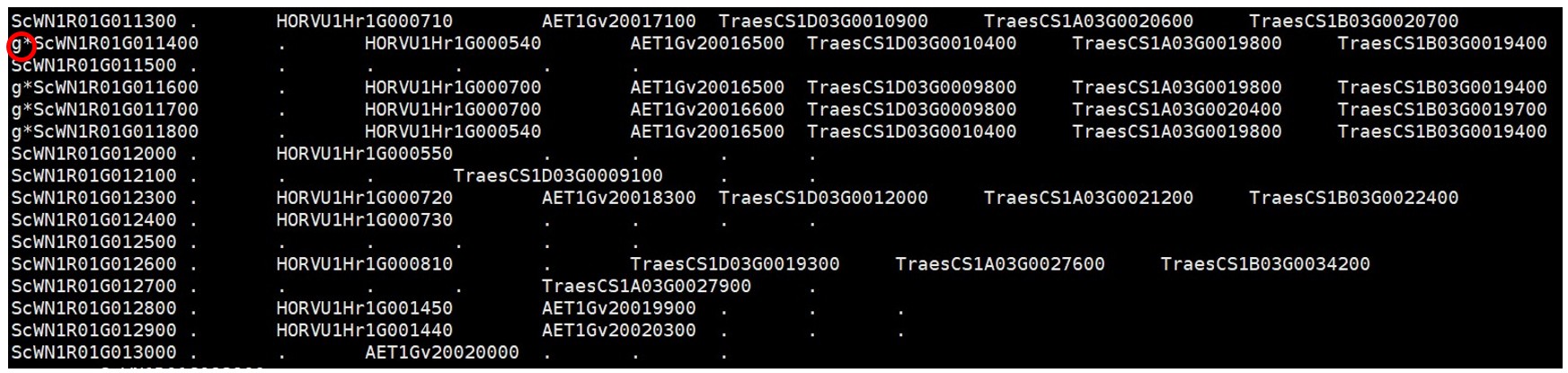
block文件格式:每一列为各物种的同源基因ID(最佳匹配,通过compara 参数--iter=1指定),点表示该同源基因在物种中没有被注释到,可以通过该文件指定感兴趣基因的线条颜色,如图中红圈所示
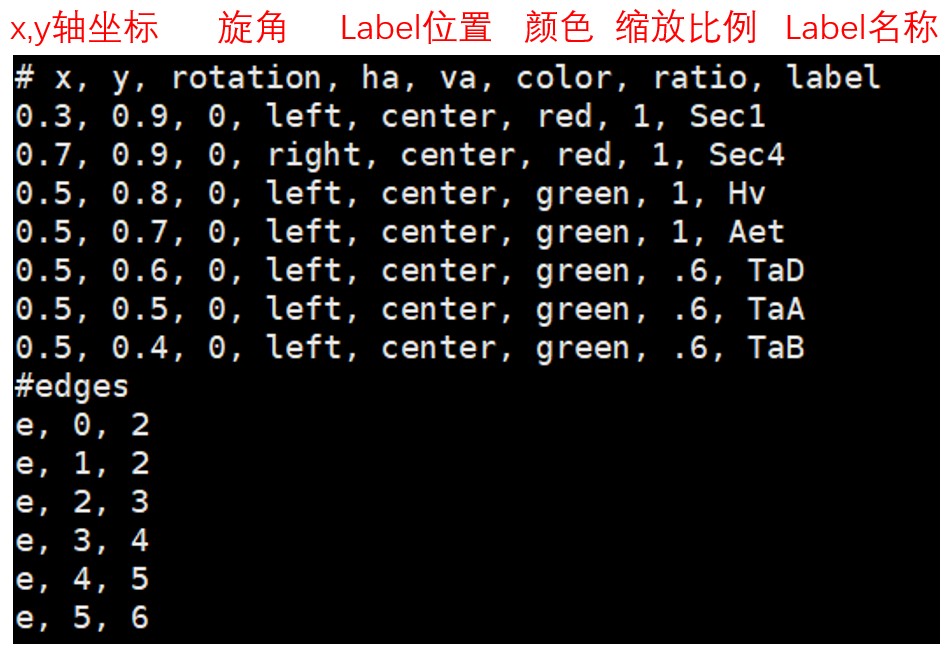
layout.csv文件格式:上半部分指定了track的各种信息,edges中制定了绘制的物种共线性关系,如图中e,0,2指定了绘制block文件中第一列和第三列物种的共线性关系
运行代码:
python -m jcvi.graphics.synteny blocks bed layout.csv
效果图如下:
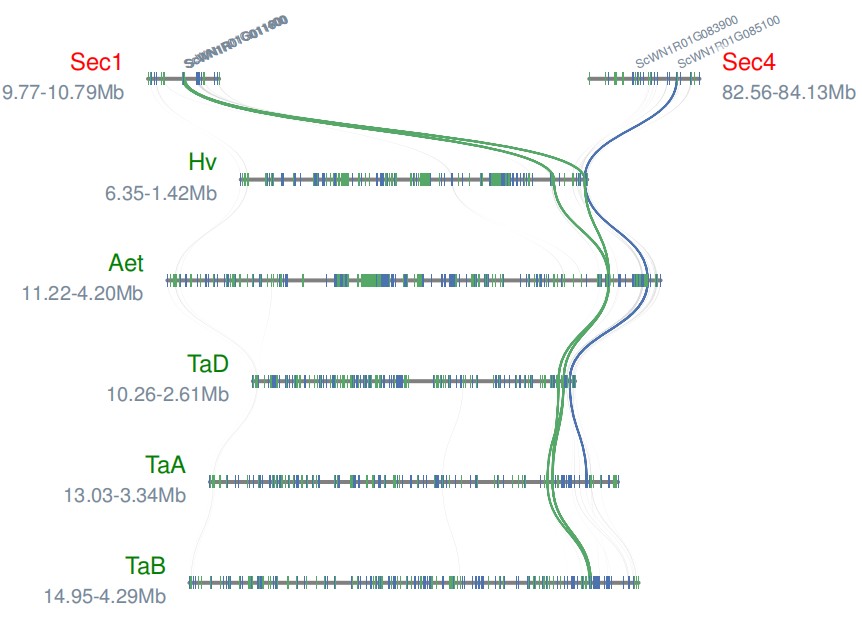
参考:
https://blog.csdn.net/u012110870/article/details/127425032
- 发表于 2023-08-15 18:06
- 阅读 ( 3237 )
- 分类:基因组学
你可能感兴趣的文章
- 已知目标基因,如何进行局部共线性绘图? 6884 浏览
- python 版本的mcscan 绘图输出报错, need more than 3 values to unpack 2171 浏览
- canda 安装的jcvi 遇到字体错误: UserWarning: findfont: Font family [u'sans-serif'] not found. Falling back to DejaVu Sans (prop.get_family(), self.defaultFamily[fontext])) 8202 浏览
- AttributeError: 'LayoutLine' object has no attribute 'color' 7974 浏览
0 条评论
请先 登录 后评论
