picard进行ReorderSam时报错
picard ReorderSam报错解决
在使用GATK 进行SNP calling前,我们通常需要使用picard中的ReorderSam功能对原始sam文件中的染色体进行重新排序:
java -XX:ParallelGCThreads=5 -Xmx50g -jar picard.jar ReorderSam -I bam -O bam -SD dict
下面是两种常见的报错和解决办法:
1.bam has invalid uncompressedLength
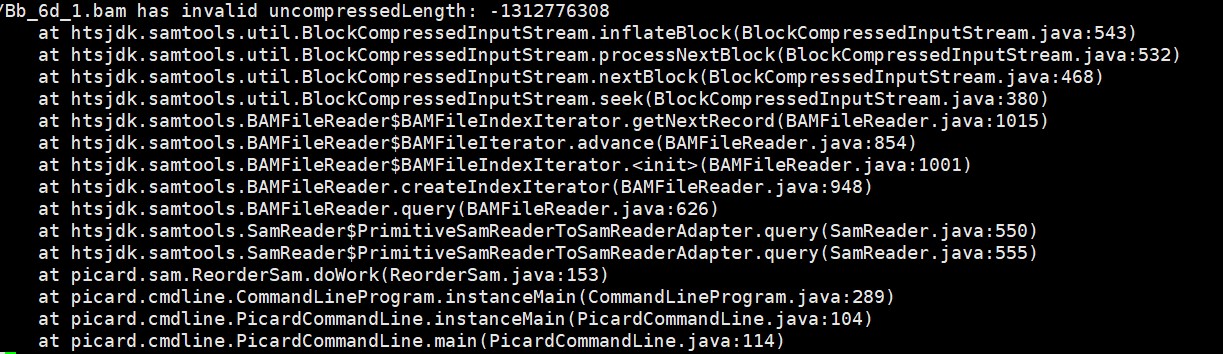
以上问题的出现通常是由于bam文件无法和索引文件产生对应,对索引文件(bai)进行更新即可解决问题。
2.No space left on device
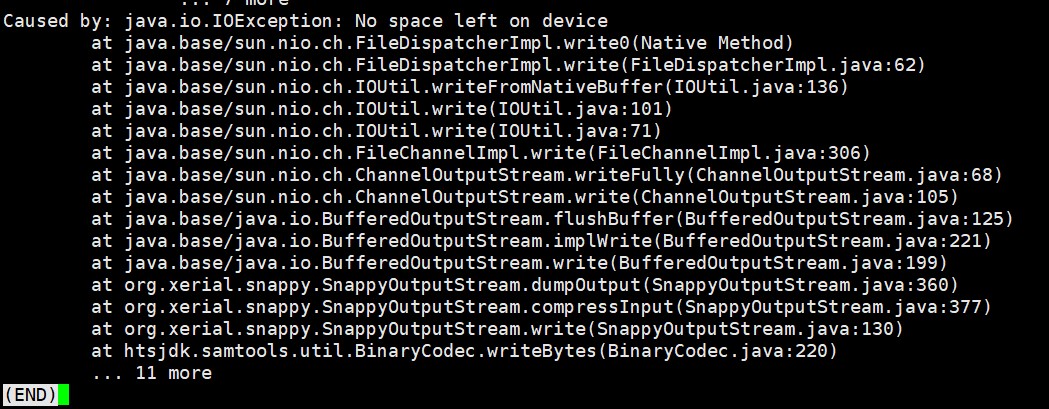 原因是缓存把tmp占满了,解决办法是在运行jar包时指定自己的缓存存储目录:
原因是缓存把tmp占满了,解决办法是在运行jar包时指定自己的缓存存储目录:
java -XX:ParallelGCThreads=5 -Xmx50g -Djava.io.tmpdir=./tmp -jar picard.jar ReorderSam -I bam -O bam -SD dict
- 发表于 2023-09-06 09:51
- 阅读 ( 1168 )
- 分类:其他
你可能感兴趣的文章
相关问题
0 条评论
请先 登录 后评论
