小尺度的种群历史分析——∂a∂i原理解析
1.频谱生成
∂a∂i是一个强大的软件工具,用于模拟多个群体间遗传变异的联合频谱,并将其用于群体遗传推断,而频谱文件可以从VCF文件中获得。
在种群分化之前存在的基因型是祖先基因型(ancestral),而在分化后突变产生的基因型被认为是衍生基因型(derived),频谱文件本质是对衍生等位基因分布的统计,我们以下图为例,为大家解释频谱文件的生成。
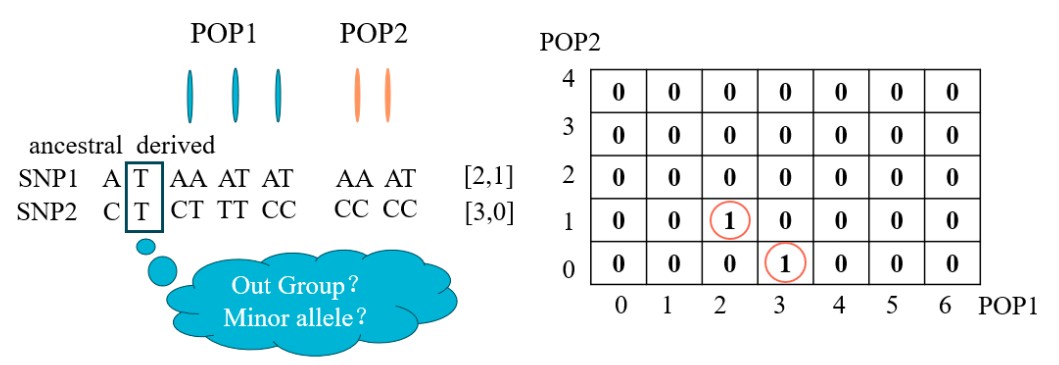
对于二倍体物种而言,等位基因数目是个体的两倍。例如,上图中第一个群体(POP1)有三个个体,总的等位基因数量就是6,第二个群体(POP2)有两个个体因此有4个等位基因。我们只需要统计在两个群体中衍生基因型的数目,当没有外群,也就是无法得知衍生基因型是什么的时候,默认次等位基因型也就是Minor allele为衍生基因型。
假设上图中SNP1的祖先基因型是A,衍生基因型是T,我们只需要在群体中统计T等位基因出现的数量即可:在第一个种群中有2个衍生基因型(T),第二个种群中有1个(T),因此在2,1对应的位置记录这个SNP。SNP2同理,第一个群体里有3个衍生基因型,第二个群体中为0,因此在3,0记录。这个位置的数字表示在两个群体中呈现这样衍生基因型分布的SNP数目。
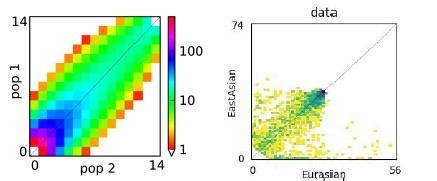
将表格进行可视化,即为频谱热图。左图是有外群情况下的热图;右图为无指定外群的热图(unfold SFS),次等位基因是第二多的基因型,因此不会超过总数的1/2,热图只有一半(fold SFS)。
2. 频谱可反映群体历史
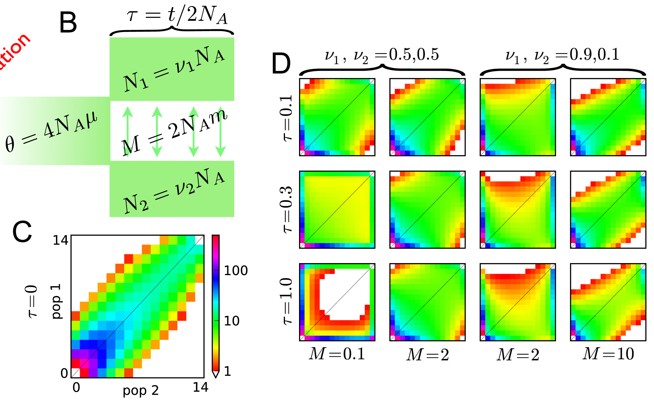
从热图上可以看出群体之间的频谱变化主要与两群体分化时间 τ,基因交流程度M,群体大小υ相关。当种群之间开始分化、产生基因交流的时候,热图会向边际转移。上图中,每一行分化时间是一样的,每一列的种群交流是一致的,当分化时间越长、交流很少的时候会出现左下角两个群体频谱几乎全部分开的情况,而往右随着交流的增加热图也越来越集中于对角线,说明遗传背景越来越近,同样,往上表示分化时间的越来越短,热图也逐渐集中。
最后总结一下(如下图):当两个群体关系比较近的时候,衍生等位基因的分布一趋于一致,那么频谱集中在对角线的地方(下图)。频谱右上角等位基因频率越来越高这些等位基因趋于在群体中固定,左下角等位基因频率很低区域丢失。
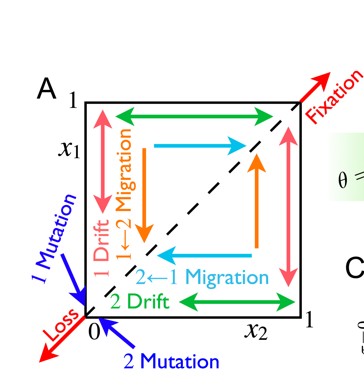
3.∂a∂i推断种群历史原理
由于种群的分化过程中的频谱我们无法获得,只能得到现在的两个群体频谱。∂a∂i就是根据现在的频谱文件去模拟之前所经历的群体事件,根据最大似然法推断什么分化模型得到的结果是最符合当前频谱的,因此可以回答分化时间顺序、种群大小和基因交流这三个层面的问题。其在运行过程之前,需要我们对历史事件进行预设,即通过参数设置提供给它一些分析模型,而它对每个模型进行多次模拟,根据最大似然法对每一次模拟进行打分,得分越高,则认为越接近真实群体历史的模型。
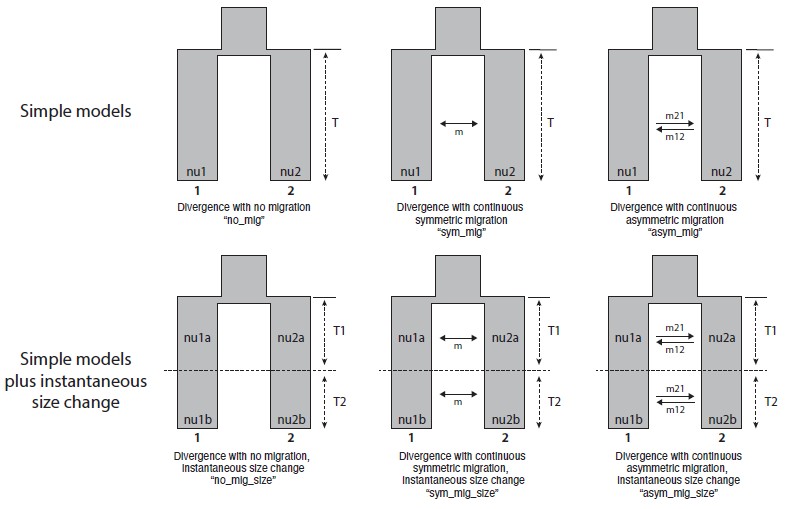
4.文献示例展示
例如2013年发表在 Nature Genetics 上的关于黄瓜驯化与群体多样性的文章 “A genomic variation map provides insights into the genetic basis of cucumber domestication and diversity”,在这个文章中,作者对来自印度、西双版纳、欧洲和东亚四个地方的黄瓜进行了种群分化的研究,同时通过遗传多样性和选择性清除定位了与驯化密切相关的候选区域并开发了标记。
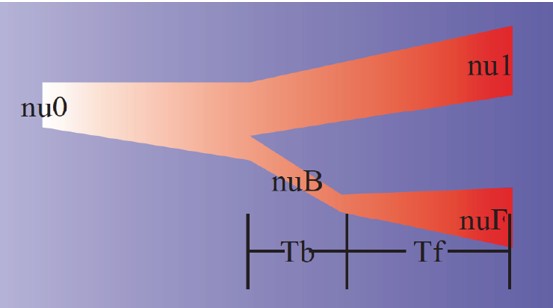
作者利用∂a∂i将两个栽培种(东亚种群和欧洲种群)与印度种群进行了比较,最终得到了以下模型:“nu0”是衍生群体起源的祖先群体的大小。在“Tb+Tf”时间点两个总群发生了分化,有效大小为“nuB”。在“Tf”时间,群体发生了扩为“nuF”。
利用∂a∂i分析群体之间近期的演化历史,我们已经更新到遗传进化视频课程中,大家如果想要自己动手分析可以观看以下课程:https://bdtcd.xet.tech/s/3QzQFb
- 发表于 2023-09-26 11:21
- 阅读 ( 2397 )
