重测序遗传图无验证刊发在《GIGA SCIENCE》上
使用分子标记构建遗传连锁图谱是进行基因定位的最有效方法之一,通过测序技术开发SNP标记进而构建遗传图谱的技术已经十分成熟。随着Illumina发布了novaseq,测序成本的进一步降低,以及越多越多的物种公布了基因组,可以确定的是重测序将在越来越多的物种里取代简化测序成为构建遗传图谱的首选方法。
下面我们就用一篇2017年发表在《GIGA SCIENCE》(if 6.8)上的文章来给大家简单介绍下重测序遗传图谱的定位效果吧。
实验材料及方法:
1. 研究物种:谷子(Foxtail millet)
2. 群体来源:父本Zhanggu,母本A2,构建了重组自交系F10(RILs)取其中184株进行重测序
3. 测序深度:共得到140Gb clean data,平均每个样品约2×
4. 观测性状:旗叶长度/宽度、叶片颜色、刺毛颜色、花药颜色、株高、穗位高、抽穗期、穗硬度、烯禾啶(一种除草剂)抗性,所有性状记录两年的数据。
主要结果概述:
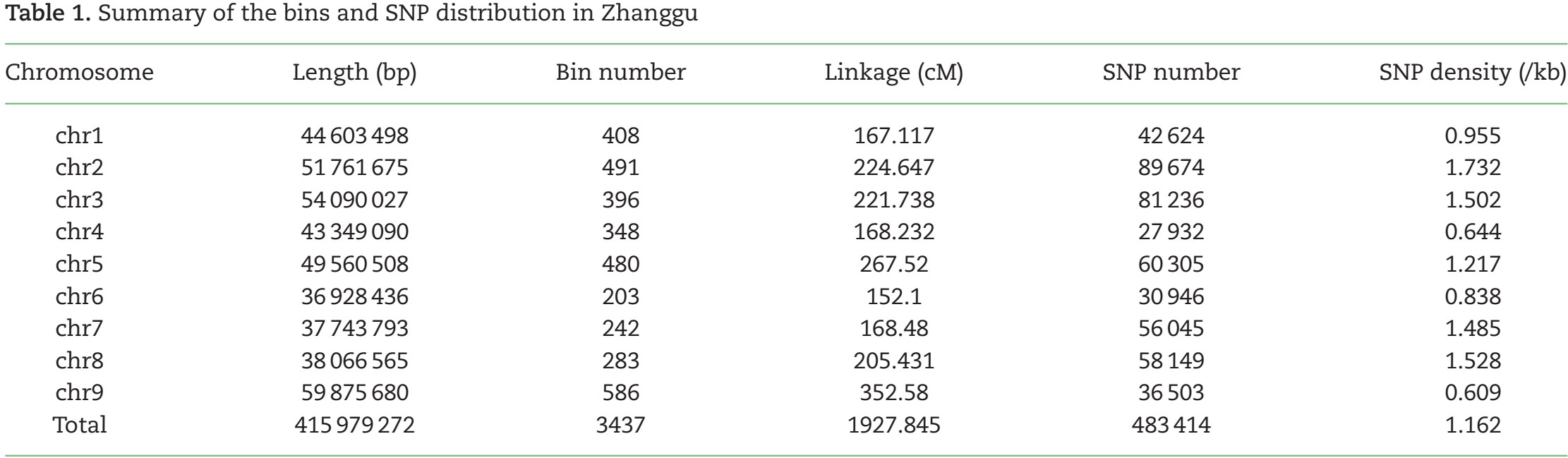
1. 遗传图谱的构建:使用3437个bins构建了包含9条连锁群、总图距为1927.8 cM的高密度遗传图谱
2. 性状的基因定位:5个质量性状(叶片颜色、刺毛颜色、花药颜色、穗位高、烯禾啶抗性)被证明为单基因控制的显性性状,且都被准确的定位到了染色体上确定的位置。数量性状中,株高、穗位高、抽穗期这三个性状都在多条染色体上找到了和主效、微效基因关联的标记,但是并没有找到和旗叶长度\宽度多年性状都一致关联的标记。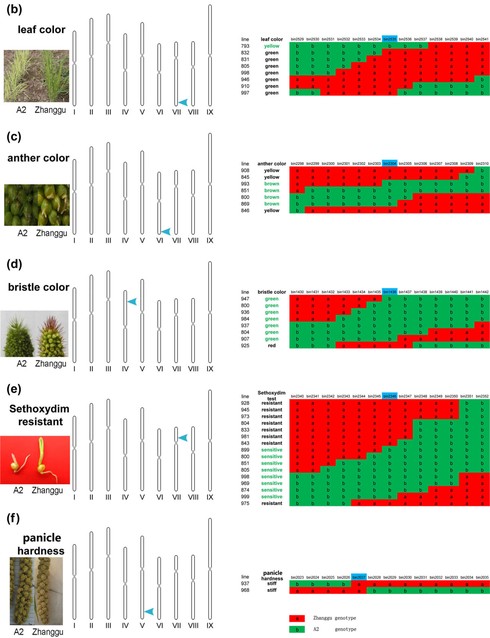
3. 对谷子已有基因组的升级:将之前未锚定的16M scaffold整合到了Zhanggu 基因组上,并填补了3158个gaps;对谷子Yugu基因进行了组装错误的修订,并填补了2874个gaps。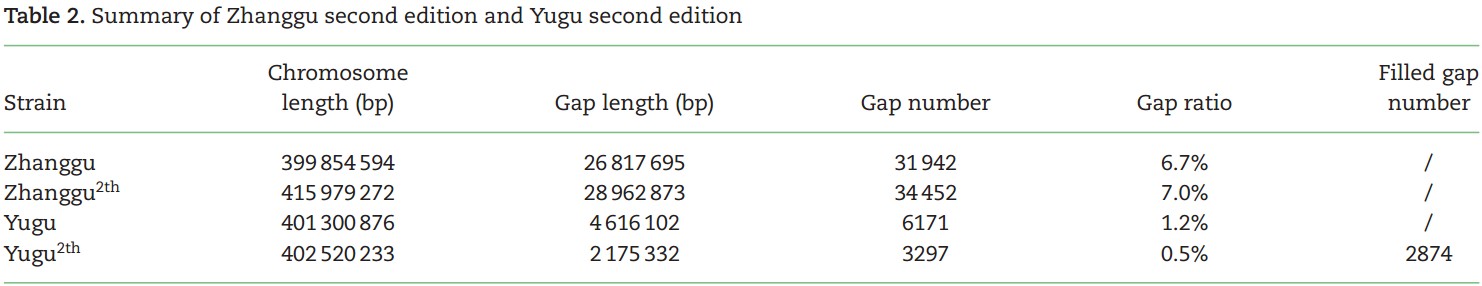
平平无奇?为什么能发6分文章
大家可以看到这篇文章选用的实验材料、定位的农艺性状、定位的方法虽然没有特别值得一提的地方,但也都是反复论证过的经典做法,结合重测序带来的超高密度的分子标记,得到的定位效果无疑是令人满意的。另外对物种基因组的完善也是简化测序所办不到的。
就像前几年简化测序代替了SSR来作图,重测序替代简化测序进行作图的大势已经到来,手里有遗传群体的老师们赶紧来试试吧!
- 发表于 2018-04-21 20:41
- 阅读 ( 3848 )
- 分类:文献解读
你可能感兴趣的文章
- 遗传图绘制: 1210 浏览
- 遗传图谱构建及QTL定位的基础知识点 7319 浏览
- 构建遗传图谱---作图群体如何选择? 9868 浏览
- 遗传图不同群体 3000 浏览
