GSEA法基因功能富集分析原理详解!
在组学大讲堂之前的一篇微信文章:关注的功能基因集在转录组结果中表现如何?中,跟大家介绍了GSEA(Gene Set Enrichment Analysis)及其分析结果。GSEA是一种基于基因集的富集分析方法,在对基因表达数据分析时,首先确定分析的目的,即选择MSigDB中的一个或多个功能基因集进行分析,然后基于基因表达数据与表型的关联度(也可以理解为表达量的变化)的大小进行排序。然后判断每个基因集内的基因是否富集于表型相关度排序后基因列表的上部或下部,从而判断此基因集内基因的协同变化对表型变化的影响。以上其实就是GSEA的分析原理,光看文字大家是不是还是很疑惑?下面我们就借助一张图来帮助大家更好的理解GSEA的分析原理。
GSEA分析原理:
第一步 基因排序:
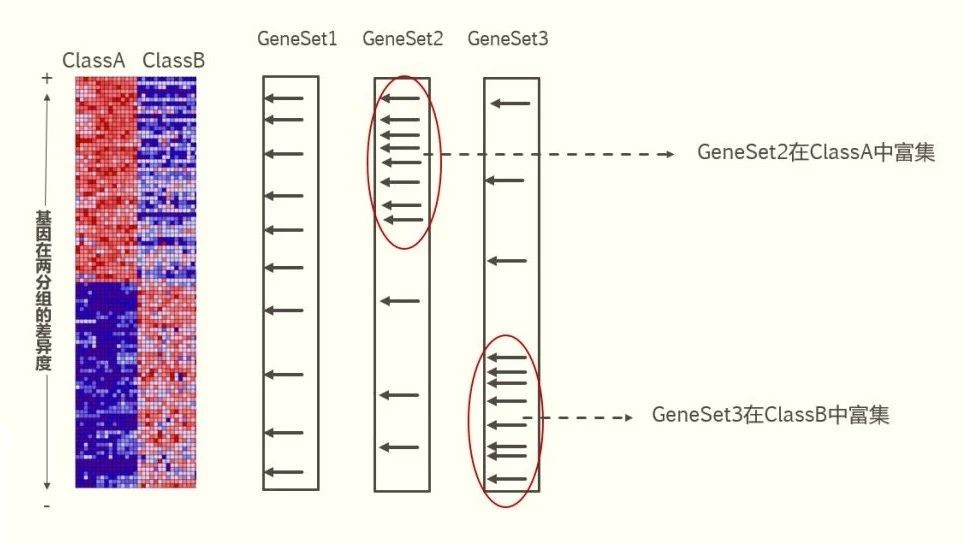
如上图左边的热图所示,GSEA分析的第一步就是利用所有基因的表达数据,然后计算每个基因在两个分组(或者表型)ClassA、ClassB中的差异度(GSEA提供了6种算法,默认方法是signal2 noise,GSEA官网有提供公式),然后按照在两个表型种的差异度从大到小排序,形成一个排好序的基因列表。
第二步 分析基因集是否富集:
这里的基因集,是事先根据功能或者其他的一些原理把很多的基因分类成不同的基因集合,具体一个基因集可以是某一个通路或者go term中的所有基因,也可以是一个miRNA靶标对应的多个基因。GSEA提供了多个分类基因集,在分析数据时,只需要选择不同基因集就可以,当然也可以自己制作基因集。我们可以对每一个小的基因集(GeneSet )里面的基因对应一下上一步排序表里面的位置,例如上图中的GeneSet1 (一个箭头代表一个基因),看基因集里的成员在基因列表里面的分布情况是否均匀,例如GeneSet1就在基因列表中均匀分布,GeneSet2里面的成员主要分布在基因列表的顶部,GeneSet3里面的成员主要分布在基因列表的底部。如果基因集中的成员在基因列表中均匀分布,说明这个基因集不在这两个表型中富集。如果基因集中的成员在基因列表的顶端例如图中的GeneSet2,说明这个基因集在第一个表型ClassA中富集。如果基因集中的成员在基因列表的底部例如图中GeneSet3,说明这个基因集在第二个表型ClassB中富集。
GSEA分析的第三步就是计算基因集的ES值(enrichment score),之后对基因集的ES值进行显著性检验及多重假设检验,从而计算出显著富集的基因集。
以上就是GSEA的分析原理,那么进行GSEA的结果怎样解读呢?
GSEA分析结果最常见的是下图:
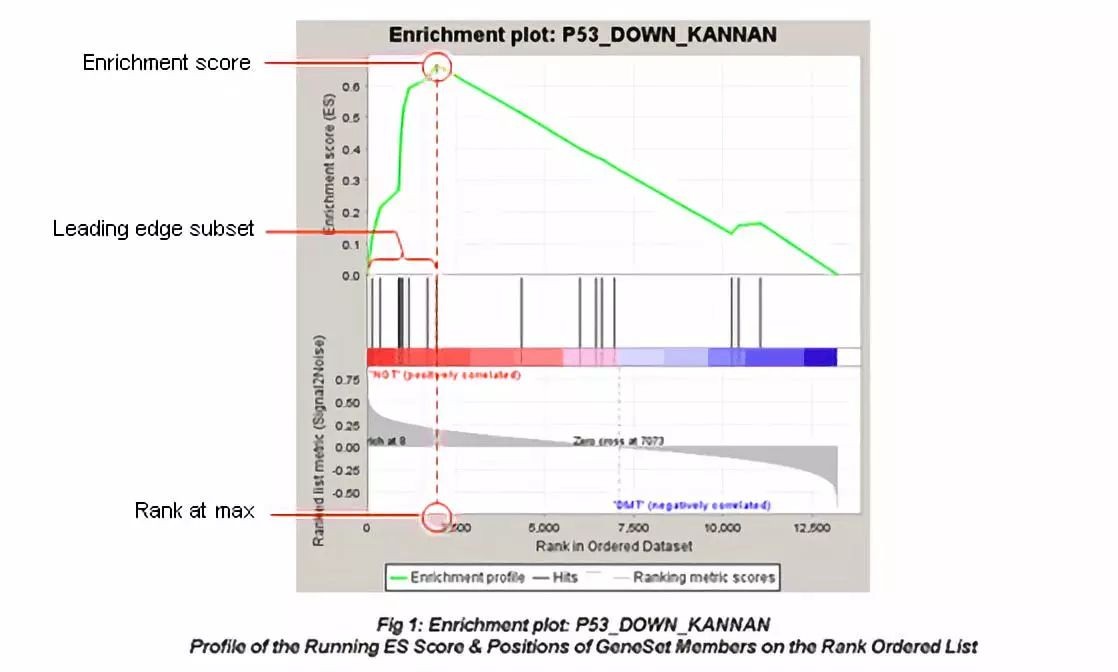
1、图最上面部分展示的是富集分数(ES, enrichment score)值计算过程,从左至右每到一个基因,计算出一个ES值,连成线。在最左侧或最右侧有一个特别明显的峰值就是基因集表型上的ES值。图中间部分每一条线代表基因集中的一个基因,及其在基因列表中的排序位置。
2、最下面部分展示的是基因与表型关联的矩阵,红色为与第一个表型(class A)正相关,在class A中表达高,蓝色与第二个表型(class B)正相关,在class B中表达高。
3、Leading-edge subset 对富集得分贡献最大的基因成员。若富集得分为正值,则是峰左侧的基因;若富集得分为负值,则是峰右侧的基因。
4、FDR GSEA默认提供所有的分析结果,并且设定FDR<0.25为可信的富集,最可能获得有功能研究价值的结果。但如果样品数目少,而且选择了gene_set作为Permumation type则需要使用更为严格的标准,比如FDR<0.05。
GSEA的优势
与常规基于超几何检验的基因功能富集分析方法相比,GSEA分析有如下的优点:
1. 不需要对基因进行差异显著的筛选,这样能保留那些表达变化不大,但是功能重要的基因,而传统的GO和KEGG富集分析是针对有差异的基因进行富集分析,相比之下GSEA分析保留了更多信息。举个例子:这里我们找到了两个基因在细胞增值通路里有显著差异,同时呢,也找到了两个基因在细胞凋亡通路里有显著差异,这时候用传统的方法无法确认我们研究的细胞表型是和凋亡还是和增值相关?这时候GSEA分析的优势来了,我们用GESA进行富集分析发现凋亡通路里面的基因除了那两个显著差异的基因表达显著上升外其他的相关基因也有整体的上升,而增值通路里面的基因就没有这种现象,因此我们就可以确定我们研究的表型是和凋亡相关的。
2. 分析的是基因集而不是单个的基因,因为生物体要出现表型差异,要找到与表型差异相关的基因,单单通过差异分析是不够的,有时候甚至得到假阳性的结果,因为生物体出现某种表型(一两个基因表达存在差异)往往会有一系列与之相关的上游或者下游的基因发生变化,但不一定会有显著差异,因此我们对功能相关的基因作为一个整体做GSEA分析,比较集合中基因整体的表达量差异得到的分析结果更可靠。
3.目前GSEA提供的功能基因分类数据库有以下8种,主要是与人类基因相关的分类数据库MSigDB,动植物目前没有,所以GSEA的分析方法大多在人类相关的研究中应用。但是如果你可以自行按照GSEA官方说明制作基因功能分类数据库,就可以应用到任何动植物了。

更多生物信息课程:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程、基因家族文献思路解读
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘、转录组文献解读
5. 微生物16S/ITS/18S分析原理及结果解读、OTU网络图绘制、cytoscape与网络图绘制课程
6. 生物信息入门到精通必修基础课:linux系统使用、perl入门到精通、perl语言高级、R语言入门、R语言画图
7. 医学相关数据挖掘课程,不用做实验也能发文章:TCGA-差异基因分析、GEO芯片数据挖掘、GEO芯片数据标准化、GSEA富集分析课程、TCGA临床数据生存分析、TCGA-转录因子分析、TCGA-ceRNA调控网络分析
- 发表于 2018-06-25 18:16
- 阅读 ( 43402 )
- 分类:基因组学
你可能感兴趣的文章
- enrichGSEA_pip.r GSEA富集分析 3351 浏览
- 手把手教你使用GSEA分析! 10378 浏览
- 如何将GEO芯片注释文件整理成GSEA能使用的芯片注释文件? 9354 浏览
- GSEA分析需要的文件及格式 22667 浏览
- GSEA分析需要的基因集Molecular Signatures Database是什么? 10594 浏览
