Molecular Plant | 一个接近完整的拟南芥基因组组装
模式植物拟南芥的基因组很小,不超过150M,只有5对染色体,因此它也是最早实现全序列分析的植物,同时其基因组序列的测定极大地促进了植物分子生物学的研究。20多年后,拟南芥基因组TAIR10仍然存在165个缺口,但随着技术的发展,大家致力于实现没有gap的、端粒到端粒的基因组组装,因此拟南芥的基因组也进行了更新。目前已经发表的高完整度拟南芥基因组有XJTU和CEN,填补了着丝粒等缺失区域。但这两个新的组装仍不完整且有相互矛盾之处。在这篇文章中,作者组装了一个高质量的基因组,包含三个无间隙的染色体和两个仅在核仁组织区有缺失的染色体,填补了在Col-CEN和Col-XJTU中的大多数剩余缺口。

作者首先根据ONT的一致性序列用nextdenovo得到初步的基因组,随后结合ONT、HiFi、illumina数据对基因组进行了优化,获得polished组装,此时一共有8个contig,按照长度排列为1-8,其中Contig1-4的双端和Contig5的一端含有拟南芥的端粒重复元件。NT比对将contig8归为叶绿体,随后被移除。随后在TAIR 10的框架内在染色体水平锚定重叠群,对Chr4进行重排后,利用HiFi组装的支架填充两个缺口。在最后基于ONT的比对纠正了区域中的结构错误和小的错误组装,然后使用HiFi重叠群和reads比对检查核仁组织区(NOR)中的潜在缺失。
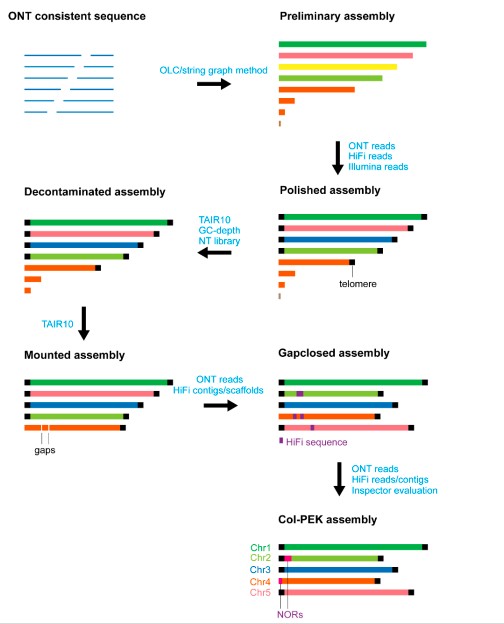
图1
与最近发布的高质量组装体(包括Col-CEN和Col-XJTU,图2 A)的比较表明,Col-PEK是高度完整的,长度更长,并且已经填充了长于40 kb的多个剩余缺口。例如Col-XJTU留下的Chr 2上的108.7-kb缺口被补全(图2 B)和Col-CEN中一个232.8kb的未知缺口现已被鉴定并填充在Chr 2(图2 C)。Col-PEK组装总长度133.92 Mb,比TAIR10组装长14.77 Mb。在填补缺口之外,Col-PEK还修订了Col-CEN等组装中的拼接错误。
作者基于转录组数据对TAIR 10构建了新的基因注释,发现了145个隐藏的重复基因。同时通过从头预测,同源性搜索和转录组组装三种方法在新序列上得到了68个基因。此外在基因组上一共识别到35M的重复序列,又利用LASTZ得到着丝粒特征重复CEN 180的集合,构成特异性motif文库,确定CEN 180存在的潜在范围。又通过Barrnap预测rDNA并采用BLASTn进行验证,得到了5S rRNA簇的密度分布。
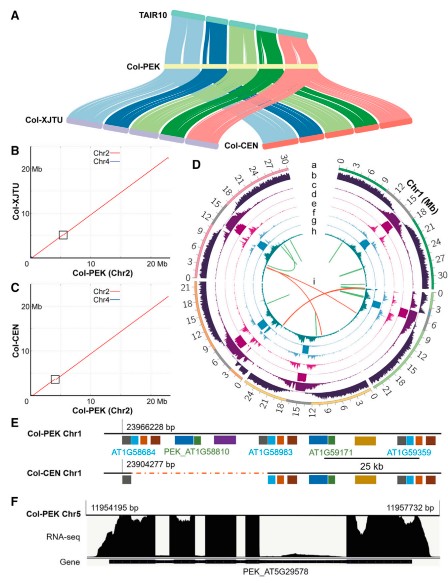
图2
Col-PEK 组装填补了包括五个着丝粒在内各区域中的绝大多数缺口,完成了1、3、5号染色体从端粒到端粒的完整组装,纠正了之前的错误组装。对其初步分析展示了重复序列的分布规律,并揭示了一批新基因。Col-PEK 参考基因组为国内外植物学科研工作者提供了新的参照序列和重要数据资源。
- 发表于 2023-10-12 16:26
- 阅读 ( 2327 )
- 分类:文献解读
