绘制HIC热图
绘制HIC热图
使用HiC-Pro和EndHiC软件可以实现以下HIC热图绘制。
一.运行HiC-Pro
文件准备:
1.准备index文件
bowtie2-build genome.final.fa genome.final
2.准备annotation文件
要有两个:
第一个是bed文件,通过软件包里面HiC-Pro-3.1.0/bin/utils/digest_genome.py脚本生成。
digest_genome.py -r mboi -o genome.final genome.final.fa #指定HIC数据使用的酶 mboi:GATC;dpnii:GATC ;bglii:AGATCT;hindiii:AAGCTT
文件格式如下:
chr2 0 579 HIC_chr2_1 0 +
chr2 579 1817 HIC_chr2_2 0 +
chr2 1817 3145 HIC_chr2_3 0 +
chr2 3145 3155 HIC_chr2_4 0 +
chr2 3155 3200 HIC_chr2_5 0 +
chr2 3200 3951 HIC_chr2_6 0 +
chr2 3951 4097 HIC_chr2_7 0 +
chr2 4097 4367 HIC_chr2_8 0 +
chr2 4367 4423 HIC_chr2_9 0 +
chr2 4423 4765 HIC_chr2_10 0 +
第二个是基因组每个常染色体长度文件,可以使用samtools建索引获取
samtools faidx genome.final.fa
cut -f 1,2 genome.final.fa.fai > genome.final.sizes
文件格式如下:
chr2 8484449
chr3 6619411
chr4 6348584
chr6 6552022
chr1 5951489
chr5 5110245
chr7 4434767
contig9 20021
contig12 13510
contig15 8861
3.HIC数据整理
HIC数据需要分级存放,创建data目录存放数据,在data目录下分别创建不同样品目录,原始数据分别放到不同样品目录下 。如下所示:
|-data
| |-sample1
| | |-sample1_R1.fastq.gz
| | |-sample1_R2.fastq.gz
| |-sample2
| | |-sample2_R1.fastq.gz
| | |-sample2_R2.fastq.gz
...
4.准备配置文件
配置文件模板可从HiC-Pro安装目录下复制,名称:config-hicpro.txt。也可直接从这里复制,文件内容放在最后。
配置文件需要修改的内容有:
1):BOWTIE2_IDX_PATH,bowtie2索引目录 (绝对路径)
2):REFERENCE_GENOME,基因组文件名称前缀(文件名称,去掉.fa或.fasta)
3):GENOME_SIZE,基因组染色体长度大小文件路径 (绝对路径)
4):GENOME_FRAGMENT,bed文件路径(绝对路径)
config-hicpro.txt
# Please change the variable settings below if necessary
#########################################################################
## Paths and Settings - Do not edit !
#########################################################################
TMP_DIR = tmp
LOGS_DIR = logs
BOWTIE2_OUTPUT_DIR = bowtie_results
MAPC_OUTPUT = hic_results
RAW_DIR = rawdata
#######################################################################
## SYSTEM AND SCHEDULER - Start Editing Here !!
#######################################################################
N_CPU = 2
SORT_RAM = 1000M
LOGFILE = hicpro.log
JOB_NAME =
JOB_MEM =
JOB_WALLTIME =
JOB_QUEUE =
JOB_MAIL =
#########################################################################
## Data
#########################################################################
PAIR1_EXT = _R1
PAIR2_EXT = _R2
#######################################################################
## Alignment options
#######################################################################
MIN_MAPQ = 10
BOWTIE2_IDX_PATH = /share/nas5/project/denovo/
BOWTIE2_GLOBAL_OPTIONS = --very-sensitive -L 30 --score-min L,-0.6,-0.2 --end-to-end --reorder
BOWTIE2_LOCAL_OPTIONS = --very-sensitive -L 20 --score-min L,-0.6,-0.2 --end-to-end --reorder
#######################################################################
## Annotation files
#######################################################################
REFERENCE_GENOME = genome.final
GENOME_SIZE = /share/nas5/project/denovo/genome.final.sizes
#######################################################################
## Allele specific analysis
#######################################################################
ALLELE_SPECIFIC_SNP =
#######################################################################
## Capture Hi-C analysis
#######################################################################
CAPTURE_TARGET =
REPORT_CAPTURE_REPORTER = 1
#######################################################################
## Digestion Hi-C
#######################################################################
GENOME_FRAGMENT = /share/nas5/project/denovo/genome.final.bed
LIGATION_SITE = AAGCTAGCTT
MIN_FRAG_SIZE = 100
MAX_FRAG_SIZE = 100000
MIN_INSERT_SIZE = 100
MAX_INSERT_SIZE = 600
#######################################################################
## Hi-C processing
#######################################################################
MIN_CIS_DIST =
GET_ALL_INTERACTION_CLASSES = 1
GET_PROCESS_SAM = 0
RM_SINGLETON = 1
RM_MULTI = 1
RM_DUP = 1
#######################################################################
## Contact Maps
#######################################################################
BIN_SIZE = 20000 40000 150000 500000 1000000
MATRIX_FORMAT = upper
#######################################################################
## Normalization
#######################################################################
MAX_ITER = 100
FILTER_LOW_COUNT_PERC = 0.02
FILTER_HIGH_COUNT_PERC = 0
EPS = 0.1
运行HiC-Pro:
HiC-Pro -i /share/nas5/project/denovo/data -c /share/nas5/project/denovo/config-hicpro.txt -o /share/nas5/project/denovo/hicpro_out
#-i:指定HIC数据存放目录; -c :指定配置文件路径; -o:指定输出目录
结果目录中找到 hicpro_out/hic_results/matrix/sample1/raw/20000/路径下的两个文件bed和matrix。
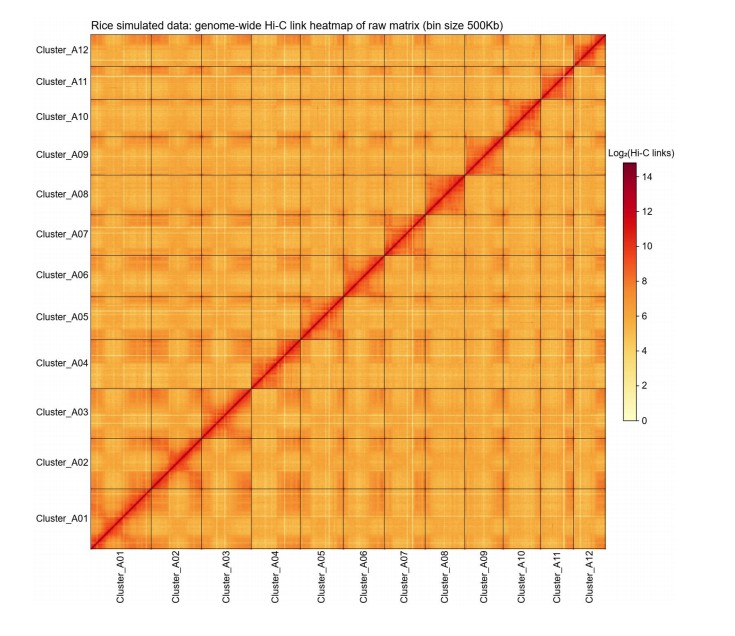
绘制HIC热图
使用EndHiC软件绘制。
matrix2heatmap.py sample1_20000_abs.bed sample1_20000.matrix
- 发表于 2023-10-25 10:42
- 阅读 ( 3295 )
- 分类:软件工具
你可能感兴趣的文章
相关问题
2 条评论
请先 登录 后评论
