TGS-gapcloser 安装与使用
基因组组装过程中会得到草图的scaffold,需要进一步进行gaps的关闭。TGS-gapcloser 是华大基因开发的,一个利用其他组装或是长度长测序数据对草图进行gap填充的一款软件。
基因组组装过程中会得到草图的scaffold,需要进一步进行gaps的关闭。TGS-gapcloser 是华大基因开发的,一个利用其他组装或是长度长测序数据对草图进行gap填充的一款软件。
1. 安装
下载
git clone https://github.com/BGI-Qingdao/TGS-GapCloser.git
如果已经安装了minimap2软件,在此次软链接一下软件地址
rm -rf TGS-GapCloser/minimap2
ln -s MINIMAP2所在文件夹 TGS-GapCloser/
编译
cd TGS-GapCloser/
make
编译时出现问题:
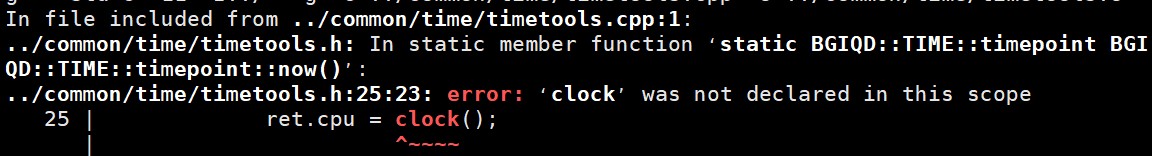
解决:
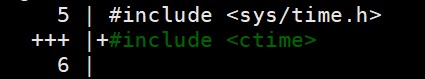
cd ./common/time
# 在timetools.h文件前面加上#include <ctime>
vi timetools.h
# 退回TGS-GapCloser/
cd ../../
make
2.使用
采用之前组装的fa文件对现在的scaffold补gap:
tgsgapcloser \
--scaff scaffold-path/scaffold.fasta \# 需要补gap的scaffold
--reads tgs-reads-path/tgs.reads.fasta \#之前组装的fa
--output test_ne \#输出前缀
--ne # 不需要进行polish
采用ONT数据补gap:
seqkit fq2fa ont.fastq.gz > ont.fasta
tgsgapcloser \
--scaff scaffold-path/scaffold.fasta \
--reads ont.fasta \ #ont数据转换的fa文件
--output test_racon \
--racon racon-path/bin/racon #指定racon地址
- 发表于 2023-10-30 17:19
- 阅读 ( 4411 )
- 分类:软件工具
你可能感兴趣的文章
相关问题
0 条评论
请先 登录 后评论
