一行代码检查GWAS分析中表型数据的错误
在进行GWAS分析时,如果表型数据出现了数字或NA以外的其他字符,在分析时就容易报错。
但是表型性状一般都信息非常多,肉眼难以检查出错误,那接下来带大家用一行代码来检查表型数据的错误。
随机构建一个表型数据如下:
<Trait> SC ZG YC YK FN LC LK sample1 19 117 22 6 16 8 6 sample2 16 111 NA 8 6 7 8 sample3 18 102 21 4 NA NA 8 sample4 21 98 16 4 5 14 4 sample5 16 99 NA 4 20 12 7 sample6 22 102 17 8 14 11 8 sample7 22 103 19 -7 20 5 6 sample8 21 120 19 8 11 NA 7 sample9 23 90 21 7 11 7 4 sample10 17 106 19 6 6 6 4 sample11 20 109 22 8 8 13 6 sample12 22 110 NA 4 14 7 8 sample13 NA 110 21 6 10 11 4 sample14 - 93 23 7 7 NA 4 sample15 20 112 18 5 NA 11 5 sample16 17 120 19 6 7 6 6 sample17 17 106 16 8 10 6 6 sample18 18 98 23 4 7 NA 7 sample19 NA 115 18 4 / 10 5 sample20 NA 117 22 5 8 8 5 sample21 21 93 22 wu 17 5 8 sample22 NA 108 NA 5 NA NA 6 sample23 NA 111 19 4 17 6 8 sample24 17 95 20 4 13 10 8 sample25 无 111 16 7 16 12 7 sample26 NA 94 18 6 10 8 7 sample27 16 117 20 6 14 NA 4 sample28 22 98 20 6 14 8 4 sample29 21 116 19 5 NA 7 5 sample30 NA 94 22 5 12 11 7
运行该代码
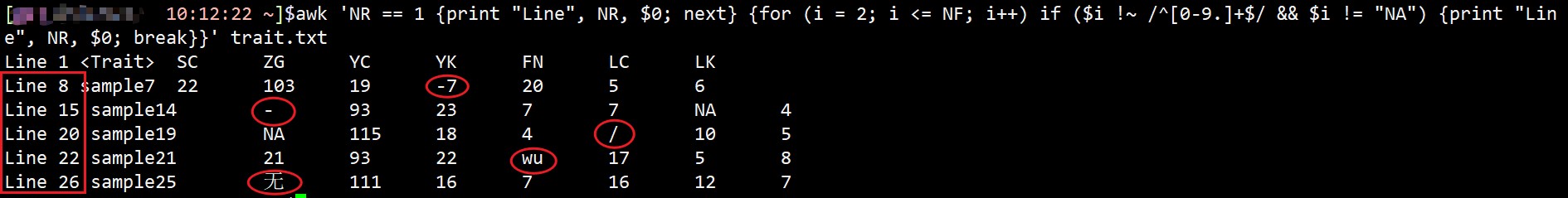
awk 'NR == 1 {print "Line", NR, $0; next} {for (i = 2; i <= NF; i++) if ($i !~ /^[0-9.]+$/ && $i != "NA") {print "Line", NR, $0; break}}' trait.txt
得到结果如下,会展示出表头及除了数字和NA之外的所有字符以及所在的行数,之后就可以根据这些出错的地方检查修改表型数据了。
- 发表于 2023-12-13 10:56
- 阅读 ( 1406 )
- 分类:软件工具
你可能感兴趣的文章
- AI 写代码之绘制GWAS 曼哈顿图 331 浏览
- TWAS分析的原理和工具 2287 浏览
- 如何从GWAS结果中获取表型解释率PVE——TASSEL、GEMMA篇 8911 浏览
- BLINK(C版本) 进行 GWAS分析的教程 4717 浏览
- 群体遗传进化与GWAS分析镜像更新2.0 1856 浏览
相关问题
- 全基因组关联分析GWAS教程中的beagle填充问题 1 回答
- GWAS分析的教程中06.impute的beagle填充出现问题 2 回答
- 老师您好,针对这个问题https://www.omicsclass.com/question/7725。你们提到:你这个数据里面是不是P值有0的情况,取了对数就无穷大了,导致出问题。但是,我发现里面没有P值为0的情况,那是不是脚本gwas_manhattan_plot.r有问题呢?需要修改gwas_manhattan_plot.r脚本的什么地方才可以解决顶部只能画半圆的问题呢?还有,我发现P-Value的原始文件有NaN的情况,是什么原因呢?是否对GWAS有影响呢? 1 回答
- GWAS 曼哈顿 Manhattan 图像问题 1 回答
- R语言GAPIT跑GWAS,ERROR求解:不能有负长度矢量,万分感谢!!! 1 回答
- R语言GAPIT跑GWAS错误 Error in ‘[.data.frame‘(GD,,!is.na(GI.MP[,3])):选择了未定义的列 0 回答
0 条评论
请先 登录 后评论
