NCBI 数据下载
介绍如何在大型生物数据库NCBI上下载数据
已发表的文章的原始数据往往会公布出来,大家可以基于原始数据进行信息深挖,结合自己的生物学问题也可以撰写出不错的文章。
这些公开的数据大部分会上传到大型数据库,少部分会进行自己网站的构建后放在自己的网站上。今天主要是介绍如何在大型生物数据库NCBI上下载数据。
1. 直接下载:数据量小于5G的时候适用
① 检索:搜索一个sra号,进入RUN的详情页
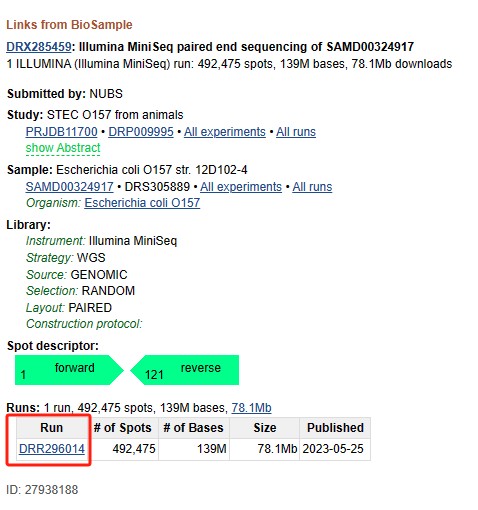
② 下载到本地:选择download一栏会直接下载到本地,不想下载到本地请看③
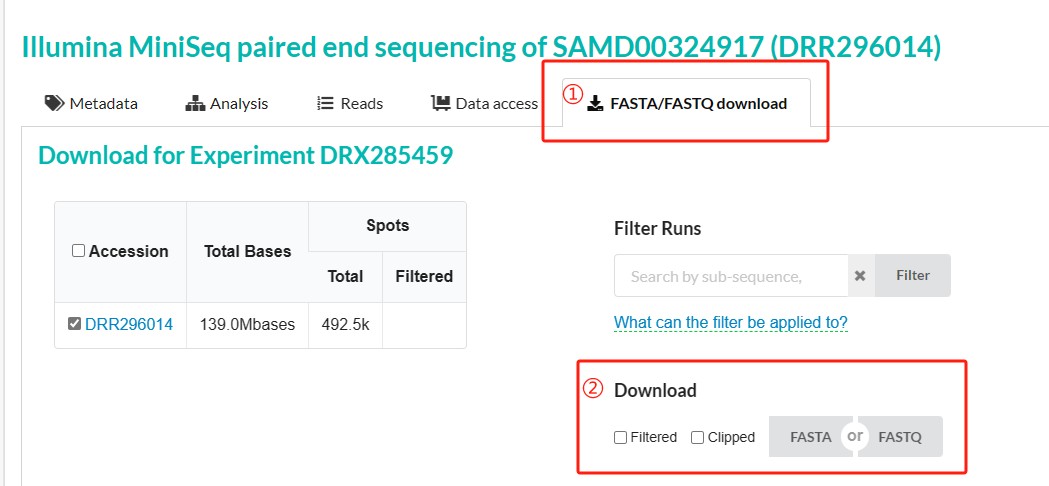
③下载到服务器:复制下载链接到linux上下载
wget -c -b https://www.be-md.ncbi.nlm.nih.gov/Traces/sra-reads-be/fastq?acc=DRR296014
2. 利用软件:SRA Toolkit 和 Aspera
(1) SRA Toolkit
① 单个下载:指定Run编号下载
prefetch DRR296014
② 批量下载 :先在NCBI下载所有run的信息
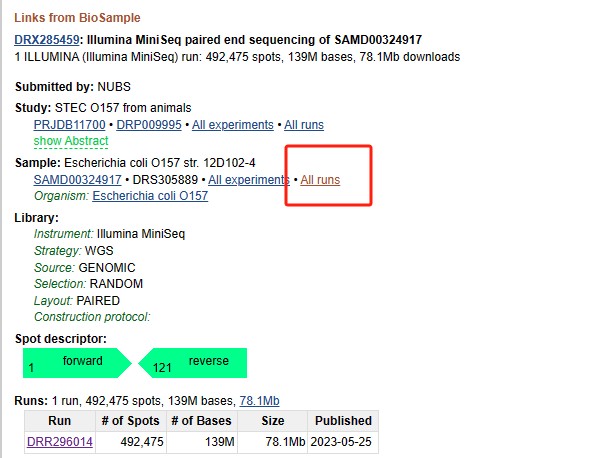
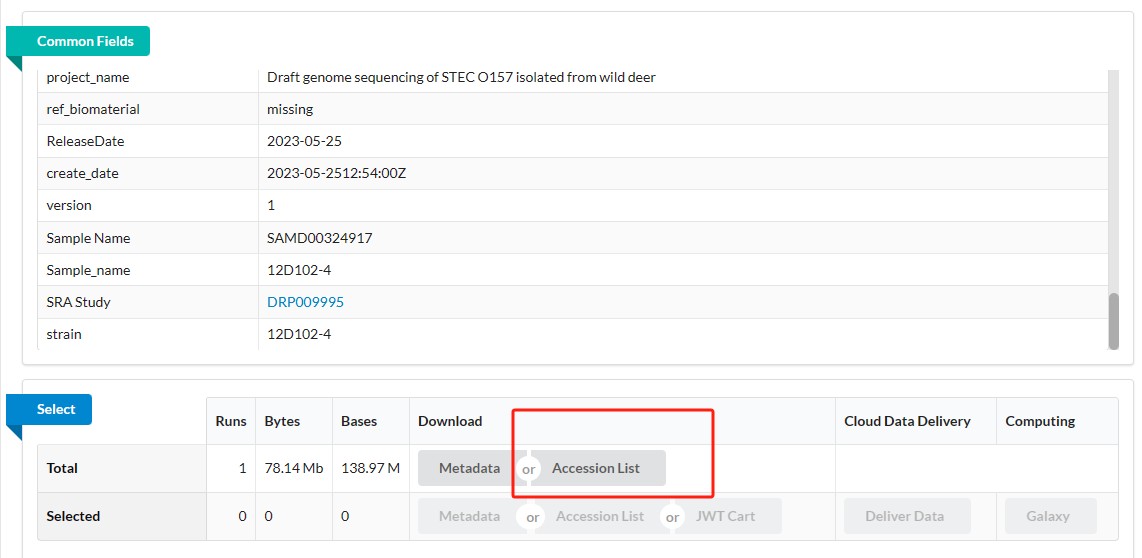
执行命令:
prefetch --option-file SRR_Acc_List.txt
③ 格式转换:sra变成fq
# 双端测序:
fastq-dump --split-3 sra文件
# 单端测序:
fastq-dump sra文件
更快的转换方式:
fasterq-dump --split-3 sra文件
(2) Aspera
直接下载,需要去除前面的域名,用如下命令下载,注意最后有一个点.,表示文件下载到当前目录并且保持原文件名
ascp -i ~/.aspera/connect/etc/asperaweb_id_dsa.openssh -l 100M -k 1 -T anonftp@ftp.ncbi.nlm.nih.gov:/refseq/release/viral/viral.2.1.genomic.fna.gz .
参考:
SRA Toolkit - prefetch 快速下载NCBI SRA数据 - 简书 (jianshu.com)
- 发表于 2023-12-18 11:25
- 阅读 ( 3795 )
- 分类:其他
你可能感兴趣的文章
相关问题
0 条评论
请先 登录 后评论
