微生物与环境因子,代谢物,功能等相关性分析linkET
微生物与环境因子,代谢物,功能等相关性分析linkET
https://github.com/Hy4m/linkET
示例代码:
library(linkET)
library(ggplot2)
library(dplyr)
#读取示例数据
#micro <- read.delim('微生物数据.txt', row.names = 1)
#env <- read.delim('环境数据.txt', row.names = 1)
data("varechem", package = "vegan")
data("varespec", package = "vegan")
#计算 Mantel 相关性
#通过 spec_select 指定数据组范围,例如这个示例数据中,微生物矩阵的第1-22列是物种丰度数据(指定名称 Taxonomy),
#第23-40列是基因丰度数据(指定名称 Function)
#默认情况下,对 spec 计算 Bray-Curtis 距离,对 env 计算 Euclidean 距离,然后计算二者 Mantel 相关
mantel <- mantel_test(varespec, varechem,
spec_select = list(Spec01 = 1:7,
Spec02 = 8:18,
Spec03 = 19:37,
Spec04 = 38:44) ,
mantel_fun = 'mantel')
#根据相关系数和显著性设置标签,以便作图时定义线宽和颜色
mantel <- mutate(mantel,
rd = cut(r, breaks = c(-Inf, 0.2, 0.4, Inf), labels = c('< 0.2', '0.2 - 0.4', '>= 0.4')),
pd = cut(p, breaks = c(-Inf, 0.01, 0.05, Inf), labels = c('< 0.01', '0.01 - 0.05', '>= 0.05'))
)
mantel
#绘制相关图
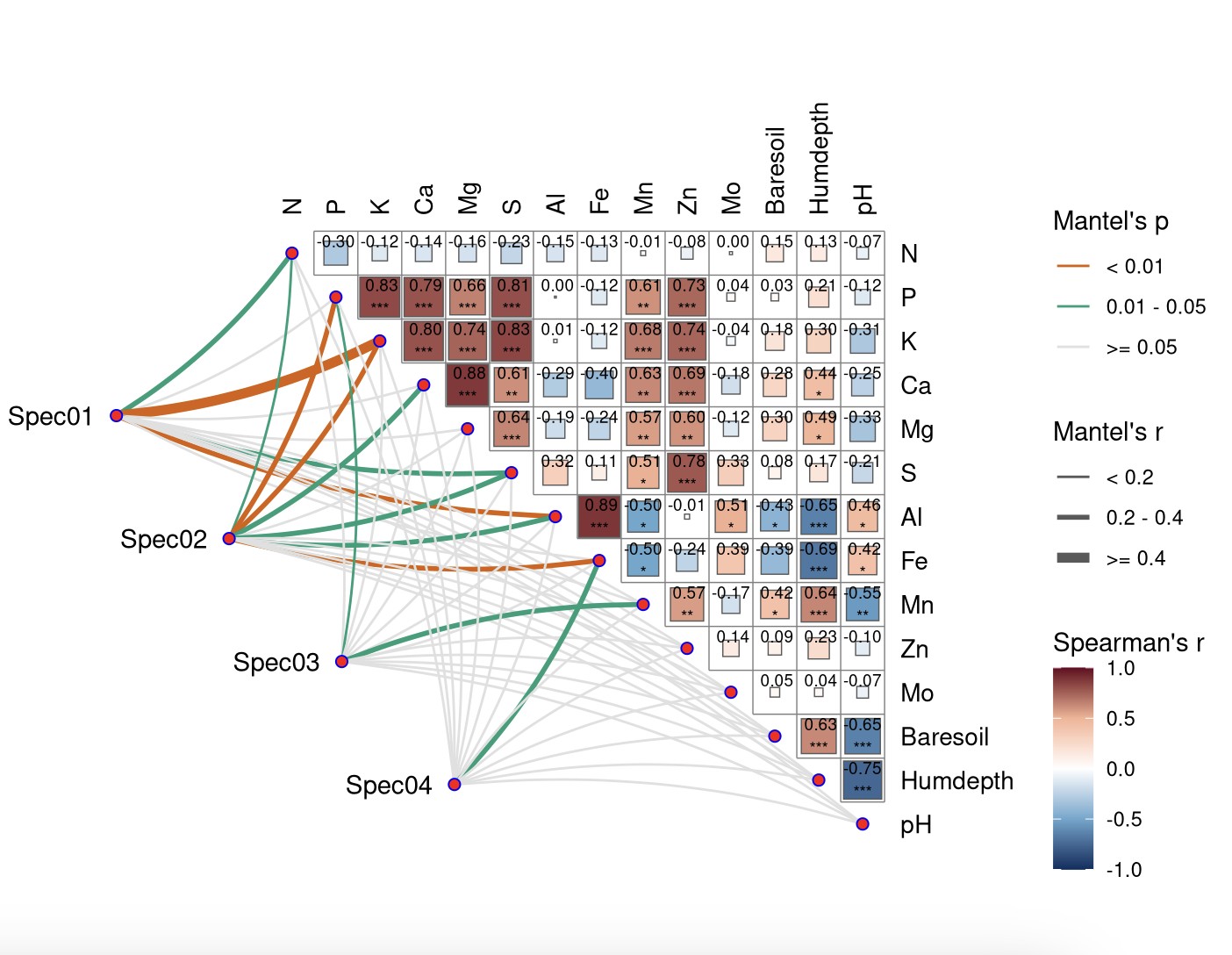
qcorrplot(correlate(varechem, method = 'spearman'), type = 'upper', diag = FALSE) + #环境变量矩阵计算 Spearman 相关系数
geom_square() + #绘制 Spearman 相关系数热图
geom_mark(sep = '\n', size = 2.5, sig.thres = 0.05) + #显示 Spearman 相关系数和显著性
geom_couple(aes(color = pd, size = rd), data = mantel, curvature = nice_curvature()) + #环境和微生物的相关性展示为上述 Mantel 相关
scale_fill_gradientn(colors = c('#053061', '#68A8CF', 'white', '#F7B394', '#67001F'), limits = c(-1, 1)) + #根据 Spearman 相关指定热图颜色
scale_size_manual(values = c(0.5, 1, 2)) + #根据 Mantel 相关指定线条粗细
scale_color_manual(values = c('#D95F02', '#1B9E77', '#E0E0E0')) + #根据 Mantel 相关 p 值指定线条颜色
guides(color = guide_legend(title = "Mantel's p", order = 1), #图例标题和排序
size = guide_legend(title = "Mantel's r", order = 2),
fill = guide_colorbar(title = "Spearman's r", order = 3)) +
theme(legend.key = element_blank())
- 发表于 2023-12-19 08:20
- 阅读 ( 3187 )
- 分类:宏基因组
你可能感兴趣的文章
- 宏基因组结果解读和个性化分析课程上架 1478 浏览
- 紧扣前沿热点--多种研究培训,总有你需要的! 5936 浏览
- 微生物多样性扩增子(16S/18S/ITS)课程更新v2.0 2691 浏览
- PICRUSt2扩增子功能预测与差异比较分析 6221 浏览
- 宏病毒组分析一区 1657 浏览
0 条评论
请先 登录 后评论
