cytoscape软件的安装与使用_v3.10.2
一、简介
Cytoscape 是一个专注于开源网络可视化和分析的软件。它的核心是提供基础的功能布局和查询网络,并依据基本的数据的结合成可视化网络。
Cytoscape 源自系统生物学,用于将生物分子交互网络与高通量基因表达数据和其他的分子状态信息整合在一起,其最强大的功能还是用于大规模蛋白质-蛋白质相互作用、蛋白质-DNA和遗传交互作用的分析。
通过Cytoscape,可以在可视化的环境下将这些生物网络跟基因表达、基因型等各种分子状态信息整合在一起,还能将这些网络跟功能注释数据库链接在一起。
Cytoscape 的核心是网络,简单的网络图包括节点(node)和边(edge),每个节点可以是基因、miNRA或蛋白质等等;节点与节点之间的连接 (edge) 代表着这些节点之间的相互作用,包括蛋白与蛋白相互作用(pp),DNA与蛋白相互作用(pd)等。
二、Cytoscape安装
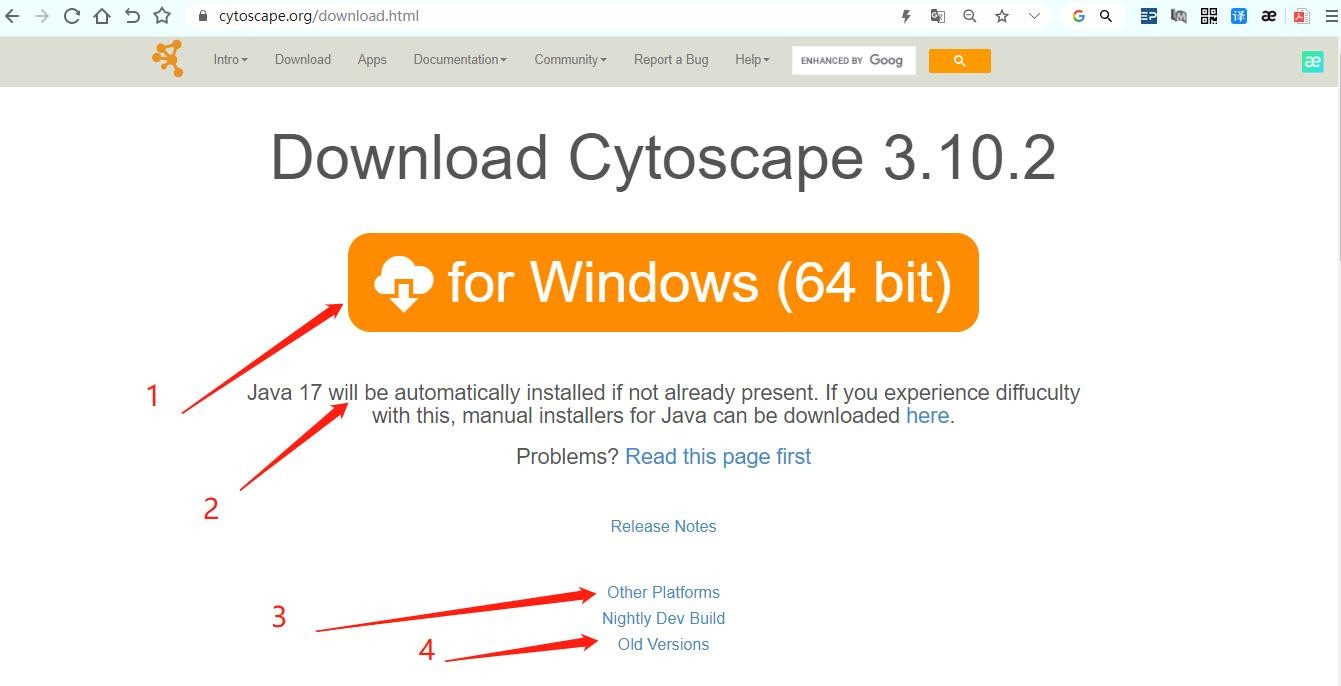
1. 软件下载地址:https://cytoscape.org/ 最新版本3.10.2。下载安装非常简捷,进入网址点击“Download 3.10.2”直接下载即可。
2. Cytoscape安装需要配置Java环境,不过不用担心,现在安装cytoscape软件过程中会默认下载相应版本的java工具"Java 17"。
3. 想要下载其他操作系统的例如linux、Mac OS系统的可以点击“Other Platforms”下载
4. 想要下载旧版本的可以点击“Old Versions”下载,注意配套JAVA的版本。
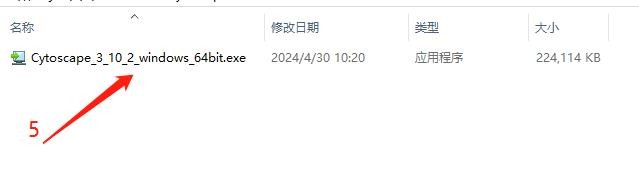
5. 下载之后在本地电脑中得到“Cytoscape_3_10_2_windows_64bit.exe”的应用程序,直接点击安装即可,在选择安装路径时放在全英文路径下。
6. 安装好之后点击“Cytoscape.exe”运行即可。

三、Cytoscape插件的安装
Cytoscape是一个功能强大的网络可视化和分析软件,它提供了许多插件和扩展功能,用于增强其功能和适应不同的研究需求。大家可以根据自己的分析需求进行配置。
1. 插件的下载需要到App Store里面下载。点击菜单栏 -> “Apps” -> “App Store” -> “show App Store” 即可弹出插件面板。或者直接点击最左侧“App Store”也一样。
2. 在插件面板中搜索自己想要下载的插件,输入关键字然后回车,进入网页版Cytoscape App Store。
3. 红框框起来的是安装软件时默认下载好的插件和已经下载好的插件。
4. 进入网页版Cytoscape App Store。选择自己想下载的插件,进入插件介绍页面,点击右侧“Install”即可下载。之后即可在自己软件App Store中查看到并使用


另外以下是一些常用的Cytoscape插件和扩展功能:
cytoHubba插件:cytoHubba是一个用于识别关键基因和子网络的插件。它基于拓扑网络算法,可以根据节点在网络中的属性进行排名,并提供多种拓扑分析方法,如Degree、Betweenness、Closeness等。
BiNGO插件:BiNGO是一个用于基因本体分析的插件。它可以将基因列表与基因本体数据库进行比较,帮助用户了解基因在功能和过程上的富集情况,从而揭示基因的生物学意义。
clusterMaker2插件:clusterMaker2是一个用于聚类和可视化分析的插件。它提供了多种聚类算法和可视化布局算法,可以帮助用户发现网络中的模块和群集结构。
MCODE插件:MCODE是一个用于模块发现的插件。它可以识别网络中的高度相互关联的节点群集,并根据节点的连接密度和节点数目进行评分,从而帮助用户发现功能相关的模块。
iRegulon插件:iRegulon是一个用于转录因子调控网络分析的插件。它可以根据转录因子结合位点的富集情况和转录因子的调控模式,预测转录因子在给定基因集中的调控活性,并生成转录因子调控网络图。
KEGGscape插件:KEGGscape是一个用于KEGG通路分析的插件。它可以将基因列表映射到KEGG通路,并提供丰富的可视化功能,帮助用户理解基因在通路中的功能和相互作用。
StringApp插件:StringApp是一个用于蛋白质相互作用网络分析的插件。它可以将基因列表映射到STRING数据库中的蛋白质相互作用网络,并提供丰富的网络分析和可视化功能。
四、Cytoscape软件的一些应用
1. 基因家族蛋白互作网络分析
2. 泛基因家族共表达网络分析
3. 富集分析中的通路注释网络图
4. ceRNA调控网络分析
5. 微生物OTU网络图
- 发表于 2024-04-30 17:41
- 阅读 ( 4889 )
- 分类:软件工具
你可能感兴趣的文章
- cytoscape-cytoHubba插件的安装与使用 22006 浏览
- cytoscape软件安装与使用-绘制蛋白互作网络图 7806 浏览
- Cytoscape如何基于点的属性快速对点和相连的线进行颜色分类 17162 浏览
- 网络概念以及Cytoscape导入数据格式理解(ceRNA等多元关系网络数据) 12167 浏览
- cytoscape插件BinGO安装以及GO富集分析和网络可视化 292175 浏览
