monocle3分析结果解读
Monocle3背景简述
在外界刺激或者发育过程中,细胞会在特定条件下进行分化,从一个状态过渡到另一个状态,表达完全不同的基因集,有的基因被沉默,而有的基因被激活。这些状态通常很难表征,因为想要纯化中间状态的细胞很困难或不可能。但是通过单细胞测序技术捕获一群细胞的转录快照,可以使用轨迹推断(trajectory inference)的方法,对测序的细胞根据表达模式的相似性进行排序。由此一来就可以模拟细胞的动态变化。可能有的小伙伴有疑问了,为什么叫做伪时序分析呢?如果想要得到细胞分化轨迹,分化的时序分析,理论上的做法是设置多个时间点,每个时间点取一次样,进行测序以及后续的分析。但是这种方法很难做,而且费时费力费钱。通过单细胞测序可以从一群细胞中得到转录瞬时快照,可以从中推断出细胞的分化轨迹。这种方法因为只取了一个时间点,所以叫做伪时序分析。
拟时序分析与Monocle3软件介绍
Monocle按照细胞的转录本丰度信息进行排序, 模拟细胞的分化轨迹. Monocle对比之前的算法, 其优点在于不依赖先验知识(前人证明的分化轨迹的biomaker). 而是采用无监督的方法,最大化连续模拟细胞之间的转录相似性。相比以往熟知的Monocle2,Monocle 3经过了重新设计,使之可以用于分析大型、复杂的单细胞数据集。Monocle 3的核心算法具有高度可扩展性,可以处理数百万个细胞。软件主要可以执行三类分析:1)细胞降维聚类、分群和计数,当然,Monocle3也可以直接导入Seurat的降维结果进行分析 ;2)构建单细胞轨迹;3)差异表达分析
分析结果介绍:
1.降维聚类分群分析
Monocle有自己的一套降维聚类分群分析方法,分析完之后和seurat的降维分群分析结果会有差异,为与Seurat结果保持一致推荐使用seurat分析好的UMAP结果展示分群信息及后续拟时序分析:

2. 将细胞划分为不连续的轨迹
细胞的分化总是具有连续性和离散性的。当数据量很大时,Monocle可能会错误地把本来不在同一个轨迹上的细胞认为是在同一个轨迹上,而monocle3可以把细胞划分为不同的分化轨迹,也就是partition,以下结果划分了4个partition
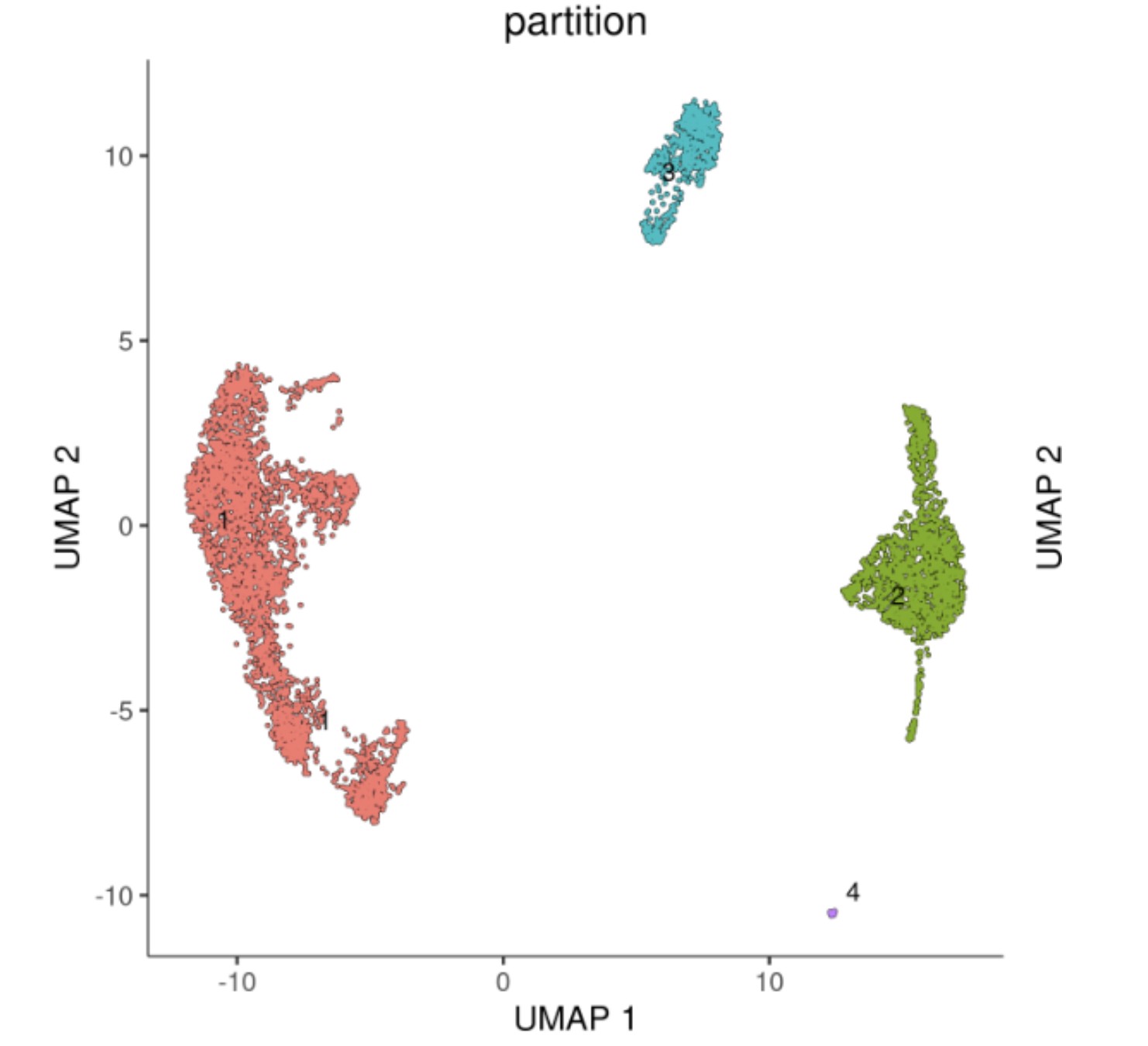
3. 细胞分化轨迹分析
Monocle3在相同的低维空间进行轨迹图学习,来表示细胞在发育过程中可能采取的路径。对此,Monocle3使用了一个基于SimplePPT算法的图嵌入过程。另外,Monocle3还有几点升级:1)直接在UMAP空间(默认为三维的)中绘制轨迹主图,避免直接处理成千上万个单细胞数据;2)能够平滑和细化主图以排除小分支,从而消除噪声分支;3)适用于环路轨迹,而不要求分化轨迹一定是树形结构。

注:黑色的线显示的是graph的结构。数字带灰色圆圈表示不同的结局,也就是叶子。数字带黑色圆圈代表分叉点,从这个点开始,细胞可以有多个结局。这些数字可以通过label_leaves和label_branch_points参数设置。
4. 计算伪时间
为了计算细胞级别的伪时间,Monocle3开发了一种适用于具有数百万细胞数据集的投影策略。该策略的原理是,以主图为指导,在所有单元上构造一个图ψ,然后通过计算每个细胞与选择的一个根或多个根的距离作为伪时间。Monocle3是一个半监督的拟时序分析,因此需要通过生物学背景来选择合适的起始点。
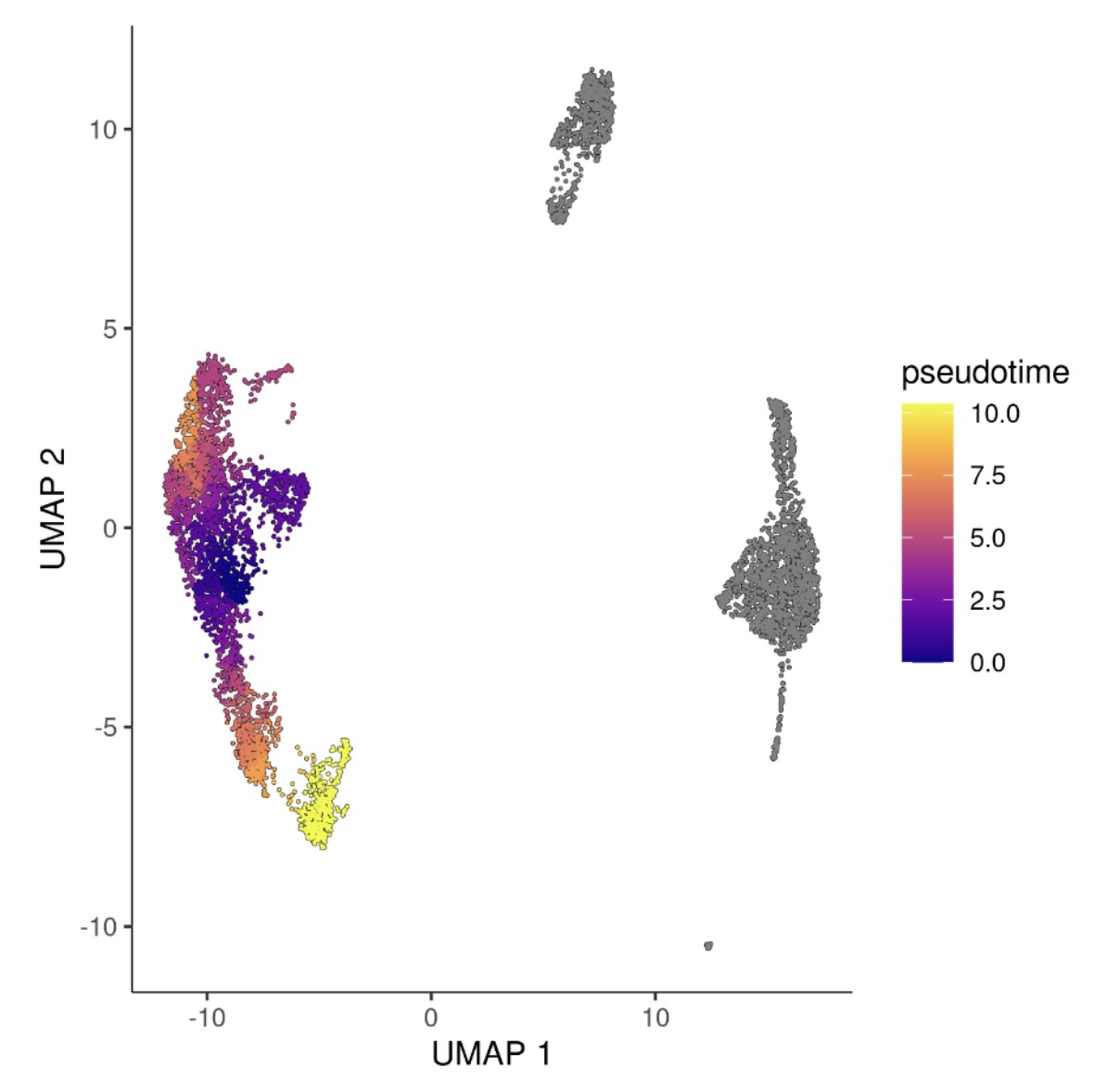
注:颜色由紫色到黄色,表示分化的时间由早及晚。数字表示分化的过程,数字越小,表示分化的时间越早。另外,如果自己指定的起始点,无法直接访问到某一部分细胞,这部分细胞就有无穷伪时间,用灰色表示。
5. 识别差异表达基因
为了识别在发育轨迹中表达变化不同的基因,Monocle3运用了一种常用于分析空间数据的统计检验。Moran’s I统计量(莫兰指数)是一个多向和多维空间自相关的度量。该统计量通过最近邻图编码数据点之间的空间关系,使其特别适合于分析大型单细胞转录组数据集。Moran指数的范围在-1~1之间,0代表此基因没有空间共表达效应,1代表此基因在空间距离相近的细胞中表达值高度相似,小于0的Moran指数一般都没有统计学意义。

注:每个点的颜色表示不同的细胞群,横轴从左到右表示时间的早晚。从图中可以看出基因随时间发展的表达量变化。
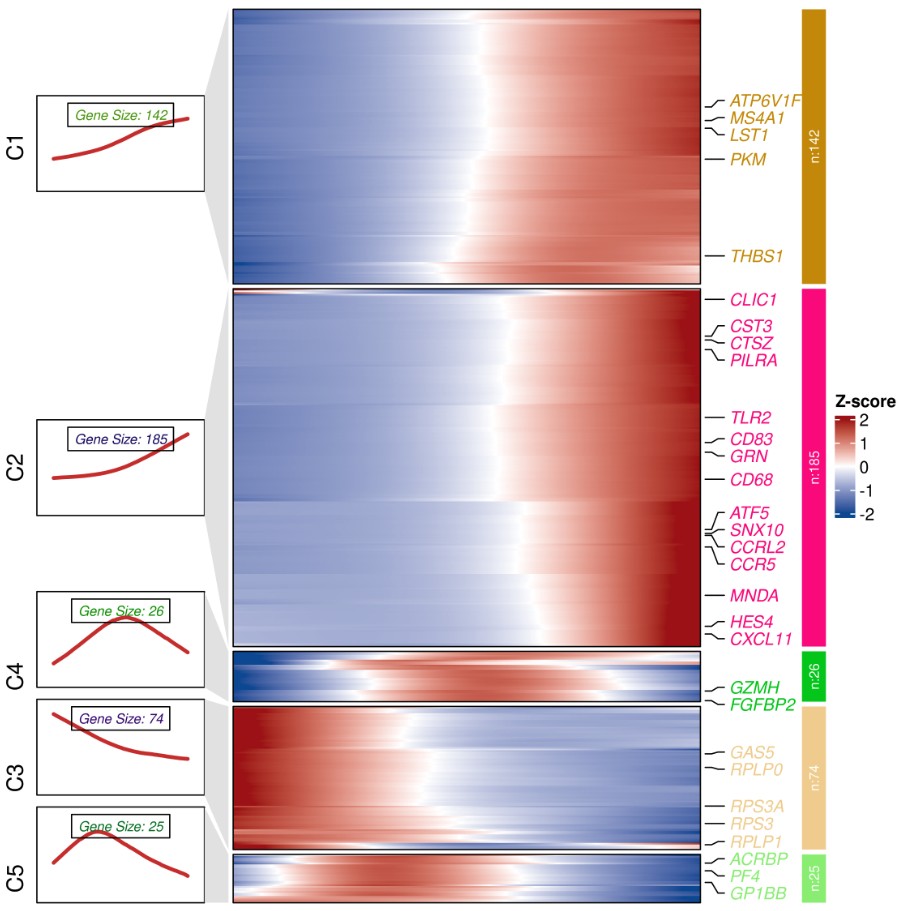
6 差异基因模块划分与共表达分析
哪些模块在不同的细胞系中高表达:寻找共表达模块Module,就可以说是这个模块导致他们状态发生变化,然后查看一个模块里的基因,再讲一些东西

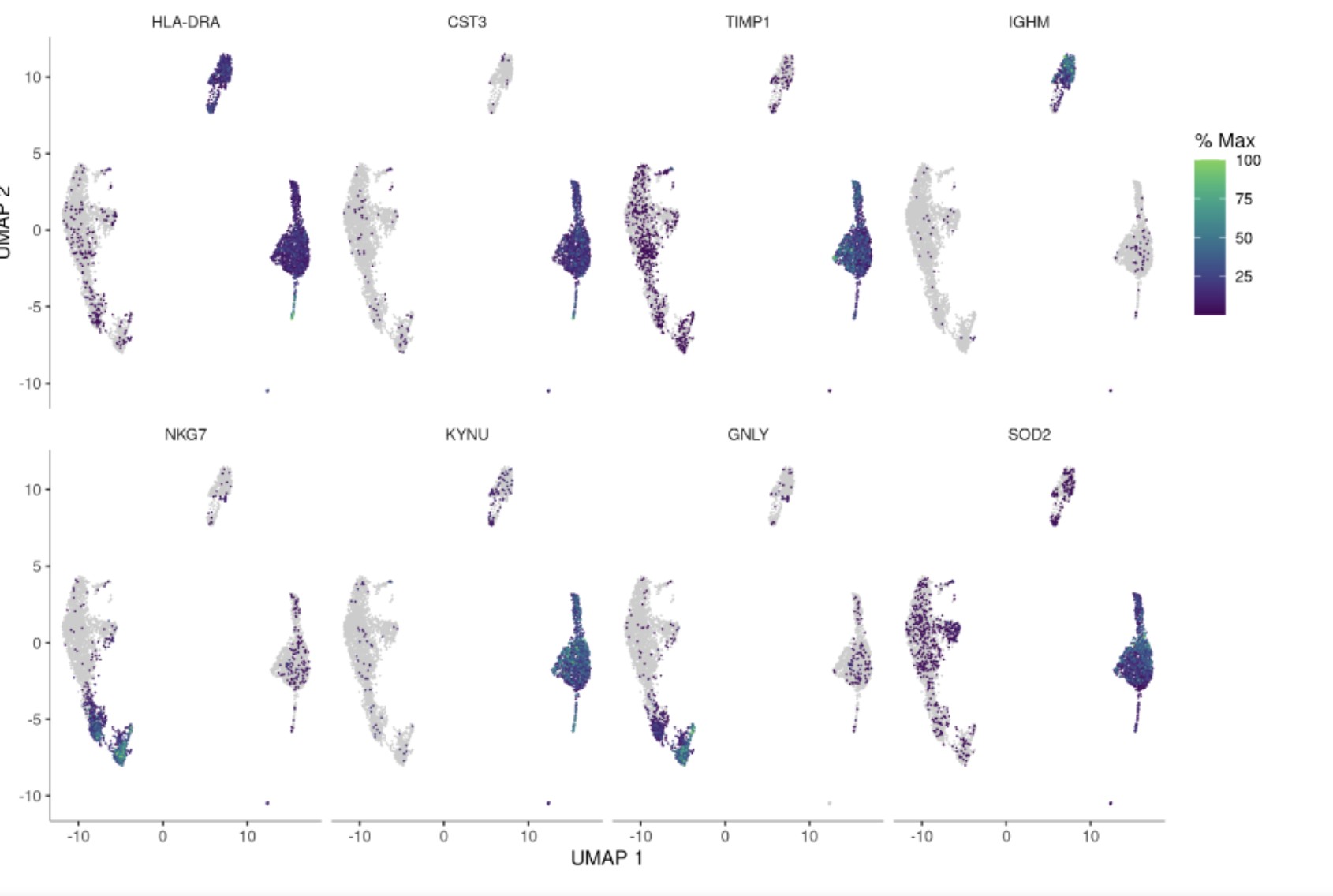
7.关键基因表达可视化
对于感兴趣的基因可以单独展示umap图中,颜色越浅表示越score值高越重要:score_significant
8.输出monocle2 类似结果并聚类
差异表达的基因表达模式聚类分群展示
单细胞转录组视频课程推荐:

- 发表于 2024-06-12 17:08
- 阅读 ( 8933 )
- 分类:转录组
你可能感兴趣的文章
- 植物单细胞转录组文章 1378 浏览
- 拟南芥叶单细胞分析流程 1390 浏览
- 单细胞转录组镜像代码更新4.0 2041 浏览
- 单细胞转录组数据挖掘流程记录-头颈癌(HNSCC)(GSE181919) 2192 浏览
- 非负矩阵分解(cNMF) 3739 浏览
- 单细胞转录组如何将UCell CytoTRACE2 infercnv等结果添加到总的rds中 1652 浏览
相关问题
- 本地镜像代码运行不了 1 回答
- 老师,拟时序基础初步分析时,运行以下代码时Rscript $scripts/cell_trajectory_monocle3.r --rds ../04.cell_type_ann_leaf/leaf.added.celltype.qs \ --use.seurat.umap --groupby mycelltype -o monocle3.1 --cpu 6,出现图中报错,是因为聚类分群的时候resolution值太低了吗 2 回答
- 拟时分析筛选出的差异基因数量太少,有没有可以调整的参数去增加筛选数量 1 回答
- 单细胞测序使用subset脚本提出子集,再进行拟时分析报错plot_genes_by_group 1 回答
- 使用monocle3进行轨迹分析报错 2 回答
- monocle3 trajectory analysis 1 回答
