单细胞RNA速率(RNA velocity)分析结果解读
RNA速率分析细胞发育轨迹的方法相对于monocle3的分析方法更具优势,因为RNA速率直接从基因的转录剪切层面出发评估基因的转录与表达水平,进而推断细胞发育轨迹,方法更具优势。下面介绍其分析原理与结果解读:
基因启动表达会出现未剪切mRNA的增加,而相反转录的抑制或缺失导致未剪切mRNA的减少。因此,通过未剪切的mRNA与成熟的剪切mRNA变化,就可以得到RNA速率。不同的细胞中相同基因的mRNA的RNA速率不同,可以用来估计细胞的未来状态。作者找了小鼠的肝脏细胞分不同时间点做转录组分析,并看一下昼夜周期节律相关的基因,发现剪切的mRNA和未剪切的mRNA量的确随时间发生周期变化,因此作者证明RNA速率可以用于推断细胞发育轨迹。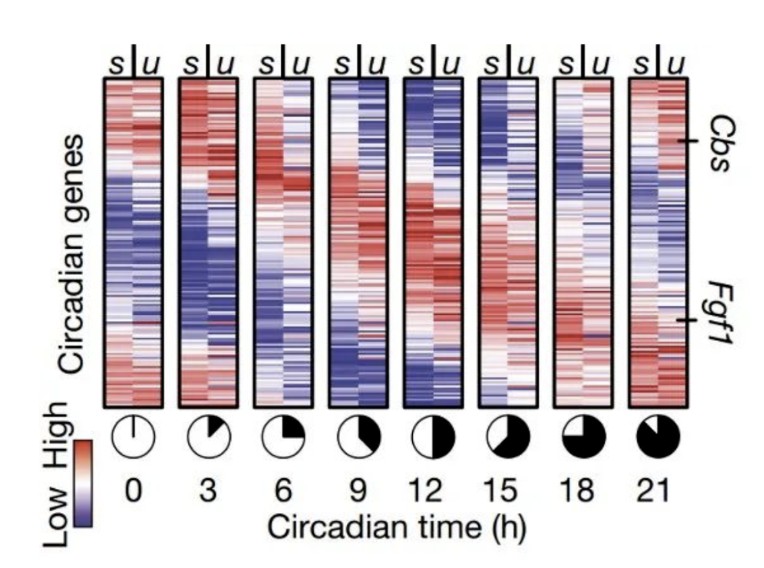
那么单细胞转录组数据中是否可以检测到剪切的mRNA和未剪切的m RNA呢,作者分析了常见的单细胞 RNA测序方法均依赖于oligo-dT 引物来富集聚腺苷酸化 mRNA 方法,发现基于 SMART-seq2、inDrop 和 10x Genomics Chromium 的单细胞 RNA-seq 数据,都可以检测到 15-25% 的reads包含未剪切的内含子序列。因此,单细胞转录组看可以RNA速率分析:
RNA速率公式
基因转录速率 α, 未剪切 mRNA 量 (u) ,剪切 m RNA量(s) ,剪切速率(β),降解速率 γ 。作者为简化RNA速率公式假设 β=1。在这样的模型下,当转录速率 α 恒定时,基因的表达渐近地接近稳态,剪接 (s) 和未剪接 (u) mRNA的稳态丰度由 α 决定。当转录结束时,剪切 (s) 和未剪切 (u) mRNA下降,基因处于下调状态。
RNA速率模型
对于 RNA 速率,通过将测量结果与潜在的 mRNA 剪切动力学相连接来推断细胞的发育轨迹: 特定基因的转录诱导导致(新转录的)前体未剪切 mRNA 的增加,相反地,转录抑制或缺乏导致未剪切 mRNA 的减少。因此,通过区分未剪切的 mRNA 和剪接的 mRNA,可以近似地了解 mRNA 丰度(RNA 速度)的变化。然后,通过 mRNA 的速度组合可以用来估计单个细胞的未来状态。目前 RNA 速率分析可以用三种现有的模型来解决:
- steady-state / deterministic model (using steady-state residuals) 稳态/确定性模型(使用稳态残差)
- stochastic model (using second-order moments) 随机模型(使用二阶矩)
- dynamical model (using a likelihood-based framework) 动态模型(使用基于可能性的框架)
Velocyto 中使用的 steady-state / deterministic model 描述如下: 假设转录阶段(诱导和抑制)持续足够长的时间以达到稳态平衡(活跃和不活跃),速度被量化为观察到的比率与其稳态比率的偏差。稳态下的 mRNA 水平近似于: 在假定稳态状态下线性回归模型的上下分位数。这种简化方式主要基于两个基本假设: 跨基因的共同剪接率和数据中待反映稳态 mRNA 水平。当细胞转录不符合上述假设时,就可能导致速度估计和细胞状态的错误,特别是在一个细胞类型包含多个异质亚种群动态情况下。Stochastic model 旨在更好地捕捉稳态。通过将转录、剪接和降解视为概率事件,由此产生的马尔可夫过程近似力矩方程。其中包含二阶矩,它不仅利用了未剪接到剪接 mRNA 水平的平衡,而且利用了它们的共变。在内分泌胰腺上已经证明,随机性增加了有价值的信息,总体上比确定性模型产生更高的一致性,同时也保持了很高的效率。Dynamical model (最强大,但计算成本最高) 解决了每个基因剪接动力学的全部困难。它使 RNA 速度分析适应广泛变化的细胞发育状态,如非固定种群,它不依赖于共同剪接率,消除了采样稳定状态的限制。
RNA速率结果解读:
饼图展示剪切计数/非剪切mRNA计数的比例。不同的测序方案(Drop-Seq、Smart Seq)都会获得 10%-25%的非重复内含子序列比例。比例符合预期:在循环导管细胞中非剪切比例略低,在 CD4 Naive T细胞中非剪切比例偏高(表示许多基因开始转录)。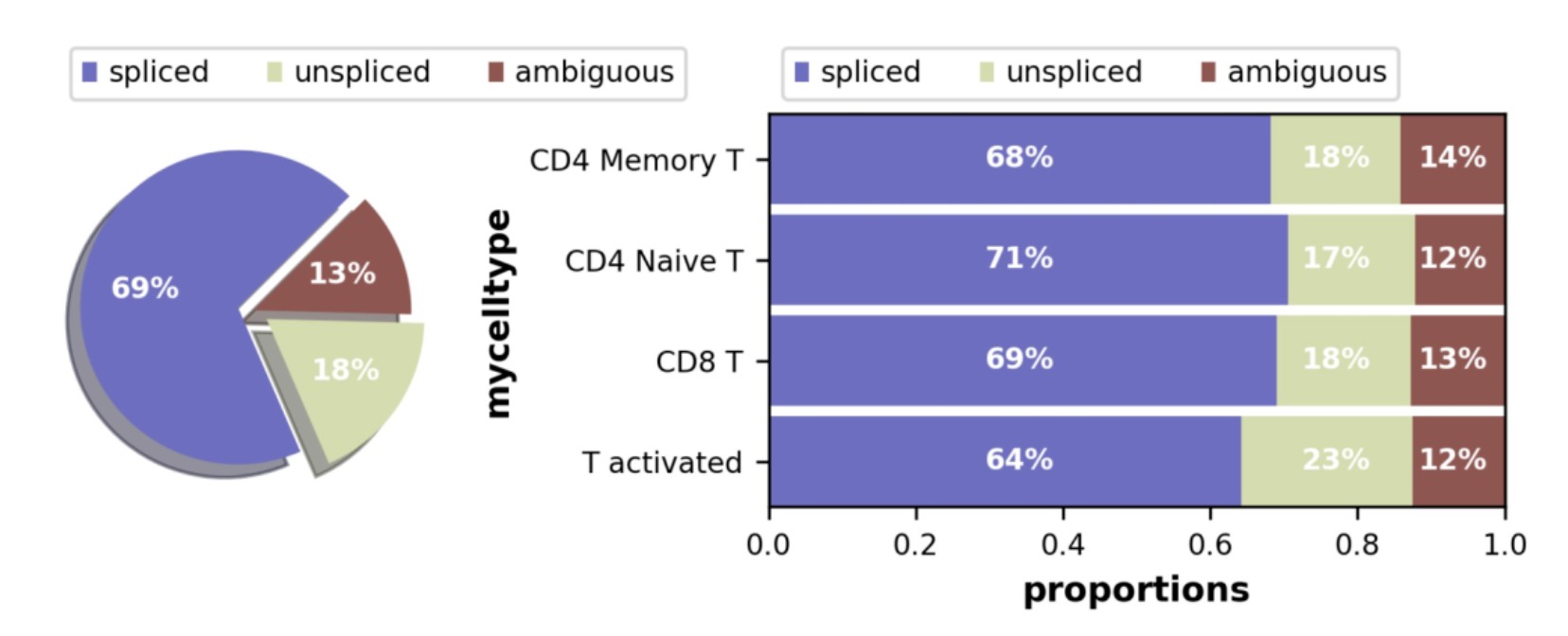
细胞速率可视化
最后,将细胞速率投影到二维空间中,速率流线图准确地描绘了细胞分化轨迹。
velocity length: the speed or rate of differentiation is given by the length of the velocity vector.
velocity confidence: the coherence of the vector field (i.e., how a velocity vector correlates with its neighboring velocities) provides a measure of confidence.
- Speed: length of the velocity vector
- Coherence: how well a velocity vector correlates to its neighbors
参考文献
La Manno, G., Soldatov, R., Zeisel, A. et al. RNA velocity of single cells. Nature 560, 494–498 (2018). https://doi.org/10.1038/s41586-018-0414-6
Bergen, V., Lange, M., Peidli, S. et al. Generalizing RNA velocity to transient cell states through dynamical modeling. Nat Biotechnol 38, 1408–1414 (2020). https://doi.org/10.1038/s41587-020-0591-3
单细胞转录组分析视频:

- 发表于 2024-06-18 12:20
- 阅读 ( 9489 )
- 分类:转录组
你可能感兴趣的文章
- 植物单细胞转录组文章 1500 浏览
- 拟南芥叶单细胞分析流程 1470 浏览
- 单细胞转录组镜像代码更新4.0 2111 浏览
- 单细胞转录组数据挖掘流程记录-头颈癌(HNSCC)(GSE181919) 2284 浏览
- 非负矩阵分解(cNMF) 3877 浏览
- 单细胞转录组如何将UCell CytoTRACE2 infercnv等结果添加到总的rds中 1687 浏览
