使用peer根据表达量数据推断隐藏因子
在做eQTL分析时需要提供隐藏因子作为分析的部分协变量,分析中隐藏因子的引入主要是用来排除影响表达量数据的一些混淆因素,例如样本的采样环境、样本批次、其他位置因素等,这些因素视作分析中的无关因素(即一些无法测量的,但是会影响基因表达的因素),需要进行预先处理。
在做eQTL分析时需要提供隐藏因子作为分析的部分协变量,分析中隐藏因子的引入主要是用来排除影响表达量数据的一些混淆因素,例如样本的采样环境、样本批次、其他位置因素等,这些因素视作分析中的无关因素(即一些无法测量的,但是会影响基因表达的因素),需要进行预先处理。
关于计算多少个隐藏因子,则与样本的数量有关,可以参考如下的标准:
The number of PEER factors was selected as function of sample size (N):
- 15 factors for N < 150
- 30 factors for 150 ≤ N < 250
- 45 factors for 250 ≤ N < 350
- 60 factors for N ≥ 350
根据文章的提示,使用的是peer这个工具:
 peer有两个版本,python和R,我们这里使用R包封装的peer:
peer有两个版本,python和R,我们这里使用R包封装的peer:
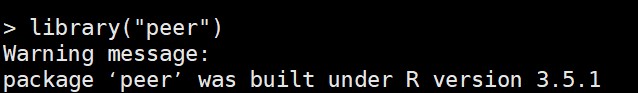
 需要说明,peer包是基于R3.5构建的,因此需要使用R3.x进行安装和使用:
需要说明,peer包是基于R3.5构建的,因此需要使用R3.x进行安装和使用:
使用peer计算前15个隐藏因子的方法:
> library(peer)#加载peer包
> expr = read.csv('examples/data/expression.csv', header=FALSE)#载入表达量数据
> dim(expr)#查看表达量数据的维度
#这里需要特殊说明一下,表达量数据默认是行为样本,列为基因
> model = PEER()#设置分析模型
> PEER_setPhenoMean(model,as.matrix(expr))
NULL#表示没有报错
> dim(PEER_getPhenoMean(model))
[1] 200 100
> PEER_setNk(model,15)#计算前15个隐藏因子
NULL
> PEER_getNk(model)
[1] 10
> PEER_update(model)#开始计算,注意一定要得到一个收敛的结果,如果1000次计算还是不能收敛,需要调整隐藏因子的数量
iteration 0/1000
iteration 1/1000
iteration 2/1000
iteration 3/1000
iteration 4/1000
iteration 5/1000
iteration 6/1000
iteration 7/1000
iteration 8/1000
Converged (var(residuals)) after 8 iterations
NULL
#可以得到推断的混杂因素(NxK矩阵)的后验均值、它们的权重(GxK矩阵)、权重的精度(逆方差)(Kx1矩阵)和残差数据集(NxG矩阵):
> factors = PEER_getX(model)
> dim(factors)
[1] 200 10
> weights = PEER_getW(model)
> dim(weights)
[1] 100 10
> precision = PEER_getAlpha(model)
> dim(precision)
[1] 10 1
> residuals = PEER_getResiduals(model)
> dim(residuals)
[1] 200 100
> plot(precision)
> write.table(factors,"Express_hidden_factors.csv",sep="\t",quote = FALSE,col.names = TRUE,row.names = FALSE)
peer网址:https://github.com/PMBio/peer/wiki/Tutorial
参考:
https://github.com/broadinstitute/gtex-pipeline/tree/master/qtl
- 发表于 2024-06-19 15:39
- 阅读 ( 2588 )
- 分类:R
你可能感兴趣的文章
相关问题
0 条评论
请先 登录 后评论
