eQTpLot包的安装
eQTpLot 是一个直观且用户友好的 R 包,专为可视化 eQTL 和 GWAS 结果的共定位而开发。eQTpLot 将标准 GWAS 和 eQTL 统计结果以及可选的成对 LD 信息作为输入,以生成一系列图表,可视化给定基因-性状对的 eQTL 和 GWAS 信号之间的共定位、相关性和富集。
eQTpLot 是一个直观且用户友好的 R 包,专为可视化 eQTL 和 GWAS 结果的共定位而开发。eQTpLot 将标准 GWAS 和 eQTL 统计结果以及可选的成对 LD 信息作为输入,以生成一系列图表,可视化给定基因-性状对的 eQTL 和 GWAS 信号之间的共定位、相关性和富集。借助 eQTpLot,研究人员可以轻松生成一系列可自定义的图表,清晰地说明给定基因-性状对的以下情况:
- GWAS 和 eQTL 信号之间的共定位
- GWAS 与 eQTL p 值之间的相关性
- 性状显著变异中 eQTL 的富集
- 所研究基因座的 LD 状态
- eQTL 信号效应方向与共定位 GWAS 峰效应方向之间的关系
需要提前安装一些依赖项:
"biomaRt", "dplyr", "GenomicRanges", "ggnewscale", "ggplot2", "ggplotify", "ggpubr", "gridExtra", "Gviz", "LDheatmap", "patchwork"
eQTplot包可以使用devtools直接从github下载:
devtools::install_github("RitchieLab/eQTpLot")
但是有时候因为网络的原因会失败:
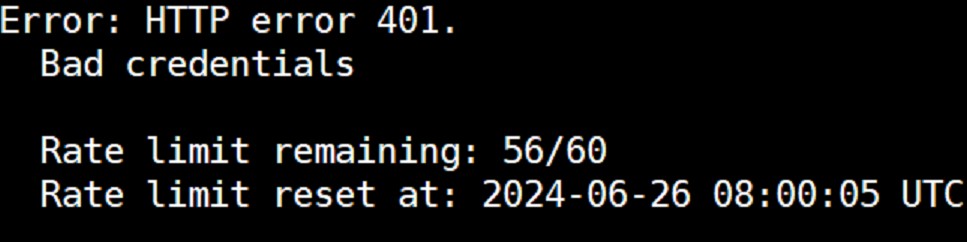
亲测可行的方式,直接从github上下载压缩包,然后手动安装,下载网址:
https://github.com/RitchieLab/eQTpLot/archive/refs/heads/master.zip
R CMD build 包目录的名字 #在含有包目录的文件夹下执行这条指令,会生成一个压缩文件
R CMD INSTALL 压缩文件
进入R,执行指令,查看是否安装成功:
library("eQTpLot ")
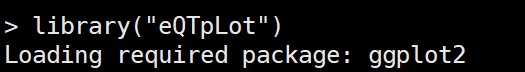
可以了。
- 发表于 2024-06-26 16:06
- 阅读 ( 502 )
- 分类:R
你可能感兴趣的文章
相关问题
0 条评论
请先 登录 后评论
