pySCENIC转录因子分析结果解读
1 单细胞转录组转录因子分析介绍:
TF (transcription factors, TFs)通过与DNA特定区域结合,促进或阻碍DNA的转录过程从而调控基因的表达。了解TF对于了解细胞功能及疾病进程等生命活动有重要意义。
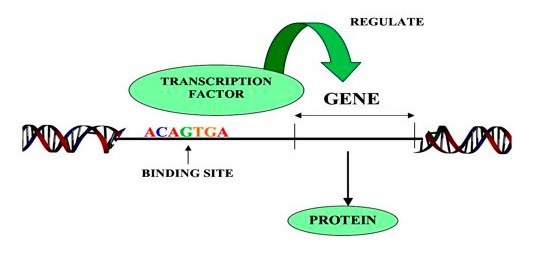
2017年11月,SCENIC发表于Nature Methods,作为主流的单细胞转录因子分析工具,SCENIC在单细胞转录组数据构建基因调控网络、转录因子分析以及细胞状态鉴定方面上表现优异。目前主要有R和python两个版本,支持分析人、小鼠和果蝇的数据。
1.1 pyscenic分析流程
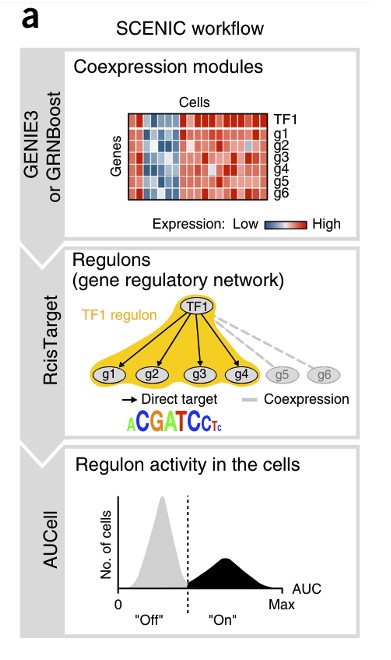
1.1.1 构建共表达网络
输入的数据是标准化的count矩阵(行是基因和列是细胞),从中找出TFs调节的基因构建共表达网络。GENIE3(随机森林)/GRNBoost(Gradient Boosting):基于共表达情况鉴定每个TF的潜在靶点,推断转录因子与候选靶基因之间的共表达模块。每个模块包含一个转录因子及其靶基因。
这个共表达网络只是基于TF和gene表达量相关性推测的,TF和gene之间是否现实存在调控关系还需要进一步验证。验证的方法主要从TF功能结构入手,从上图1我们可以看出,TF是通过直接与DNA结合而发挥作用的,因此我们可以通过反向查看gene上是否存在TF结合的motif序列来验证TF与gene的靶向关系。
1.1.2 motif富集分析
进行TF-motif富集分析,识别直接靶标。仅保留具有正确的上游调节子且显著富集的motif modules,并对它们进行过滤以除去缺乏motif支持的间接靶基因。这些处理后的每个TF及其潜在的直接targets genes被称作一个regulon。
由于上一步GENIE3只是推断共表达,因此会有假阳性和间接targets。使用RcisTarget基于DNA-motif分析识别具有正确上游调控子且显著富集的motif(转录因子直接结合的motif),修剪掉缺乏motif支持的间接靶标。修剪后的每个TF和其潜在的直接作用的target genes被称为为一个regulon。(这一步是SCENIC和其他大多数共表达算法的重要区别)
Regulon调控子:受同一个TF调控的一群基因的集合,即one Regulon = one TF + target genes
1.1.3 AUCell对每个细胞的每个regulon活性进行打分
对于一个regulon来说,比较细胞间的AUCell得分可以鉴定出哪种细胞有显著更高的sub network活性。
原理:AUCell基于基因集(Regulons中所有基因)打分,所得到的分数即为AUC(Area Under Curve)表示Regulons在细胞中的“活性”。打分过程是针对每个细胞,将细胞中所有基因按照表达量从高到低进行排序,根据Regulons中的基因在序列中的位置,计算累计曲线面积 (AUC)。
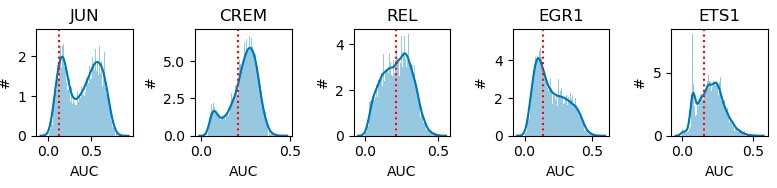
1.1.4 AUCell分数二值化
由于不同regulons包含的基因不同,基于AUC值在所有细胞中的双峰分布特征,增加了Rgulons“on/off”的概念,认为双峰之间的低谷为判断Regulons活性开放的阈值,如果AUC值小于阈值,则判定为该Regulons在该细胞中未开放,即未发挥调控作用。进一步转化为二进制regulon活性矩阵(binarized activity matrix),这将最大化细胞类型的差异,确定regulon在哪些细胞中处于“开放”状态。
- 发表于 2024-07-09 12:05
- 阅读 ( 4311 )
- 分类:转录组
你可能感兴趣的文章
- 植物单细胞转录组文章 1361 浏览
- 拟南芥叶单细胞分析流程 1368 浏览
- 单细胞转录组镜像代码更新4.0 2023 浏览
- 单细胞转录组数据挖掘流程记录-头颈癌(HNSCC)(GSE181919) 2168 浏览
- 非负矩阵分解(cNMF) 3700 浏览
- 单细胞转录组如何将UCell CytoTRACE2 infercnv等结果添加到总的rds中 1638 浏览
