单细胞转录组数据挖掘流程记录-结直肠癌 CRC(GSE146771)
单细胞转录组数据挖掘流程记录-结直肠癌 CRC(GSE146771)
数据介绍:
来自谷歌翻译:
单细胞 RNA 测序 (scRNA-seq) 是定义肿瘤细胞多样性的强大工具,但其在剖析免疫调节疗法潜在机制方面的应用很少。我们对结直肠癌患者的免疫和基质群体进行了 scRNA-seq 分析,确定了特定的巨噬细胞和传统树突状细胞 (cDC) 亚群作为肿瘤微环境中细胞串扰的关键介质。定义小鼠肿瘤中可比较的骨髓细胞群,能够表征它们对骨髓靶向免疫治疗的反应。抗CSF1R治疗优先耗尽具有炎症特征的巨噬细胞,但保留小鼠和人类中表达促血管生成/肿瘤生成基因的巨噬细胞群。用 CD40 激动剂抗体治疗优先激活传统树突状细胞群,并增加 Bhlhe40+ Th1 样细胞和 CD8+ 记忆 T 细胞。我们对人类和小鼠关键骨髓亚群的全面分析确定了调节肿瘤免疫的关键细胞相互作用,并定义了目前正在进行临床测试的骨髓靶向免疫疗法的机制。
数据下载地址:
https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE146771
数据下载与预处理代码:
这个数据有点特殊是smartseq的单细胞数据,为全长数据,并且已经做TPM标准化:
#数据下载
wget -c https://ftp.ncbi.nlm.nih.gov/geo/series/GSE146nnn/GSE146771/suppl/GSE146771%5FCRC.Leukocyte.Smart%2Dseq2.Metadata.txt.gz
wget -c https://ftp.ncbi.nlm.nih.gov/geo/series/GSE146nnn/GSE146771/suppl/GSE146771%5FCRC.Leukocyte.Smart%2Dseq2.TPM.txt.gz
#空格改成tab,引号去掉
zcat GSE146771_CRC.Leukocyte.Smart-seq2.TPM.txt.gz |sed -e 's/ /\t/g' -e 's/"//g' |gzip - > Smart-seq2.tpm.tsv.gz
#CellName 放在第一列作为细胞ID和TPM矩阵保持一致
zcat GSE146771_CRC.Leukocyte.Smart-seq2.Metadata.txt.gz |awk -F '\t' 'BEGIN{OFS="\t"}{print $2,$0}' |gzip - >metadata.tsv.gz
单细胞分析:
#读入数据并质控 Rscript $scripts/seurat_sc_qc.r --count Smart-seq2.tpm.tsv.gz \ --project GSE146771 \ --nUMI.min 500 \ --nUMI.max 50000 \ --nGene.min 250 \ --mito.gene.pattern "^MT.*-" \ --percent_mito 2 \ -o 01.qc \ -p GSE146771 --metadata metadata.tsv.gz #单细胞聚类分群分析 # 由于 smartseq数据输入的为TPM数据,已经做了标准化,这里不需要Normalization,也不支持 sctransform Rscript $scripts/seurat_sc_cluster.r --rds 01.qc/GSE146771.afterQC.rds \ --noNormalization \ -p GSE146771 --resolution 0.5 -d 30 \ -o 02.cluster --integrate.method harmony --batch.id Sample \ --vars.to.regress nUMI percent_mito --high.variable.genes 2000
结果展示:
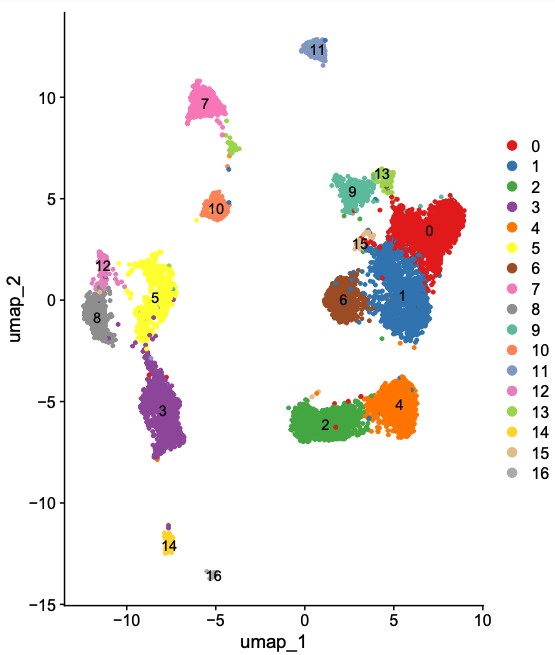
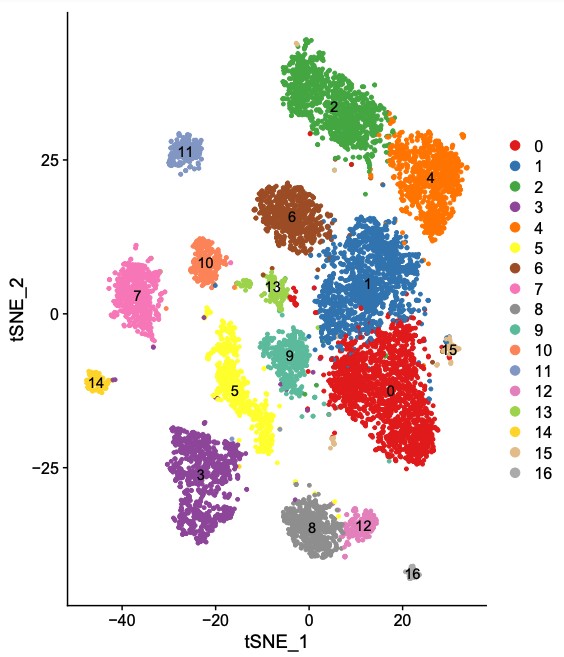
单细胞转录组分析课程推荐:https://bdtcd.xetslk.com/s/4i88K6
- 发表于 2024-07-24 16:38
- 阅读 ( 1032 )
- 分类:转录组
你可能感兴趣的文章
- 拟南芥叶单细胞分析流程 328 浏览
- 单细胞转录组镜像代码更新4.0 981 浏览
- 单细胞转录组数据挖掘流程记录-头颈癌(HNSCC)(GSE181919) 686 浏览
- 非负矩阵分解(cNMF) 1262 浏览
- 单细胞转录组如何将UCell CytoTRACE2 infercnv等结果添加到总的rds中 753 浏览
- 单细胞转录组-infer CNV拷贝数变异分析介绍 1441 浏览
0 条评论
请先 登录 后评论


